Coronavírus da síndrome respiratória aguda grave 2: diferenças entre revisões
m Acréscimo de predefinição |
|||
| Linha 1: | Linha 1: | ||
{{Ver desambig|este=o vírus|a doença|COVID-19|a pandemia atualmente em curso|Pandemia de COVID-19}} |
{{Ver desambig|este=o vírus|a doença|COVID-19|a pandemia atualmente em curso|Pandemia de COVID-19}} |
||
{{Evento atual}} |
|||
{{Info/Taxonomia |
{{Info/Taxonomia |
||
| nome = Coronavírus da Síndrome Respiratória Aguda Grave 2 |
| nome = Coronavírus da Síndrome Respiratória Aguda Grave 2 |
||
| Linha 40: | Linha 39: | ||
| sem_classificação = [[Vírus]] |
| sem_classificação = [[Vírus]] |
||
}} |
}} |
||
O '''vírus da síndrome respiratória aguda grave 2''' ('''SARS-CoV-2'''),<ref name="CoronavirusStudyGroup" /><ref name=":2">{{citar jornal|url=https://www.bbc.com/news/world-asia-china-51466362 |título=Coronavirus disease named Covid-19 |data=11 de fevereiro de 2020 |obra=[[BBC News Online]] |arquivourl=https://web.archive.org/web/20200215204154/https://www.bbc.com/news/world-asia-china-51466362 |arquivodata=15 de fevereiro de 2020 |urlmorta= não|acessodata=15 de fevereiro de 2020 }}</ref> anteriormente denominado de forma provisória "2019-nCoV",<ref name=WHO21Jan2020>{{citar relatório|vauthors=((World Health Organization)) |ano=2020 |título=Surveillance case definitions for human infection with novel coronavirus (nCoV): interim guidance v1, January 2020 |autorlink =World Health Organization |publicado=World Health Organization |hdl=10665/330376 |id=WHO/2019-nCoV/Surveillance/v2020.1 }}</ref><ref>{{citar web|título=Healthcare Professionals: Frequently Asked Questions and Answers |website=United States [[Centers for Disease Control and Prevention]] (CDC) |data=11 de fevereiro de 2020 |url=https://www.cdc.gov/coronavirus/2019-ncov/hcp/faq.html |acessodata=15 de fevereiro de 2020 |arquivourl=https://web.archive.org/web/20200214023335/https://www.cdc.gov/coronavirus/2019-ncov/hcp/faq.html |arquivodata=14 de fevereiro de 2020 |urlmorta=unfit }}</ref><ref>{{citar web|título=About Novel Coronavirus (2019-nCoV) |website=United States [[Centers for Disease Control and Prevention]] (CDC) |data=11 de fevereiro de 2020 |url=https://www.cdc.gov/coronavirus/2019-ncov/about/index.html |arquivourl=https://web.archive.org/web/20200211105920/https://www.cdc.gov/coronavirus/2019-ncov/about/index.html |arquivodata=11 de fevereiro de 2020 |urlmorta=unfit |acessodata=25 de fevereiro de 2020}}</ref> é um [[Vírus (+)ssRNA|vírus ARN de cadeia simples positiva]].<ref name=":xinhuanet9Jan2020">{{citar jornal|url=http://www.xinhuanet.com/english/2020-01/09/c_138690570.htm |título=New-type coronavirus causes pneumonia in Wuhan: expert |acessodata=9 de janeiro de 2020 |urlmorta= não|arquivourl=https://web.archive.org/web/20200109084208/http://www.xinhuanet.com/english/2020-01/09/c_138690570.htm |arquivodata=9 de janeiro de 2020 |agência=[[Xinhua]] }}</ref><ref name=":gisaid">{{citar web|url=https://platform.gisaid.org/epi3/start/CoV2020 |título=CoV2020 |website=GISAID EpifluDB |urlmorta= não|acessodata=12 de janeiro de 2020 |url-access=registration|arquivourl=https://web.archive.org/web/20200112130540/https://platform.gisaid.org/epi3/start/CoV2020 |arquivodata=12 de janeiro de 2020}}</ref> É contagioso entre seres humanos e é a causa da doença [[COVID-19]], da qual existe uma [[Pandemia de COVID-19|pandemia em curso]] .<ref name="NYT 20200130" /><ref name="Chan24Jan2020" /> |
|||
| ⚫ | |||
O '''Coronavírus da Síndrome Respiratória Aguda Grave 2''' (abreviado para '''SARS-CoV-2''', do [[Língua inglesa|inglês]] ''Severe Acute Respiratory Syndrome Coronavirus 2''), como denotado pelo [[Comitê Internacional de Taxonomia de Vírus]] (ICTV),<ref>{{Citar periódico|data=2020-02-11|titulo=Coronavirus disease named Covid-19|url=https://www.bbc.com/news/world-asia-china-51466362|jornal=BBC News|lingua=en-GB}}</ref><ref>{{Citar periódico|ultimo=Gorbalenya|primeiro=Alexander E.|data=2020-02-11|titulo=Severe acute respiratory syndrome-related coronavirus – The species and its viruses, a statement of the Coronavirus Study Group|url=https://www.biorxiv.org/content/10.1101/2020.02.07.937862v1|jornal=bioRxiv|lingua=en|paginas=2020.02.07.937862|doi=10.1101/2020.02.07.937862}}</ref> anteriormente denominado '''novo coronavírus de 2019''' (2019-nCoV), é um [[coronavírus]], relatado pela primeira vez em 2019 e [[Genoma#Sequenciamento e mapeamento|sequenciado geneticamente]] após [[Teste de ácido nucleico|testes de ácido nucleico]] em uma amostra de paciente positiva em um paciente com [[pneumonia]] durante o [[Pandemia de COVID-19|surto da COVID-19]].<ref>{{citar web|url=http://www.chinacdc.cn/dfdt/201912/t20191226_209404.html|título=中国疾病预防控制中心|publicado=China CDC|acessodata=20 de janeiro de 2020|língua=chinês}}</ref><ref name=":0">{{citar web|url=http://www.xinhuanet.com/english/2020-01/09/c_138690570.htm|título=New-type coronavirus causes pneumonia in Wuhan: expert – Xinhua {{!}} English.news.cn|publicado=Xinhua Net|acessodata=20 de janeiro de 2020|língua=inglês}}</ref><ref name=":1">{{citar web|url=https://platform.gisaid.org/epi3/start/CoV2020|título=CoV2020|publicado=Gisaid|urlmorta=não|acessodata=20 de janeiro de 2020|língua=inglês}}</ref> Esse surto já infectou mais de {{Número de casos de COVID-19|confirmados}} pessoas, em 156 territórios, matando {{Número de casos de COVID-19|mortes}} pessoas, em todo o mundo.<ref name=DXY>{{citar web|url=https://3g.dxy.cn/newh5/view/pneumonia|script-title=zh:疫情地图|publicado=DXY|data=28 de janeiro de 2020|acessodata=1 de fevereiro de 2020|língua=zh-cn|arquivourl=https://web.archive.org/web/20200123232022/https://3g.dxy.cn/newh5/view/pneumonia|arquivodata=23 de janeiro de 2020|urlmorta= não}}</ref><ref name=BNO>{{citar web|título=Tracking coronavirus: Map, data and timeline|url=https://bnonews.com/index.php/2020/01/the-latest-coronavirus-cases|trabalho=BNO News|acessodata=1 de fevereiro de 2020|língua=inglês|urlmorta=não}}</ref><ref>{{citar web|url=https://g1.globo.com/jornal-nacional/noticia/2020/03/06/passa-de-100-mil-o-numero-de-infectados-pelo-novo-coronavirus-no-mundo.ghtml|publicado=Globo.com|título=Passa de 100 mil o número de infectados pelo novo coronavírus no mundo|acessodata=8 de março de 2020}}</ref> |
|||
O SARS-CoV-2 apresenta [[Distância genética|proximidade genética]] com os [[coronavírus]] de morcegos, dos quais se terá provavelmente [[Vírus transmitidos por morcegos|originado]].<ref name="NatureZhou">{{citar periódico| vauthors = Zhou P, Yang XL, Wang XG, Hu B, Zhang L, Zhang W, Si HR, Zhu Y, Li B, Huang CL, Chen HD, Chen J, Luo Y, Guo H, Jiang RD, Liu MQ, Chen Y, Shen XR, Wang X, Zheng XS, Zhao K, Chen QJ, Deng F, Liu LL, Yan B, Zhan FX, Wang YY, Xiao GF, Shi ZL | display-authors = 6 |título= A pneumonia outbreak associated with a new coronavirus of probable bat origin |periódico= Nature | volume = 579 |número= 7798 |páginas= 270–273 |data=fevereiro de 2020 | pmid = 32015507 | doi = 10.1038/s41586-020-2012-7 }}</ref> |
|||
Segundo a OMS, pode ter havido transmissão limitada de humano para humano deste coronavírus nas famílias de pacientes, e um surto mais amplo é possível. No momento, nenhum tratamento específico para o novo vírus está disponível, mas os [[Antiviral|antivirais]] existentes podem ser "propostos".<ref>{{citar web|url=https://www.reuters.com/article/us-china-health-pneumonia-who/who-says-new-china-coronavirus-could-spread-warns-hospitals-worldwide-idUSKBN1ZD16J|título=WHO says new China coronavirus could spread, warns hospitals worldwide|publicado=[[Reuters]]|data=14 de janeiro de 2020|acessodata=20 de janeiro de 2020|língua=inglês}}</ref> |
|||
<ref name="LancetNowcasting">{{citar periódico| vauthors = Perlman S |título= Another Decade, Another Coronavirus |periódico= [[The New England Journal of Medicine]] | volume = 382 |número= 8 |páginas= 760–762 |data=fevereiro de 2020 | pmid = 31978944 | doi = 10.1056/NEJMe2001126 }}</ref><ref name=":4" /> Pensa-se que antes de ser introduzido aos seres humanos tenha também estado envolvido um [[Reservatório natural|reservatório]] animal intermédio, como o [[pangolim]].<ref name=":0">{{citar relatório| vauthors=((World Health Organization)) |ano=2020 |título=Novel Coronavirus (2019-nCoV): situation report, 22 |publicado=[[World Health Organization]] | hdl=10665/330991 }}</ref><ref name=":1">{{citar jornal|url=https://www.dw.com/en/coronavirus-from-bats-to-pangolins-how-do-viruses-reach-us/a-52291570|título=Coronavirus: From bats to pangolins, how do viruses reach us?|vauthors=Shield C|data=7 de fevereiro de 2020|publicado=[[Deutsche Welle]]|acessodata=13 de março de 2020}}</ref> Do ponto de vista [[Taxonomia|taxonómico]], o SARS-CoV-2 está [[Classificação dos vírus|classificado]] como [[estirpe]] da espécie [[coronavírus relacionado com a síndrome respiratória aguda grave]] (SARSr-CoV).<ref name="CoronavirusStudyGroup">{{citar periódico| vauthors = Gobalenya AE, Baker SC, Baric RS, de Groot RJ, Drosten C, Gulyaeva AA, Haagmans BL, Lauber C, Leontovich AM, Neuman BW, Penzar D, Perlman S, ((Poon LLM)), Samborskiy DV, Sidorov IA, Sola I, Ziebuhr J|display-authors=3 |título= The species Severe acute respiratory syndrome-related coronavirus: classifying 2019-nCoV and naming it SARS-CoV-2 |periódico= Nature Microbiology |páginas= 1–9 |data=março de 2020 | pmid = 32123347 | doi = 10.1038/s41564-020-0695-z | url = https://www.nature.com/articles/s41564-020-0695-z }}</ref> |
|||
Uma vez que a estirpe foi descoberta em [[Wuhan]], na China, é por vezes denominada "vírus de Wuhan virus" ou "coronavírus de Wuhan",<ref name="HuangNPR">{{citar web|url=https://www.npr.org/sections/goatsandsoda/2020/01/22/798277557/how-does-wuhan-coronavirus-compare-to-mers-sars-and-the-common-cold|título=How Does Wuhan Coronavirus Compare with MERS, SARS and the Common Cold?|vauthors=Huang P|data=22 de janeiro de 2020|website=[[NPR]]|urlmorta= não|arquivourl=https://web.archive.org/web/20200202094021/https://www.npr.org/sections/goatsandsoda/2020/01/22/798277557/how-does-wuhan-coronavirus-compare-to-mers-sars-and-the-common-cold|arquivodata=2 de fevereiro de 2020|acessodata=3 de fevereiro de 2020}}</ref><ref name="Fox2020">{{citar periódico|vauthors=Fox D|data=24 de janeiro de 2020|título=What you need to know about the Wuhan coronavirus|periódico=[[Nature (journal)|Nature]]|doi=10.1038/d41586-020-00209-y|issn=0028-0836}}</ref><ref>{{citar jornal|url=https://www.nbcnews.com/news/asian-america/cdc-chief-spurns-term-chinese-coronavirus-used-gop-lawmakers-n1156656|título=GOP lawmakers continue to use 'Wuhan virus' or 'Chinese coronavirus'|vauthors=Yam K|publicado=[[NBC News]]|urlmorta= não|data=12 de março de 2020|acessodata=19 de março de 2020}}</ref><ref>{{citar jornal|url=https://www.foxnews.com/politics/kevin-mccarthy-pelosi-omar-chinese-coronavirus-racist|título=McCarthy knocks Dems after they claim saying 'Chinese coronavirus' is racist|vauthors=Dorman S|publicado=[[Fox News]]|urlmorta= não|acessodata=12 de março de 2020|data=11 de março de 2020}}</ref> embora a [[Organização Mundial de Saúde]] (OMS) desaconselhe a utilização de nomes baseados na localização.<ref>{{citar relatório| vauthors = ((World Health Organization)) |ano= 2015 |título= World Health Organization best practices for the naming of new human infectious diseases |autorlink = World Health Organization |publicado= World Health Organization | hdl = 10665/163636 | id = WHO/HSE/FOS/15.1 }}</ref> Para evitar confusão com a doença [[síndrome respiratória aguda grave]] (SARS), em comunicados públicos a OMS por vezes refere-se ao vírus como "vírus responsável pela COVID-19" ou "vírus da COVID-19".<ref name="COVIDname">{{citar web|url=https://www.who.int/emergencies/diseases/novel-coronavirus-2019/technical-guidance/naming-the-coronavirus-disease-(covid-2019)-and-the-virus-that-causes-it|título=Naming the coronavirus disease (COVID-2019) and the virus that causes it|publicado=World Health Organization|acessodata=24 de fevereiro de 2020|citação=From a risk communications perspective, using the name SARS can have unintended consequences in terms of creating unnecessary fear for some populations.... For that reason and others, WHO has begun referring to the virus as "the virus responsible for COVID-19" or "the COVID-19 virus" when communicating with the public. Neither of these designations are [sic] intended as replacements for the official name of the virus as agreed by the ICTV.}}</ref> Tanto o vírus como a doença são muitas vezes denominados "coronavírus" pelo público em geral, embora cientistas e jornalistas usem termos mais precisos.<ref name=NYT6>{{citar jornal| website = [[The New York Times]] | url = https://www.nytimes.com/2020/03/04/us/coronavirus-recovery.html | vauthors = Harmon A |título= We Spoke to Six Americans with Coronavirus |data=4 de março de 2020 |acessodata=16 de março de 2020}}</ref> |
|||
Um novo estudo, de março de 2020, financiado pelo ''[[National Institutes of Health]]'' afirma que o novo coronavírus pode permanecer em superfícies de plástico e inox contaminadas por até três dias. Para o estudo, os pesquisadores compararam a "estabilidade superficial" do novo coronavírus com a do vírus [[SARS-CoV]], que desencadeou a epidemia do [[Síndrome respiratória aguda grave|SARS]] iniciada em 2002. Os pesquisadores descobriram que o novo coronavírus poderia permanecer no cobre por até quatro horas, em papelão por até 24 horas e em plástico e aço inoxidável por dois a três dias.<ref>{{citar web|url=https://edition.cnn.com/world/live-news/coronavirus-outbreak-03-11-20-intl-hnk/h_c7f0bf6a228e20054cbf4fb4fb700812|publicado=CNN|título=Novel coronavirus can last on contaminated plastic and stainless surfaces for up to 3 days|data=11 de março de 2020|acessodata=11 de março de 2020|língua=en}}</ref> |
|||
== |
==Virologia== |
||
===Infeção=== |
|||
Nenhum tratamento específico está disponível no momento; portanto, o tratamento é focado no alívio dos sintomas, que podem incluir [[febre]], [[Tosse|tosse seca]] e [[dispneia]]. O Centro Chinês para Controle e Prevenção de Doenças (CCDC) está testando os tratamentos existentes contra pneumonia quanto à eficácia no tratamento de pneumonia relacionada ao coronavírus. Os [[Antiviral|antivirais]] existentes estão sendo estudados, incluindo inibidores de protease como [[indinavir]], [[saquinavir]] e [[Associação lopinavir/ritonavir|lopinavir/ritonavir]], bem como o inibidor da RNA polimerase [[remdesivir]]. A [[Interferona de tipo I|interferona beta]] também está sendo estudada. A eficácia dos [[Anticorpo monoclonal|anticorpos monoclonais]] previamente identificados (mAbs) também está sob investigação.<ref name="wmhc2020-01-112">{{citar web|url=http://wjw.wuhan.gov.cn/front/web/showDetail/2020011109036|título=Experts explain the latest bulletin of unknown cause of viral pneumonia|data=11 de janeiro de 2020|website=Wuhan Municipal Health Commission|urlmorta=não|arquivourl=https://web.archive.org/web/20200111031745/http://wjw.wuhan.gov.cn/front/web/showDetail/2020011109036|arquivodata=11 de janeiro de 2020|acessodata=1 de fevereiro de 2020|língua=inglês}}</ref><ref name="Hui14Jan2020">{{citar periódico|autor=Hui DS, I Azhar E, Madani TA, Ntoumi F, Kock R, Dar O, Ippolito G, Mchugh TD, Memish ZA, Drosten C, Zumla A, Petersen E|título=The continuing 2019-nCoV epidemic threat of novel coronaviruses to global health – The latest 2019 novel coronavirus outbreak in Wuhan, China|periódico=Int J Infect Dis|data=14 de janeiro de 2020|volume=91|issue=|página=264–266|pmid=31953166|doi=10.1016/j.ijid.2020.01.009|língua=inglês}}</ref><ref>{{citar web|url=https://www.who.int/news-room/q-a-detail/q-a-coronaviruses|title=Q&A on coronaviruses|website=[[World Health Organization]] (WHO)|acessodata=1 de fevereiro de 2020|língua=inglês|arquivourl=https://web.archive.org/web/20200120174649/https://www.who.int/news-room/q-a-detail/q-a-coronaviruses|arquivodata=20 de janeiro de 2020|urlmorta=não}}</ref> |
|||
A transmissão do SARS-CoV-2 entre seres humanos foi confirmada pela primeira vez durante a [[Pandemia de COVID-19|pandemia de coronavírus de 2019-20]].<ref name="Chan24Jan2020"/> A principal forma de transmissão são gotículas produzidas no sistema respiratório e expulsas ao tossir ou espirrar até um raio de 1,8 m.<ref>{{citar jornal|url=https://www.nbcnews.com/health/health-news/how-does-new-coronavirus-spread-n1121856|título=How does coronavirus spread?|vauthors=Edwards E|data=25 de janeiro de 2020|publicado=[[NBC News]]|acessodata=13 de março de 2020|arquivourl=https://web.archive.org/web/20200128081650/https://www.nbcnews.com/health/health-news/how-does-new-coronavirus-spread-n1121856|arquivodata=28 de janeiro de 2020|urlmorta= não}}</ref><ref>{{citar web|url=https://www.cdc.gov/coronavirus/2019-ncov/about/transmission.html|título=How COVID-19 Spreads |data=27 de janeiro de 2020|website=U.S. [[Centers for Disease Control and Prevention]] (CDC)|acessodata=29 de janeiro de 2020|arquivourl=https://web.archive.org/web/20200128152653/https://www.cdc.gov/coronavirus/2019-ncov/about/transmission.html|arquivodata=28 de janeiro de 2020|urlmorta= não}}</ref> Outra possível causa de infeção é o contacto indireto através de superfícies contaminadas.<ref>{{citar web|url=https://www.who.int/docs/default-source/coronaviruse/getting-workplace-ready-for-covid-19.pdf|título=Getting your workplace ready for COVID-19|data=27 de fevereiro de 2020|website=World Health Organization|urlmorta= não|arquivourl=|arquivodata=|acessodata=3 de março de 2020}}</ref> A investigação preliminar sugere que o vírus possa permanecer ativo em [[plástico]] e [[aço]] até três dias, embora não consiga sobreviver em cartão mais do que um dia ou em [[cobre]] mais do que quatro horas.<ref>{{citar periódico|título=Correspondence: Aerosol and Surface Stability of SARS-CoV-2 as Compared with SARS-CoV-1|ano=2020|vauthors=van Doremalen N, Bushmaker T, Morris DH, Holbrook MG, Gamble A, Williamson BN, Tamin A, Harcourt JL, Thornburg NJ, Gerber SI, Lloyd-Smith JO, de Wit E, Munster VJ|display-authors=3|periódico=[[The New England Journal of Medicine]]|doi=10.1056/NEJMc2004973|pmid=32182409|data=17 de março de 2020}}</ref> Foi também observado ARN viral em [[fezes]] de pacientes infetados.<ref>{{citar periódico| vauthors = Holshue ML, DeBolt C, Lindquist S, Lofy KH, Wiesman J, Bruce H, Spitters C, Ericson K, Wilkerson S, Tural A, Diaz G, Cohn A, Fox L, Patel A, Gerber SI, Kim L, Tong S, Lu X, Lindstrom S, Pallansch MA, Weldon WC, Biggs HM, Uyeki TM, Pillai SK | display-authors = 3 |título= First Case of 2019 Novel Coronavirus in the United States |periódico= [[The New England Journal of Medicine]] | volume = 382 |número= 10 |páginas= 929–936 |data=março de 2020 | pmid = 32004427 | doi = 10.1056/NEJMoa2001191 }}</ref> |
|||
Ainda não é claro se o vírus é infecioso durante o [[período de incubação]].<ref>{{citar periódico|título=Study claiming new coronavirus can be transmitted by people without symptoms was flawed |periódico=Science |vauthors=Kupferschmidt K|data=fevereiro de 2020 | doi=10.1126/science.abb1524}}</ref> Em 1 de fevereiro de 2020 a OMS indicava que "a transmissão a partir de casos assintomáticos provavelmente não é uma das principais formas de transmissão".<ref>{{citar relatório| vauthors=((World Health Organization)) |ano=2020 |título=Novel Coronavirus (2019-nCoV): situation report, 12 |publicado=[[World Health Organization]] | hdl=10665/330777 }}</ref> Acredita-se que a maior parte das infeções em seres humanos seja o resultado de transmissão entre pessoas que manifestam sintomas de [[COVID-19]]. No entanto, um modelo epidemiológico do início so surto na China sugere que a transmissão pré-sintomática pode ser típica entre as infeções documentadas.<ref>{{citar periódico|vauthors= Li R, Pei S, Chen B, Song Y, Zhang T, Yang W, Shaman J |título=Substantial undocumented infection facilitates the rapid dissemination of novel coronavirus (SARS-CoV2) |periódico=Science |páginas=eabb3221 |data=16 de março de 2020 |acessodata=17 de março de 2020 |display-authors=3 |doi=10.1126/science.abb3221 |pmid=32179701 |url=https://science.sciencemag.org/content/early/2020/03/13/science.abb3221}}</ref> |
|||
Em 26 de fevereiro de 2020, os [[Estados Unidos da América|Estados Unidos]] iniciaram testes em humanos de um medicamento para tratar o novo coronavírus. O primeiro participante é um [[norte-americano]] que contraiu [[COVID-19]] e foi evacuado do navio [[Diamond Princess (navio)|Diamond Princess]] atracado no [[Japão]].<ref>{{citar web|url=https://edition.cnn.com/2020/02/26/us/coronavirus-us-american-treatment-trial/index.html|publicado=CNN|título=The US has started human testing of a drug to treat the novel coronavirus|data=26 de fevereiro de 2020|acessodata=26 de fevereiro de 2020|autor= Madeline Holcombe, Eliott C. McLaughlin and Steve Almasy|língua=en}}</ref> |
|||
== |
===Reservatório=== |
||
[[Imagem:SARS-CoV-2 49531042877.jpg|thumb|SARS-CoV-2 a emergir de uma célula humana em laboratório (coloração digital)]] |
|||
As sequências do SARS-CoV-2 mostram semelhanças com os [[betacoronavírus]] encontrados nos [[morcego]]s, mas são geneticamente distintas de outros coronavírus, como o coronavírus relacionado à síndrome respiratória aguda grave (SARS) e o coronavírus relacionado à [[síndrome respiratória por coronavírus do Oriente Médio]] (MERS).<ref name=":1"/> Como o SARS-CoV, é um membro da linhagem Beta-CoV B.<ref>{{citar web|título=Phylogeny of SARS-like betacoronaviruses|url=https://nextstrain.org/groups/blab/sars-like-cov|publicado=Next Strain|acessodata=20 de janeiro de 2020|língua=inglês}}</ref> |
|||
A OMS considera serem os morcegos o mais provável [[reservatório natural]] de SARS-CoV-2,<ref>{{citar periódico|url=https://www.who.int/docs/default-source/coronaviruse/who-china-joint-mission-on-covid-19-final-report.pdf|título=Report of the WHO-China Joint Mission on Coronavirus Disease 2019 (COVID-19)|publicado=[[World Health Organization]] (WHO)|data=24 de fevereiro de 2020|acessodata=5 de março de 2020}}</ref> embora algumas diferenças entre os coronavírus dos morcegos e o SARS-CoV-2 sugiram que os seres humanos foram infetados através de um hospedeiro intermédio.<ref name="nature feb2020" /> |
|||
As primeiras infeções conhecidas pela estirpe SARS-CoV-2 foram descobertas em [[Wuhan]], na China.<ref name="NatureZhou" /> Ainda não é claro qual foi a fonte original de transmissão viral para os seres humanos nem quando é que a estirpe se tornou [[Patógeno|patogénica]],<ref name=early>{{citar periódico| vauthors= Cohen J |título=Wuhan seafood market may not be source of novel virus spreading globally |periódico=Science |data=janeiro de 2020 | issn=0036-8075 | doi=10.1126/science.abb0611 }}</ref><ref>{{citar web|url=https://www.popsci.com/story/health/wuhan-coronavirus-china-wet-market-wild-animal/|título=We're still not sure where the Wuhan coronavirus really came from|vauthors=Eschner K|data=28 de janeiro de 2020|website=[[Popular Science]]|urlmorta= não|arquivourl=https://archive.today/NXjMq|arquivodata=29 de janeiro de 2020|acessodata=30 de janeiro de 2020}}</ref><ref>{{citar periódico|url= http://www.chinaxiv.org/abs/202002.00033 |título=Decoding evolution and transmissions of novel pneumonia coronavirus using the whole genomic data|data=21 de fevereiro de 2020| vauthors = Yu WB, Tang GD, Zhang L, Corlett RT |doi=10.12074/202002.00033 |doi-broken-date=2020-02-26}}</ref><ref name="Proximal" /> embora já se tenha determinado que a estirpe é de origem natural.<ref name="Proximal">{{citar periódico|vauthors=Andersen KG, Rambaut A, Lipkin WI, Holmes EC, Garry RF|título=Correspondence: The proximal origin of SARS-CoV-2 |url=https://www.nature.com/articles/s41591-020-0820-9 |display-authors=3 |data=17 de março de 2020 |periódico=[[Nature Medicine]] |doi=10.1038/s41591-020-0820-9 |acessodata=18 de março de 2020 }}</ref> A investigação do [[reservatório natural]] da estirpe de vírus que causou a [[Síndrome respiratória aguda grave|pandemia de SARS em 2002-2004]] permitiu a descoberta de diversos coronavírus semelhantes à SARS em [[morcego]]s, a maior parte com origem no género ''[[Rhinolophus]]'' dos [[Rhinolophidae|morcegos-de-ferradura]], e duas [[Sequência de ADN|sequências de ácido nucleico]] encontradas em amostras de ''[[Rhinolophus sinicus]]'' revelaram uma semelhança de 80% em relação ao SARS-CoV-2.<ref name=":4">{{citar periódico| vauthors = Benvenuto D, Giovanetti M, Ciccozzi A, Spoto S, Angeletti S, Ciccozzi M |título= The 2019-new coronavirus epidemic: Evidence for virus evolution |periódico= Journal of Medical Virology | volume = 92 |número= 4 |páginas= 455–459 |data=abril de 2020 | pmid = 31994738 | doi = 10.1002/jmv.25688 }}</ref><ref>{{citar periódico|título=Bat SARS-like coronavirus isolate bat-SL-CoVZC45, complete genome |publicado=[[National Center for Biotechnology Information]] (NCBI) |data=15 de fevereiro de 2020 | url=https://www.ncbi.nlm.nih.gov/nuccore/MG772933 |acessodata=15 de fevereiro de 2020}}</ref><ref>{{citar periódico|título=Bat SARS-like coronavirus isolate bat-SL-CoVZXC21, complete genome |publicado=[[National Center for Biotechnology Information]] (NCBI) |data=15 de fevereiro de 2020 | url=https://www.ncbi.nlm.nih.gov/nuccore/MG772934 |acessodata=15 de fevereiro de 2020}}</ref> Uma terceira sequência de ácido nucleico de ''[[Rhinolophus affinis]]'' recolhida na [[província de Yunnan]] revelou uma semelhança de 96% em relação ao SARS-CoV-2.<ref name="NatureZhou" /><ref>{{citar periódico|url=https://www.ncbi.nlm.nih.gov/nuccore/1802633852|título=Bat coronavirus isolate RaTG13, complete genome|publicado=[[National Center for Biotechnology Information]] (NCBI)|data=10 de fevereiro de 2020|acessodata=5 de março de 2020}}</ref> |
|||
Cinco genomas do novo coronavírus foram isolados e relatados, incluindo BetaCoV/Wuhan/IVDC-HB-01/2019, BetaCoV/Wuhan/IVDC-HB-04/2020, BetaCoV/Wuhan/IVDC-HB-05/2019, BetaCoV/Wuhan/WIV04/2019 e BetaCoV/Wuhan/IPBCAMS-WH-01/2019 do Instituto Nacional de Controle e Prevenção de Doenças Virais da China, Instituto de Biologia de Patógenos e Hospital Wuhan Jinyintan.<ref name=":1"/><ref>{{citar web|url=http://virological.org/t/initial-genome-release-of-novel-coronavirus/319|título=Initial genome release of novel coronavirus|data=11 de janeiro de 2020|publicado=Virological|acessodata=20 de janeiro de 2020|língua=inglês}}</ref><ref>{{citar web|url=https://www.ncbi.nlm.nih.gov/nuccore/MN908947|título=Wuhan seafood market pneumonia virus isolate Wuhan-Hu-1, complete genome|publicado=NCBI|acessodata=20 de janeiro de 2020|língua=inglês}}</ref> Sua sequência de [[RNA]] tem aproximadamente 30k [[nucleotídeo]]s de comprimento.<ref name=":1"/> |
|||
Um estudo [[Metagenômica|metagenómico]] publicado em 2019 concliui que o [[SARS-CoV]], a estirpe que causa a SARS, era o coronavírus com maior distribuição entre uma amostra de [[Pangolim-malaio|pangolins-malaio]].<ref>{{citar periódico| vauthors = Liu P, Chen W, Chen JP |título= Viral Metagenomics Revealed Sendai Virus and Coronavirus Infection of Malayan Pangolins (Manis javanica) |periódico= Viruses | volume = 11 |número= 11 |páginas= 979 |data=outubro de 2019 | pmid = 31652964 | pmc = 6893680 | doi = 10.3390/v11110979 }}</ref> Em 7 de fevereiro de 2020, foi anunciado que investigadores de [[Guangzhou]] tinham descoberto uma amostra de pangolim com uma sequência de ácido nucleico 99% idêntica ao SARS-CoV-2,<ref>{{citar periódico|vauthors=Cyranoski D |título=Did pangolins spread the China coronavirus to people? |url=https://www.nature.com/articles/d41586-020-00364-2 |periódico=Nature |doi=10.1038/d41586-020-00364-2 |data=7 de fevereiro de 2020}}</ref> com a diferença apenas de um aminoácido.<ref>{{citar periódico|vauthors=Xiao K, Zhai J, Feng Y|título=Isolation and Characterization of 2019-nCoV-like Coronavirus from Malayan Pangolins|data=fevereiro de 2020 |periódico=[[bioRxiv]] |tipo=preprint |doi=10.1101/2020.02.17.951335}}</ref> Embora a lei chinesa proteja os pangolins, a sua captura e comércio ilegais para uso na [[medicina tradicional chinesa]] continuam a ser comuns.<ref>{{citar jornal|jornal=The Telegraph|url=https://www.telegraph.co.uk/science/2016/03/15/pangolins-13-facts-about-the-worlds-most-hunted-animal/|último =Kelly|primeiro =Guy|data=1 de janeiro de 2015|título=Pangolins: 13 facts about the world's most hunted animal|acessodata=9 de março de 2020}}</ref> |
|||
| ⚫ | |||
{{Artigo principal|Pesquisa de vacina para o COVID-19}} |
|||
Em paralelo, microbiólogos e geneticistas no [[Texas]] encontraram evidências de [[rearranjo]] em coronavírus, o que sugere o envolvimento de pangolins na origem do SARS-CoV-2.<ref name="WongRecombination">{{citar periódico|vauthors=Wong MC, Cregeen SJ, Ajami NJ, Petrosino JF|título=Evidence of recombination in coronaviruses implicating pangolin origins of nCoV-2019 |periódico=[[bioRxiv]] |tipo=preprint |doi=10.1101/2020.02.07.939207 |data=fevereiro de 2020}}</ref> No entanto, os coronavírus em pangolins encontrados até hoje partilham apenas 92% do genoma com o SARS-CoV-2, o que é insuficiente para provar que os pangolins sejam o hospediro intermédio. Em comparação, o vírus SARS responsável pelo surto de 2002-2004 partilhava 99,8% do seu genoma com os coronavírus da [[civeta]].<ref name="nature feb2020">{{citar periódico|título= Mystery deepens over animal source of coronavirus|periódico= Nature |volume=579|páginas= 18–19 |data=26 de fevereiro de 2020|doi= 10.1038/d41586-020-00548-w|pmid= 32127703|vauthors=Cyranoski D|número= 7797}}</ref> |
|||
Em 29 de janeiro de 2020, a [[epidemiologista]] da Universidade de Zhejiang, Li Lanjuan, anunciou um prazo de aproximadamente três meses para a produção e teste de uma vacina. A amostra de pacientes permitiram que Li isolasse a sequência do vírus, da qual são necessárias quatro semanas para criar as vacinas, duas semanas para testá-las, seis semanas para aprovação oficial.<ref>{{citar web|título=Leading Chinese epidemiologist: Development of vaccine for novel coronavirus is underway|url=https://youtube.com/watch?v=mo91abdwnR8|data=29 de janeiro de 2020|publicado=China Global Television Network|acessodata=1 de fevereiro de 2020|língua=inglês}}</ref> |
|||
===Filogenética e taxonomia=== |
|||
<!-- infobox genoma --> |
|||
O SARS-CoV-2 pertence a uma grande família de vírus denominada [[coronavírus]]. É um [[Vírus (+)ssRNA|vírus ARN de cadeia simples positiva]] (+ssRNA). Os coronavírus têm a capacidade de causar várias doenças em seres humanos, desde a simples [[constipação]] até doenças mais graves como a [[síndrome respiratória do Médio Oriente]] (MERS). O SARS-CoV-2 é o sétimo coronavírus conhecido a poder infetar seres humanos, sendo os restantes o [[Coronavírus humano 229E|229E]], [[Coronavírus humano NL63|NL63]], [[Coronavírus humano OC43|OC43]], [[Coronavírus humano HKU1|HKU1]], [[MERS-Cov]] e o [[SARS-CoV]] original.<ref name="NEJM">{{citar periódico| vauthors = Zhu N, Zhang D, Wang W, Li X, Yang B, Song J, Zhao X, Huang B, Shi W, Lu R, Niu P, Zhan F, Ma X, Wang D, Xu W, Wu G, Gao GF, Tan W | display-authors = 3 |título= A Novel Coronavirus from Patients with Pneumonia in China, 2019 |periódico= [[The New England Journal of Medicine]] | volume = 382 |número= 8 |páginas= 727–733 |data=fevereiro de 2020 | pmid = 31978945 | doi = 10.1056/NEJMoa2001017 }}</ref> |
|||
Tal como a estirpe que causou o surto de SARS em 2003, o SARS-CoV-2 é um membro do sug-género ''[[Sarbecovirus]]'' ([[betacoronavírus]] linhagem B).<ref name="Hui14Jan2020">{{citar periódico| vauthors = Hui DS, I Azhar E, Madani TA, Ntoumi F, Kock R, Dar O, Ippolito G, Mchugh TD, Memish ZA, Drosten C, Zumla A, Petersen E |título= The continuing 2019-nCoV epidemic threat of novel coronaviruses to global health — The latest 2019 novel coronavirus outbreak in Wuhan, China |periódico= International Journal of Infectious Diseases | volume = 91 |páginas= 264–266 |data=fevereiro de 2020 | pmid = 31953166 | doi = 10.1016/j.ijid.2020.01.009 |display-authors=3}} {{open access}}</ref><ref>{{citar web|url=https://nextstrain.org/groups/blab/sars-like-cov |título=Phylogeny of SARS-like betacoronaviruses |website=nextstrain |acessodata=18 de janeiro de 2020 |arquivourl=https://web.archive.org/web/20200120190511/https://nextstrain.org/groups/blab/sars-like-cov |arquivodata=20 de janeiro de 2020 |urlmorta= não}}</ref><ref name="Wong2019">{{citar periódico| vauthors = Wong AC, Li X, Lau SK, Woo PC |título= Global Epidemiology of Bat Coronaviruses |periódico= Viruses | volume = 11 |número= 2 |páginas= 174 |data=fevereiro de 2019 | pmid = 30791586 | pmc = 6409556 | doi = 10.3390/v11020174 }}</ref> A sua sequência de [[ARN]] tem o comprimento de aproximadamente {{formatnum:30000}} [[nucleobase]]s.<ref name=":gisaid"/> No entanto, o SARS-CoV-2 é o único dos coronavírus a incorporar um local de clivagem polibásico, uma característica que se sabe aumentar a [[Agente patogénico|patogenicidade]] e ritmo reprodutivo de outros vírus.<ref name="Proximal" /><ref>{{citar periódico|vauthors=Walls AC, Park YJ, Tortorici MA, Wall A, McGuire AT, Veesler D|título=Structure, function and antigenicity of the SARS-CoV-2 spike glycoprotein|display-authors=3|data=9 de março de 2020 |periódico=[[Cell (journal)|Cell]] |doi=10.1016/j.cell.2020.02.058 |pmid=32155444}}</ref><ref>{{citar periódico| vauthors = Coutard B, Valle C, de Lamballerie X, Canard B, Seidah NG, Decroly E |título= The spike glycoprotein of the new coronavirus 2019-nCoV contains a furin-like cleavage site absent in CoV of the same clade |periódico= Antiviral Research | volume = 176 |páginas= 104742 |data=fevereiro de 2020 | pmid = 32057769 | doi = 10.1016/j.antiviral.2020.104742|display-authors=3 }}</ref> |
|||
A partir de um número suficiente de [[genoma]]s sequenciados, é possível reconstruir a [[árvore filogenética]] do historial de mutações de uma família de vírus. Em 12 de janeiro de 2020 foram isolados em Wuhan cinco genomas de SARS-CoV-2.<ref name=":gisaid"/><ref>{{citar web|url=http://virological.org/t/initial-genome-release-of-novel-coronavirus/319 |título=Initial genome release of novel coronavirus |data=11 de janeiro de 2020 |website=Virological |acessodata=12 de janeiro de 2020 |arquivourl=https://web.archive.org/web/20200112100227/http://virological.org/t/initial-genome-release-of-novel-coronavirus/319 |arquivodata=12 de janeiro de 2020 |urlmorta= não}}</ref> Em 30 de janeiro de 2020 eram conhecidos 42 genomas.<ref name="NextstrainJanuary">{{citar web|url=https://nextstrain.org/narratives/ncov/sit-rep/2020-01-30|título=Genomic analysis of nCoV spread: Situation report 2020-01-30|vauthors=Bedford T, Neher R, Hadfield N, Hodcroft E, Ilcisin M, Müller N|display-authors=3|website=nextstrain.org|acessodata=18 de março de 2020}}</ref> Uma análise filogenética dessas amostras revelou estarem relacionados até sete mutações com um [[ancestral comum]], o que significa que a primeira ineção em seres humanos ocorreu em novembro ou dezembro de 2019.<ref name="NextstrainJanuary" /> Em 13 de março de 2020 estavam já amostrados e disponíveis publicamente 410 genomas de SARS-CoV-2.<ref name="NextstrainMarch">{{citar web|url=https://nextstrain.org/narratives/ncov/sit-rep/2020-03-13|título=Genomic analysis of COVID-19 spread: Situation report 2020-03-13|vauthors=Hodcroft E, Müller N, Wagner C, Ilcisin M, Hadfield J, Bell SM, Neher R, Bedford T|display-authors=3|website=nextstrain.org|acessodata=18 de março de 2020}}</ref> |
|||
Em 11 defevereiro de 2020, o [[Comitê Internacional de Taxonomia de Vírus|Comité Internacional de Taxonomia de Vírus]] anunciou que, de acordo com as regras vigentes que determinam as relações hierárquicas entre os coronavírus com base nas [[sequência conservada]]s dos [[Ácido nucleico|ácidos nucleicos]], as diferenças entre o SARS-CoV-2 e o SARS-CoV responsável pelo surto de SARS eram insuficientes para serem classificados como duas [[Classificação dos vírus|espécies virais]] diferentes. Desta forma, o SARS-CoV-2 foi classificado como [[estirpe]] dos [[SARSr-CoV|coronavírus associados à síndrome respiratória aguda]] (SARSr-CoV).<ref name="CoronavirusStudyGroup" /> |
|||
=== Biologia estrutural === |
|||
[[Imagem:Coronavirus virion structure.svg|thumb|Ilustração de um virião de coronavírus]] |
|||
Cada [[virião]] de SARS-CoV-2 mede aproximadamente 50–200 [[nanómetro]]s de diâmetro.<ref>{{citar periódico|vauthors=Chen N, Zhou M, Dong X, Qu J, Gong F, Han Y, Qiu Y, Wang J, Liu Y, Wei Y, Sia J, You T, Zhang X, Zhang L |título=Epidemiological and clinical characteristics of 99 cases of 2019 novel coronavirus pneumonia in Wuhan, China: a descriptive study |url=https://www.thelancet.com/journals/lancet/article/PIIS0140-6736(20)30211-7/fulltext |periódico= [[The Lancet]] |volume = 395 |número= 10223 |páginas=507–513 |display-authors=3 |doi=10.1016/S0140-6736(20)30211-7 |pmid=32007143 |data=15 de fevereiro de 2020}}</ref> Tal como outros coronavírus, o SARS-CoV-2 tem quatro [[proteína]]s estruturais, conhecidas como proteínas S (''spike''), E (envelope), M (membrana) e N ([[Cápside|nucleocapsídeo]]). A proteína N contém o genoma ARN e em conjunto as proteínas S, E e M criam o [[envelope viral]].<ref name="WuStructure">{{citar periódico|vauthors=Wu C, Liu Y, Yang Y, Zhang P, Zhong W, Wang Y, Wang Q, Xu Y, Li M, Li X, Zheng M, Chen L, Li H|display-authors=3|título=Analysis of therapeutic targets for SARS-CoV-2 and discovery of potential drugs by computational methods |periódico=Acta Pharmaceutica Sinica B|data=fevereiro de 2020| doi=10.1016/j.apsb.2020.02.008}}</ref> A proteína S é a proteína que permite ao vírus ligar-se à [[membrana celular]] de uma célula hospedeira.<ref name="WuStructure" /> |
|||
As primeiras experiências de modelação de proteínas na proteína S do vírus sugeriram que o SARS-CoV-2 tinha suficiente afinidade com os receptores de [[Enzima conversora da angiotensina 2]] (ACE2) nas células humanas para as usar como mecanismo de penetração celular.<ref>{{citar periódico| vauthors = Xu X, Chen P, Wang J, Feng J, Zhou H, Li X, Zhong W, Hao P | display-authors = 3 |título= Evolution of the novel coronavirus from the ongoing Wuhan outbreak and modeling of its spike protein for risk of human transmission |periódico= Science China Life Sciences | volume = 63 |número= 3 |páginas= 457–460 |data=março de 2020 | pmid = 32009228 | doi = 10.1007/s11427-020-1637-5 }}</ref> Em 22 de janeiro de 2020, um grupo chinês e um grupo norte-americano, de forma independente, conseguiram demonstrar experimentalmente que a ACE2 podia ser o receptor do SARS-CoV-2.<ref name="NatureZhou" /><ref name="Letco22Jan2020">{{citar periódico|vauthors=Letko M, Munster V|título=Functional assessment of cell entry and receptor usage for lineage B β-coronaviruses, including 2019-nCoV|data=janeiro de 2020 |periódico=[[bioRxiv]] |tipo=preprint |doi=10.1101/2020.01.22.915660 }}</ref><ref>{{citar periódico| vauthors = Letko M, Marzi A, Munster V |título= Functional assessment of cell entry and receptor usage for SARS-CoV-2 and other lineage B betacoronaviruses |periódico= Nature Microbiology |data=fevereiro de 2020 | pmid = 32094589 | doi = 10.1038/s41564-020-0688-y }}</ref><ref>{{citar periódico|vauthors=((El Sahly HM))|título=Genomic Characterization of the 2019 Novel Coronavirus |url=https://www.jwatch.org/na50823/2020/02/06/genomic-characterization-2019-novel-coronavirus |periódico=[[The New England Journal of Medicine]] |acessodata=9 de fevereiro de 2020}}</ref><ref name=Gralinski2020>{{citar periódico| vauthors = Gralinski LE, Menachery VD |título= Return of the Coronavirus: 2019-nCoV |periódico= Viruses | volume = 12 |número= 2 |páginas= 135 |data=janeiro de 2020 | pmid = 31991541 | doi = 10.3390/v12020135 }}</ref><ref>{{citar periódico| vauthors = Lu R, Zhao X, Li J, Niu P, Yang B, Wu H, Wang W, Song H, Huang B, Zhu N, Bi Y, Ma X, Zhan F, Wang L, Hu T, Zhou H, Hu Z, Zhou W, Zhao L, Chen J, Meng Y, Wang J, Lin Y, Yuan J, Xie Z, Ma J, Liu WJ, Wang D, Xu W, Holmes EC, Gao GF, Wu G, Chen W, Shi W, Tan W | display-authors = 6 |título= Genomic characterisation and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding |periódico= [[The Lancet]] | volume = 395 |número= 10224 |páginas= 565–574 |data=fevereiro de 2020 | pmid = 32007145 | doi = 10.1016/S0140-6736(20)30251-8 }}</ref> Vários estudos têm demonstrado que o SARS-CoV-2 tem uma maior afinidade com a ACE2 humana do que a estirpe de SARS original.<ref name="SCI-20200219">{{citar periódico| vauthors = Wrapp D, Wang N, Corbett KS, Goldsmith JA, Hsieh CL, Abiona O, Graham BS, McLellan JS | display-authors = 6 |título= Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation |periódico= Science | volume = 367 |número= 6483 |páginas= 1260–1263 |data=fevereiro de 2020 | pmid = 32075877 | doi = 10.1126/science.abb2507 }}</ref> O SARS-CoV-2 pode também usar a proteína [[basigina]] para penetrar nas células do hospedeiro.<ref>{{citar periódico| vauthors= Wang K, Chen W, Zhou YS, Lian JQ, Zhang Z, Du P, Gong L, Zhang Y, Cui HY, Geng JJ, Wang B, Sun XX, Wang CF, Yang X, Lin P, Deng YQ, Wei D, Yang XM, Zhu YM, Zhang K, Zheng ZH, Miao JL, Guo T, Shi Y, Zhang J, Fu L, Wang QY, Bian H, Zhu P, Chen ZN | display-authors=3 |título= SARS-CoV-2 invades host cells via a novel route: CD147-spike protein |periódico= [[bioRxiv]]|tipo=preprint | doi = 10.1101/2020.03.14.988345 |ano=2020 }}</ref> Para a penetração do SARS-CoV-2 também é fundamental o ''priming'' inicial da proteína S através de [[TMPRSS2]].<ref name="HoffmanCell">{{citar periódico|título=SARS-CoV-2 Cell Entry Depends on ACE2 and TMPRSS2 and Is Blocked by a Clinically Proven Protease Inhibitor |vauthors=Hoffman M, Kliene-Weber H, Krüger N, Herrler T, Erichsen S, Schiergens TS, Herrler G, Wu NH, Nitsche A, Müller MA, Drosten C, Pöhlmann S|display-authors=3|data=16 de abril de 2020|periódico=[[Cell (journal)|Cell]] |volume=181|páginas=1–10|doi=10.1016/j.cell.2020.02.052 |pmid=32142651}}</ref> O SARS-CoV-2 produz pelo menos três [[Fator de virulência|fatores de virulência]] que promovem a libertação de novos viriões das células hospediras e inibem a [[resposta imunitária]].<ref name="WuStructure" /> |
|||
==Epidemiologia== |
|||
| ⚫ | |||
[[File:Novel Coronavirus SARS-CoV-2 (49597020718).jpg|thumb|[[Microscópio eletrónico de transmissão|Micrografia por MET]] de viriões de SARS-CoV-2 (a vermelho) isolados de um paciente]] |
|||
Com base na baixa variabilidade verificada entre as sequências [[genómica]]s conhecidas de SARS-CoV-2, pensa-se que a estirpe tenha sido detectada pelas autoridades apenas poucas semanas após ter emergido entre a população humana no fim de 2019.<ref name="early" /><ref>{{citar web|url=https://www.genengnews.com/insights/what-we-know-today-about-coronavirus-sars-cov-2-and-where-do-we-go-from-here/|título=What We Know Today about Coronavirus SARS-CoV-2 and Where Do We Go from Here|vauthors=Oberholzer M, Febbo P|data=19 de fevereiro de 2020|website=[[Mary Ann Liebert|Genetic Engineering and Biotechnology News]]|acessodata=13 de março de 2020}}</ref> O caso mais antigo de infeção humana de que se tem conhecimento data de 17 de novembro de 2029.<ref>{{citar jornal|título=Coronavirus: China's first confirmed Covid-19 case traced back to November 17| url=https://www.scmp.com/news/china/society/article/3074991/coronavirus-chinas-first-confirmed-covid-19-case-traced-back| website=[[South China Morning Post]]| vauthors=Ma J|data=13 de março de 2020|acessodata=16 de março de 2020}}</ref> Posteriormente, o vírus espalhou-se para todas as províncias da China e mais de cem países em todos os continentes.<ref name="JHU_ticker" /> Em todos os continentes foram confrimadas transmissões entre seres humanos.<ref name="Chan24Jan2020">{{citar periódico| vauthors = Chan JF, Yuan S, Kok KH, To KK, Chu H, Yang J, Xing F, Liu J, Yip CC, Poon RW, Tsoi HW, Lo SK, Chan KH, Poon VK, Chan WM, Ip JD, Cai JP, Cheng VC, Chen H, Hui CK, Yuen KY | display-authors = 6 |título= A familial cluster of pneumonia associated with the 2019 novel coronavirus indicating person-to-person transmission: a study of a family cluster |periódico= [[The Lancet]] | volume = 395 |número= 10223 |páginas= 514–523 |data=fevereiro de 2020 | pmid = 31986261 | doi = 10.1016/S0140-6736(20)30154-9 }}</ref><ref>{{citar periódico| vauthors = Rothe C, Schunk M, Sothmann P, Bretzel G, Froeschl G, Wallrauch C, Zimmer T, Thiel V, Janke C, Guggemos W, Seilmaier M, Drosten C, Vollmar P, Zwirglmaier K, Zange S, Wölfel R, Hoelscher M | display-authors = 6 |título= Transmission of 2019-nCoV Infection from an Asymptomatic Contact in Germany |periódico= [[The New England Journal of Medicine]] | volume = 382 |número= 10 |páginas= 970–971 |data=março de 2020 | pmid = 32003551 | doi = 10.1056/NEJMc2001468 }}</ref><ref>{{citar periódico|título=The Coronavirus Is Now Infecting More People Outside China|url=https://www.wired.com/story/the-coronavirus-is-now-infecting-more-people-outside-china/|vauthors=Molteni M|data=30 de janeiro de 2020|periódico=[[Wired (magazine)|Wired]]|acessodata=13 de março de 2020}}</ref><ref>{{citar jornal|título=Coronavirus: Singapore reports first cases of local transmission; 4 out of 6 new cases did not travel to China| url=https://www.straitstimes.com/singapore/coronavirus-spore-reports-first-cases-of-local-transmission-4-out-of-6-new-cases-did-not| vauthors=Khalik S|data=4 de fevereiro de 2020|acessodata=5 de fevereiro de 2020|jornal=[[The Straits Times]]|arquivourl=https://web.archive.org/web/20200204101450/https://www.straitstimes.com/singapore/coronavirus-spore-reports-first-cases-of-local-transmission-4-out-of-6-new-cases-did-not|arquivodata=4 de fevereiro de 2020|urlmorta= não}}</ref><ref>{{citar jornal|url=https://www.reuters.com/article/us-china-health-ecuador/ecuador-confirms-five-new-cases-of-coronavirus-all-close-to-initial-patient-idUSKBN20O2EE|título=Ecuador confirms five new cases of coronavirus, all close to initial patient|obra=[[Reuters]]|data=2 de março de 2020|acessodata=5 de março de 2020}}</ref><ref>{{citar jornal|url=https://www.reuters.com/article/us-health-coronavirus-algeria/algeria-confirms-two-more-coronavirus-cases-idUSKBN20P00S|título=Algeria confirms two more coronavirus cases|obra=[[Reuters]]|data=2 de março de 2020|acessodata=5 de março de 2020}}</ref> Em 30 de janeiro de 2020, a OMS declarou o SARS-CoV-2 uma [[Emergência de Saúde Pública de Âmbito Internacional]],<ref name="NYT 20200130">{{citar jornal|título=W.H.O. Declares Global Emergency as Wuhan Coronavirus Spreads | vauthors=Wee SL, ((McNeil Jr. DG)), Hernández JC| website=[[The New York Times]] |data=30 de janeiro de 2020 | url=https://www.nytimes.com/2020/01/30/health/coronavirus-world-health-organization.html |acessodata=30 de janeiro de 2020 |arquivourl=https://web.archive.org/web/20200130195011/https://www.nytimes.com/2020/01/30/health/coronavirus-world-health-organization.html |arquivodata=30 de janeiro de 2020 |urlmorta= não}}</ref><ref>{{citar comunicado de imprensa|título=Statement on the second meeting of the International Health Regulations (2005) Emergency Committee regarding the outbreak of novel coronavirus (2019-nCoV) | website=[[World Health Organization]] (WHO) |data=30 de janeiro de 2020 | url=https://www.who.int/news-room/detail/30-01-2020-statement-on-the-second-meeting-of-the-international-health-regulations-(2005)-emergency-committee-regarding-the-outbreak-of-novel-coronavirus-(2019-ncov) |acessodata=30 de janeiro de 2020 |arquivourl=https://web.archive.org/web/20200131005904/https://www.who.int/news-room/detail/30-01-2020-statement-on-the-second-meeting-of-the-international-health-regulations-(2005)-emergency-committee-regarding-the-outbreak-of-novel-coronavirus-(2019-ncov) |arquivodata=31 de janeiro de 2020 |urlmorta= não}}</ref> e em 11 de março uma [[pandemia]].<ref>{{citar jornal|título= Coronavirus Declared Pandemic by World Health Organization | url=https://www.wsj.com/articles/u-s-coronavirus-cases-top-1-000-11583917794 | vauthors= McKay B, Calfas J, Ansari T | website=[[The Wall Street Journal]] |data=11 de março de 2020 |acessodata=12 de março de 2020 |urlmorta= não}}</ref><ref>{{citar comunicado de imprensa|título=WHO Director-General's opening remarks at the media briefing on COVID-19 - 11 March 2020 | website=[[World Health Organization]] (WHO) |data=11 de março de 2020 | url=https://www.who.int/dg/speeches/detail/who-director-general-s-opening-remarks-at-the-media-briefing-on-covid-19---11-march-2020 |acessodata=12 de março de 2020 |urlmorta= não}}</ref> |
|||
À data de {{Número de casos de COVID-19|data|ref=no}} a pandemia tinha resultado em {{Número de casos de COVID-19|confirmados}} casos confirmados de infeção em todo o mundo, dos quais {{Número de casos de COVID-19|mortes}} tinham resultado em morte e {{Número de casos de COVID-19|recuperados}} tinham recuperado. Embora a proporção de infeções que resulta em infeções confirmadas ou que avolui para doenga diagnosticável permaneça incerta,<ref>{{citar jornal|url=https://www.statnews.com/2020/01/30/limited-data-may-skew-assumptions-severity-coronavirus-outbreak/|título=Limited data on coronavirus may be skewing assumptions about severity|vauthors=Branswell H|data=30 de janeiro de 2020|publicado=[[Stat (website)|STAT]]|acessodata=13 de março de 2020|arquivourl=https://web.archive.org/web/20200201161634/https://www.statnews.com/2020/01/30/limited-data-may-skew-assumptions-severity-coronavirus-outbreak/|arquivodata=1 de fevereiro de 2020|urlmorta= não}}</ref> um modelo matemático estimou que o número de pessoas infetadas apenas em Wuhan em 25 de janeiro de 2020 tenha sido de {{formatnum:75815}}, data em que as infeções confirmadas eram bastante inferiores.<ref name="pmid32014114">{{citar periódico| vauthors = Wu JT, Leung K, Leung GM |título= Nowcasting and forecasting the potential domestic and international spread of the 2019-nCoV outbreak originating in Wuhan, China: a modelling study |periódico= [[The Lancet]] | volume = 395 |número= 10225 |páginas= 689–697 |data=fevereiro de 2020 | pmid = 32014114 | doi = 10.1016/S0140-6736(20)30260-9 }}</ref> |
|||
Estima-se que o ritmo reprodutivo (<math>R_0</math>) do vírus seja de entre 1,4 e 3,9.<ref>{{citar periódico| vauthors = Li Q, Guan X, Wu P, Wang X, Zhou L, Tong Y, Ren R, Leung KS, Lau EH, Wong JY, Xing X, Xiang N, Wu Y, Li C, Chen Q, Li D, Liu T, Zhao J, Li M, Tu W, Chen C, Jin L, Yang R, Wang Q, Zhou S, Wang R, Liu H, Luo Y, Liu Y, Shao G, Li H, Tao Z, Yang Y, Deng Z, Liu B, Ma Z, Zhang Y, Shi G, Lam TT, Wu JT, Gao GF, Cowling BJ, Yang B, Leung GM, Feng Z | display-authors = 6 |título= Early Transmission Dynamics in Wuhan, China, of Novel Coronavirus-Infected Pneumonia |periódico= [[The New England Journal of Medicine]] |data=janeiro de 2020 | pmid = 31995857 | doi = 10.1056/NEJMoa2001316 }}</ref><ref>{{citar periódico| vauthors = Riou J, Althaus CL |título= Pattern of early human-to-human transmission of Wuhan 2019 novel coronavirus (2019-nCoV), December 2019 to January 2020 |periódico= Euro Surveillance | volume = 25 |número= 4 |data=janeiro de 2020 | pmid = 32019669 | pmc = 7001239 | doi = 10.2807/1560-7917.ES.2020.25.4.2000058 }}</ref> Isto significa que é esperado que cada infeção pelo vírus resulte em 1,4 a 3,9 novas ineções quando nenhum membro da comunidade é [[Imunidade|imune]] e não é tomada nenhuma medida preventiva. |
|||
== Ver também == |
|||
| ⚫ | |||
{{Referências|col=2}} |
{{Referências|col=2}} |
||
=== Bibliografia === |
|||
* {{citar periódico| vauthors = Brüssow H |título= The Novel Coronavirus – A Snapshot of Current Knowledge |periódico= Microbial Biotechnology | volume = 2020 |páginas= 1–6 |data=março de 2020 | doi = 10.1111/1751-7915.13557 | pmid = 32144890 }} |
|||
* {{citar periódico| vauthors = Habibzadeh P, Stoneman EK |título= The Novel Coronavirus: A Bird's Eye View |periódico= The International Journal of Occupational and Environmental Medicine | volume = 11 |número= 2 |páginas= 65–71 |data=fevereiro de 2020 | pmid = 32020915 | doi = 10.15171/ijoem.2020.1921 }} |
|||
* {{citar relatório| vauthors = ((World Health Organization)) |título= Laboratory testing for coronavirus disease 2019 (COVID-19) in suspected human cases: interim guidance, 2 March 2020 |data=2 de março de 2020 |autorlink = World Health Organization |publicado= World Health Organization | hdl = 10665/331329 | id = WHO/COVID-19/laboratory/2020.4. License: CC BY-NC-SA 3.0 }} |
|||
== Ligações externas == |
== Ligações externas == |
||
{{Commonscat}} |
|||
{{Wikispecies|Severe acute respiratory syndrome coronavirus 2}} |
|||
*[https://www.who.int/publications-detail/surveillance-case-definitions-for-human-infection-with-novel-coronavirus-(ncov) OMS sobre o novo coronavírus] (em inglês. Consultado em 20 de janeiro de 2020. |
*[https://www.who.int/publications-detail/surveillance-case-definitions-for-human-infection-with-novel-coronavirus-(ncov) OMS sobre o novo coronavírus] (em inglês. Consultado em 20 de janeiro de 2020. |
||
Revisão das 05h34min de 21 de março de 2020
| Coronavírus da Síndrome Respiratória Aguda Grave 2 | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| Classificação científica | |||||||||||
| |||||||||||
| Distribuição geográfica | |||||||||||
 Wuhan, China; localização principal do início do surto.
| |||||||||||
O vírus da síndrome respiratória aguda grave 2 (SARS-CoV-2),[1][2] anteriormente denominado de forma provisória "2019-nCoV",[3][4][5] é um vírus ARN de cadeia simples positiva.[6][7] É contagioso entre seres humanos e é a causa da doença COVID-19, da qual existe uma pandemia em curso .[8][9]
O SARS-CoV-2 apresenta proximidade genética com os coronavírus de morcegos, dos quais se terá provavelmente originado.[10] [11][12] Pensa-se que antes de ser introduzido aos seres humanos tenha também estado envolvido um reservatório animal intermédio, como o pangolim.[13][14] Do ponto de vista taxonómico, o SARS-CoV-2 está classificado como estirpe da espécie coronavírus relacionado com a síndrome respiratória aguda grave (SARSr-CoV).[1]
Uma vez que a estirpe foi descoberta em Wuhan, na China, é por vezes denominada "vírus de Wuhan virus" ou "coronavírus de Wuhan",[15][16][17][18] embora a Organização Mundial de Saúde (OMS) desaconselhe a utilização de nomes baseados na localização.[19] Para evitar confusão com a doença síndrome respiratória aguda grave (SARS), em comunicados públicos a OMS por vezes refere-se ao vírus como "vírus responsável pela COVID-19" ou "vírus da COVID-19".[20] Tanto o vírus como a doença são muitas vezes denominados "coronavírus" pelo público em geral, embora cientistas e jornalistas usem termos mais precisos.[21]
Virologia
Infeção
A transmissão do SARS-CoV-2 entre seres humanos foi confirmada pela primeira vez durante a pandemia de coronavírus de 2019-20.[9] A principal forma de transmissão são gotículas produzidas no sistema respiratório e expulsas ao tossir ou espirrar até um raio de 1,8 m.[22][23] Outra possível causa de infeção é o contacto indireto através de superfícies contaminadas.[24] A investigação preliminar sugere que o vírus possa permanecer ativo em plástico e aço até três dias, embora não consiga sobreviver em cartão mais do que um dia ou em cobre mais do que quatro horas.[25] Foi também observado ARN viral em fezes de pacientes infetados.[26]
Ainda não é claro se o vírus é infecioso durante o período de incubação.[27] Em 1 de fevereiro de 2020 a OMS indicava que "a transmissão a partir de casos assintomáticos provavelmente não é uma das principais formas de transmissão".[28] Acredita-se que a maior parte das infeções em seres humanos seja o resultado de transmissão entre pessoas que manifestam sintomas de COVID-19. No entanto, um modelo epidemiológico do início so surto na China sugere que a transmissão pré-sintomática pode ser típica entre as infeções documentadas.[29]
Reservatório
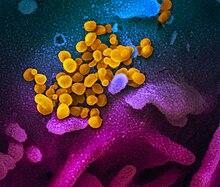
A OMS considera serem os morcegos o mais provável reservatório natural de SARS-CoV-2,[30] embora algumas diferenças entre os coronavírus dos morcegos e o SARS-CoV-2 sugiram que os seres humanos foram infetados através de um hospedeiro intermédio.[31]
As primeiras infeções conhecidas pela estirpe SARS-CoV-2 foram descobertas em Wuhan, na China.[10] Ainda não é claro qual foi a fonte original de transmissão viral para os seres humanos nem quando é que a estirpe se tornou patogénica,[32][33][34][35] embora já se tenha determinado que a estirpe é de origem natural.[35] A investigação do reservatório natural da estirpe de vírus que causou a pandemia de SARS em 2002-2004 permitiu a descoberta de diversos coronavírus semelhantes à SARS em morcegos, a maior parte com origem no género Rhinolophus dos morcegos-de-ferradura, e duas sequências de ácido nucleico encontradas em amostras de Rhinolophus sinicus revelaram uma semelhança de 80% em relação ao SARS-CoV-2.[12][36][37] Uma terceira sequência de ácido nucleico de Rhinolophus affinis recolhida na província de Yunnan revelou uma semelhança de 96% em relação ao SARS-CoV-2.[10][38]
Um estudo metagenómico publicado em 2019 concliui que o SARS-CoV, a estirpe que causa a SARS, era o coronavírus com maior distribuição entre uma amostra de pangolins-malaio.[39] Em 7 de fevereiro de 2020, foi anunciado que investigadores de Guangzhou tinham descoberto uma amostra de pangolim com uma sequência de ácido nucleico 99% idêntica ao SARS-CoV-2,[40] com a diferença apenas de um aminoácido.[41] Embora a lei chinesa proteja os pangolins, a sua captura e comércio ilegais para uso na medicina tradicional chinesa continuam a ser comuns.[42]
Em paralelo, microbiólogos e geneticistas no Texas encontraram evidências de rearranjo em coronavírus, o que sugere o envolvimento de pangolins na origem do SARS-CoV-2.[43] No entanto, os coronavírus em pangolins encontrados até hoje partilham apenas 92% do genoma com o SARS-CoV-2, o que é insuficiente para provar que os pangolins sejam o hospediro intermédio. Em comparação, o vírus SARS responsável pelo surto de 2002-2004 partilhava 99,8% do seu genoma com os coronavírus da civeta.[31]
Filogenética e taxonomia
O SARS-CoV-2 pertence a uma grande família de vírus denominada coronavírus. É um vírus ARN de cadeia simples positiva (+ssRNA). Os coronavírus têm a capacidade de causar várias doenças em seres humanos, desde a simples constipação até doenças mais graves como a síndrome respiratória do Médio Oriente (MERS). O SARS-CoV-2 é o sétimo coronavírus conhecido a poder infetar seres humanos, sendo os restantes o 229E, NL63, OC43, HKU1, MERS-Cov e o SARS-CoV original.[44]
Tal como a estirpe que causou o surto de SARS em 2003, o SARS-CoV-2 é um membro do sug-género Sarbecovirus (betacoronavírus linhagem B).[45][46][47] A sua sequência de ARN tem o comprimento de aproximadamente 30 000 nucleobases.[7] No entanto, o SARS-CoV-2 é o único dos coronavírus a incorporar um local de clivagem polibásico, uma característica que se sabe aumentar a patogenicidade e ritmo reprodutivo de outros vírus.[35][48][49]
A partir de um número suficiente de genomas sequenciados, é possível reconstruir a árvore filogenética do historial de mutações de uma família de vírus. Em 12 de janeiro de 2020 foram isolados em Wuhan cinco genomas de SARS-CoV-2.[7][50] Em 30 de janeiro de 2020 eram conhecidos 42 genomas.[51] Uma análise filogenética dessas amostras revelou estarem relacionados até sete mutações com um ancestral comum, o que significa que a primeira ineção em seres humanos ocorreu em novembro ou dezembro de 2019.[51] Em 13 de março de 2020 estavam já amostrados e disponíveis publicamente 410 genomas de SARS-CoV-2.[52]
Em 11 defevereiro de 2020, o Comité Internacional de Taxonomia de Vírus anunciou que, de acordo com as regras vigentes que determinam as relações hierárquicas entre os coronavírus com base nas sequência conservadas dos ácidos nucleicos, as diferenças entre o SARS-CoV-2 e o SARS-CoV responsável pelo surto de SARS eram insuficientes para serem classificados como duas espécies virais diferentes. Desta forma, o SARS-CoV-2 foi classificado como estirpe dos coronavírus associados à síndrome respiratória aguda (SARSr-CoV).[1]
Biologia estrutural

Cada virião de SARS-CoV-2 mede aproximadamente 50–200 nanómetros de diâmetro.[53] Tal como outros coronavírus, o SARS-CoV-2 tem quatro proteínas estruturais, conhecidas como proteínas S (spike), E (envelope), M (membrana) e N (nucleocapsídeo). A proteína N contém o genoma ARN e em conjunto as proteínas S, E e M criam o envelope viral.[54] A proteína S é a proteína que permite ao vírus ligar-se à membrana celular de uma célula hospedeira.[54]
As primeiras experiências de modelação de proteínas na proteína S do vírus sugeriram que o SARS-CoV-2 tinha suficiente afinidade com os receptores de Enzima conversora da angiotensina 2 (ACE2) nas células humanas para as usar como mecanismo de penetração celular.[55] Em 22 de janeiro de 2020, um grupo chinês e um grupo norte-americano, de forma independente, conseguiram demonstrar experimentalmente que a ACE2 podia ser o receptor do SARS-CoV-2.[10][56][57][58][59][60] Vários estudos têm demonstrado que o SARS-CoV-2 tem uma maior afinidade com a ACE2 humana do que a estirpe de SARS original.[61] O SARS-CoV-2 pode também usar a proteína basigina para penetrar nas células do hospedeiro.[62] Para a penetração do SARS-CoV-2 também é fundamental o priming inicial da proteína S através de TMPRSS2.[63] O SARS-CoV-2 produz pelo menos três fatores de virulência que promovem a libertação de novos viriões das células hospediras e inibem a resposta imunitária.[54]
Epidemiologia

Com base na baixa variabilidade verificada entre as sequências genómicas conhecidas de SARS-CoV-2, pensa-se que a estirpe tenha sido detectada pelas autoridades apenas poucas semanas após ter emergido entre a população humana no fim de 2019.[32][64] O caso mais antigo de infeção humana de que se tem conhecimento data de 17 de novembro de 2029.[65] Posteriormente, o vírus espalhou-se para todas as províncias da China e mais de cem países em todos os continentes.[66] Em todos os continentes foram confrimadas transmissões entre seres humanos.[9][67][68][69][70][71] Em 30 de janeiro de 2020, a OMS declarou o SARS-CoV-2 uma Emergência de Saúde Pública de Âmbito Internacional,[8][72] e em 11 de março uma pandemia.[73][74]
À data de 3 de março de 2024 a pandemia tinha resultado em 774 834 237[66] casos confirmados de infeção em todo o mundo, dos quais 7 037 007[66] tinham resultado em morte e [66] tinham recuperado. Embora a proporção de infeções que resulta em infeções confirmadas ou que avolui para doenga diagnosticável permaneça incerta,[75] um modelo matemático estimou que o número de pessoas infetadas apenas em Wuhan em 25 de janeiro de 2020 tenha sido de 75 815, data em que as infeções confirmadas eram bastante inferiores.[76]
Estima-se que o ritmo reprodutivo () do vírus seja de entre 1,4 e 3,9.[77][78] Isto significa que é esperado que cada infeção pelo vírus resulte em 1,4 a 3,9 novas ineções quando nenhum membro da comunidade é imune e não é tomada nenhuma medida preventiva.
Ver também
Referências
- ↑ «Coronavirus disease named Covid-19». BBC News Online. 11 de fevereiro de 2020. Consultado em 15 de fevereiro de 2020. Cópia arquivada em 15 de fevereiro de 2020
- ↑ World Health Organization (2020). Surveillance case definitions for human infection with novel coronavirus (nCoV): interim guidance v1, January 2020 (Relatório). World Health Organization. hdl:10665/330376. WHO/2019-nCoV/Surveillance/v2020.1
- ↑ «Healthcare Professionals: Frequently Asked Questions and Answers». United States Centers for Disease Control and Prevention (CDC). 11 de fevereiro de 2020. Consultado em 15 de fevereiro de 2020. Arquivado do original em 14 de fevereiro de 2020
- ↑ «About Novel Coronavirus (2019-nCoV)». United States Centers for Disease Control and Prevention (CDC). 11 de fevereiro de 2020. Consultado em 25 de fevereiro de 2020. Arquivado do original em 11 de fevereiro de 2020
- ↑ «New-type coronavirus causes pneumonia in Wuhan: expert». Xinhua. Consultado em 9 de janeiro de 2020. Cópia arquivada em 9 de janeiro de 2020
- ↑ a b c «CoV2020». GISAID EpifluDB. Consultado em 12 de janeiro de 2020. Cópia arquivada em 12 de janeiro de 2020 Verifique o valor de
|url-access=registration(ajuda) - ↑ a b Wee SL, McNeil Jr. DG, Hernández JC (30 de janeiro de 2020). «W.H.O. Declares Global Emergency as Wuhan Coronavirus Spreads». The New York Times. Consultado em 30 de janeiro de 2020. Cópia arquivada em 30 de janeiro de 2020
- ↑ a b c Chan JF, Yuan S, Kok KH, To KK, Chu H, Yang J, et al. (fevereiro de 2020). «A familial cluster of pneumonia associated with the 2019 novel coronavirus indicating person-to-person transmission: a study of a family cluster». The Lancet. 395 (10223): 514–523. PMID 31986261. doi:10.1016/S0140-6736(20)30154-9
- ↑ a b c d Zhou P, Yang XL, Wang XG, Hu B, Zhang L, Zhang W, et al. (fevereiro de 2020). «A pneumonia outbreak associated with a new coronavirus of probable bat origin». Nature. 579 (7798): 270–273. PMID 32015507. doi:10.1038/s41586-020-2012-7
- ↑ Perlman S (fevereiro de 2020). «Another Decade, Another Coronavirus». The New England Journal of Medicine. 382 (8): 760–762. PMID 31978944. doi:10.1056/NEJMe2001126
- ↑ a b Benvenuto D, Giovanetti M, Ciccozzi A, Spoto S, Angeletti S, Ciccozzi M (abril de 2020). «The 2019-new coronavirus epidemic: Evidence for virus evolution». Journal of Medical Virology. 92 (4): 455–459. PMID 31994738. doi:10.1002/jmv.25688
- ↑ World Health Organization (2020). Novel Coronavirus (2019-nCoV): situation report, 22 (Relatório). World Health Organization. hdl:10665/330991
- ↑ Shield C (7 de fevereiro de 2020). «Coronavirus: From bats to pangolins, how do viruses reach us?». Deutsche Welle. Consultado em 13 de março de 2020
- ↑ Huang P (22 de janeiro de 2020). «How Does Wuhan Coronavirus Compare with MERS, SARS and the Common Cold?». NPR. Consultado em 3 de fevereiro de 2020. Cópia arquivada em 2 de fevereiro de 2020
- ↑ Fox D (24 de janeiro de 2020). «What you need to know about the Wuhan coronavirus». Nature. ISSN 0028-0836. doi:10.1038/d41586-020-00209-y
- ↑ Yam K (12 de março de 2020). «GOP lawmakers continue to use 'Wuhan virus' or 'Chinese coronavirus'». NBC News. Consultado em 19 de março de 2020
- ↑ Dorman S (11 de março de 2020). «McCarthy knocks Dems after they claim saying 'Chinese coronavirus' is racist». Fox News. Consultado em 12 de março de 2020
- ↑ World Health Organization (2015). World Health Organization best practices for the naming of new human infectious diseases (Relatório). World Health Organization. hdl:10665/163636. WHO/HSE/FOS/15.1
- ↑ «Naming the coronavirus disease (COVID-2019) and the virus that causes it». World Health Organization. Consultado em 24 de fevereiro de 2020.
From a risk communications perspective, using the name SARS can have unintended consequences in terms of creating unnecessary fear for some populations.... For that reason and others, WHO has begun referring to the virus as "the virus responsible for COVID-19" or "the COVID-19 virus" when communicating with the public. Neither of these designations are [sic] intended as replacements for the official name of the virus as agreed by the ICTV.
- ↑ Harmon A (4 de março de 2020). «We Spoke to Six Americans with Coronavirus». The New York Times. Consultado em 16 de março de 2020
- ↑ Edwards E (25 de janeiro de 2020). «How does coronavirus spread?». NBC News. Consultado em 13 de março de 2020. Cópia arquivada em 28 de janeiro de 2020
- ↑ «How COVID-19 Spreads». U.S. Centers for Disease Control and Prevention (CDC). 27 de janeiro de 2020. Consultado em 29 de janeiro de 2020. Cópia arquivada em 28 de janeiro de 2020
- ↑ «Getting your workplace ready for COVID-19» (PDF). World Health Organization. 27 de fevereiro de 2020. Consultado em 3 de março de 2020
- ↑ van Doremalen N, Bushmaker T, Morris DH, et al. (17 de março de 2020). «Correspondence: Aerosol and Surface Stability of SARS-CoV-2 as Compared with SARS-CoV-1». The New England Journal of Medicine. PMID 32182409. doi:10.1056/NEJMc2004973
- ↑ Holshue ML, DeBolt C, Lindquist S, et al. (março de 2020). «First Case of 2019 Novel Coronavirus in the United States». The New England Journal of Medicine. 382 (10): 929–936. PMID 32004427. doi:10.1056/NEJMoa2001191
- ↑ Kupferschmidt K (fevereiro de 2020). «Study claiming new coronavirus can be transmitted by people without symptoms was flawed». Science. doi:10.1126/science.abb1524
- ↑ World Health Organization (2020). Novel Coronavirus (2019-nCoV): situation report, 12 (Relatório). World Health Organization. hdl:10665/330777
- ↑ Li R, Pei S, Chen B, et al. (16 de março de 2020). «Substantial undocumented infection facilitates the rapid dissemination of novel coronavirus (SARS-CoV2)». Science: eabb3221. PMID 32179701. doi:10.1126/science.abb3221. Consultado em 17 de março de 2020
- ↑ «Report of the WHO-China Joint Mission on Coronavirus Disease 2019 (COVID-19)» (PDF). World Health Organization (WHO). 24 de fevereiro de 2020. Consultado em 5 de março de 2020
- ↑ a b Cyranoski D (26 de fevereiro de 2020). «Mystery deepens over animal source of coronavirus». Nature. 579 (7797): 18–19. PMID 32127703. doi:10.1038/d41586-020-00548-w
- ↑ a b Cohen J (janeiro de 2020). «Wuhan seafood market may not be source of novel virus spreading globally». Science. ISSN 0036-8075. doi:10.1126/science.abb0611
- ↑ Eschner K (28 de janeiro de 2020). «We're still not sure where the Wuhan coronavirus really came from». Popular Science. Consultado em 30 de janeiro de 2020. Cópia arquivada em 29 de janeiro de 2020
- ↑ Yu WB, Tang GD, Zhang L, Corlett RT (21 de fevereiro de 2020). «Decoding evolution and transmissions of novel pneumonia coronavirus using the whole genomic data». doi:10.12074/202002.00033 (inativo 26 de fevereiro de 2020)
- ↑ a b c Andersen KG, Rambaut A, Lipkin WI, et al. (17 de março de 2020). «Correspondence: The proximal origin of SARS-CoV-2». Nature Medicine. doi:10.1038/s41591-020-0820-9. Consultado em 18 de março de 2020
- ↑ «Bat SARS-like coronavirus isolate bat-SL-CoVZC45, complete genome». National Center for Biotechnology Information (NCBI). 15 de fevereiro de 2020. Consultado em 15 de fevereiro de 2020
- ↑ «Bat SARS-like coronavirus isolate bat-SL-CoVZXC21, complete genome». National Center for Biotechnology Information (NCBI). 15 de fevereiro de 2020. Consultado em 15 de fevereiro de 2020
- ↑ «Bat coronavirus isolate RaTG13, complete genome». National Center for Biotechnology Information (NCBI). 10 de fevereiro de 2020. Consultado em 5 de março de 2020
- ↑ Liu P, Chen W, Chen JP (outubro de 2019). «Viral Metagenomics Revealed Sendai Virus and Coronavirus Infection of Malayan Pangolins (Manis javanica)». Viruses. 11 (11). 979 páginas. PMC 6893680
 . PMID 31652964. doi:10.3390/v11110979
. PMID 31652964. doi:10.3390/v11110979
- ↑ Cyranoski D (7 de fevereiro de 2020). «Did pangolins spread the China coronavirus to people?». Nature. doi:10.1038/d41586-020-00364-2
- ↑ Xiao K, Zhai J, Feng Y (fevereiro de 2020). «Isolation and Characterization of 2019-nCoV-like Coronavirus from Malayan Pangolins». bioRxiv (preprint). doi:10.1101/2020.02.17.951335
- ↑ Kelly, Guy (1 de janeiro de 2015). «Pangolins: 13 facts about the world's most hunted animal». The Telegraph. Consultado em 9 de março de 2020
- ↑ Wong MC, Cregeen SJ, Ajami NJ, Petrosino JF (fevereiro de 2020). «Evidence of recombination in coronaviruses implicating pangolin origins of nCoV-2019». bioRxiv (preprint). doi:10.1101/2020.02.07.939207
- ↑ Zhu N, Zhang D, Wang W, et al. (fevereiro de 2020). «A Novel Coronavirus from Patients with Pneumonia in China, 2019». The New England Journal of Medicine. 382 (8): 727–733. PMID 31978945. doi:10.1056/NEJMoa2001017
- ↑ Hui DS, I Azhar E, Madani TA, et al. (fevereiro de 2020). «The continuing 2019-nCoV epidemic threat of novel coronaviruses to global health — The latest 2019 novel coronavirus outbreak in Wuhan, China». International Journal of Infectious Diseases. 91: 264–266. PMID 31953166. doi:10.1016/j.ijid.2020.01.009

- ↑ «Phylogeny of SARS-like betacoronaviruses». nextstrain. Consultado em 18 de janeiro de 2020. Cópia arquivada em 20 de janeiro de 2020
- ↑ Wong AC, Li X, Lau SK, Woo PC (fevereiro de 2019). «Global Epidemiology of Bat Coronaviruses». Viruses. 11 (2). 174 páginas. PMC 6409556
 . PMID 30791586. doi:10.3390/v11020174
. PMID 30791586. doi:10.3390/v11020174
- ↑ Walls AC, Park YJ, Tortorici MA, et al. (9 de março de 2020). «Structure, function and antigenicity of the SARS-CoV-2 spike glycoprotein». Cell. PMID 32155444. doi:10.1016/j.cell.2020.02.058
- ↑ Coutard B, Valle C, de Lamballerie X, et al. (fevereiro de 2020). «The spike glycoprotein of the new coronavirus 2019-nCoV contains a furin-like cleavage site absent in CoV of the same clade». Antiviral Research. 176. 104742 páginas. PMID 32057769. doi:10.1016/j.antiviral.2020.104742
- ↑ «Initial genome release of novel coronavirus». Virological. 11 de janeiro de 2020. Consultado em 12 de janeiro de 2020. Cópia arquivada em 12 de janeiro de 2020
- ↑ a b Bedford T, Neher R, Hadfield N, et al. «Genomic analysis of nCoV spread: Situation report 2020-01-30». nextstrain.org. Consultado em 18 de março de 2020
- ↑ Hodcroft E, Müller N, Wagner C, et al. «Genomic analysis of COVID-19 spread: Situation report 2020-03-13». nextstrain.org. Consultado em 18 de março de 2020
- ↑ Chen N, Zhou M, Dong X, et al. (15 de fevereiro de 2020). «Epidemiological and clinical characteristics of 99 cases of 2019 novel coronavirus pneumonia in Wuhan, China: a descriptive study». The Lancet. 395 (10223): 507–513. PMID 32007143. doi:10.1016/S0140-6736(20)30211-7
- ↑ a b c Wu C, Liu Y, Yang Y, et al. (fevereiro de 2020). «Analysis of therapeutic targets for SARS-CoV-2 and discovery of potential drugs by computational methods». Acta Pharmaceutica Sinica B. doi:10.1016/j.apsb.2020.02.008
- ↑ Xu X, Chen P, Wang J, et al. (março de 2020). «Evolution of the novel coronavirus from the ongoing Wuhan outbreak and modeling of its spike protein for risk of human transmission». Science China Life Sciences. 63 (3): 457–460. PMID 32009228. doi:10.1007/s11427-020-1637-5
- ↑ Letko M, Munster V (janeiro de 2020). «Functional assessment of cell entry and receptor usage for lineage B β-coronaviruses, including 2019-nCoV». bioRxiv (preprint). doi:10.1101/2020.01.22.915660
- ↑ Letko M, Marzi A, Munster V (fevereiro de 2020). «Functional assessment of cell entry and receptor usage for SARS-CoV-2 and other lineage B betacoronaviruses». Nature Microbiology. PMID 32094589. doi:10.1038/s41564-020-0688-y
- ↑ El Sahly HM. «Genomic Characterization of the 2019 Novel Coronavirus». The New England Journal of Medicine. Consultado em 9 de fevereiro de 2020
- ↑ Gralinski LE, Menachery VD (janeiro de 2020). «Return of the Coronavirus: 2019-nCoV». Viruses. 12 (2). 135 páginas. PMID 31991541. doi:10.3390/v12020135
- ↑ Lu R, Zhao X, Li J, Niu P, Yang B, Wu H, et al. (fevereiro de 2020). «Genomic characterisation and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding». The Lancet. 395 (10224): 565–574. PMID 32007145. doi:10.1016/S0140-6736(20)30251-8
- ↑ Wrapp D, Wang N, Corbett KS, Goldsmith JA, Hsieh CL, Abiona O, et al. (fevereiro de 2020). «Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation». Science. 367 (6483): 1260–1263. PMID 32075877. doi:10.1126/science.abb2507
- ↑ Wang K, Chen W, Zhou YS, et al. (2020). «SARS-CoV-2 invades host cells via a novel route: CD147-spike protein». bioRxiv (preprint). doi:10.1101/2020.03.14.988345
- ↑ Hoffman M, Kliene-Weber H, Krüger N, et al. (16 de abril de 2020). «SARS-CoV-2 Cell Entry Depends on ACE2 and TMPRSS2 and Is Blocked by a Clinically Proven Protease Inhibitor». Cell. 181: 1–10. PMID 32142651. doi:10.1016/j.cell.2020.02.052
- ↑ Oberholzer M, Febbo P (19 de fevereiro de 2020). «What We Know Today about Coronavirus SARS-CoV-2 and Where Do We Go from Here». Genetic Engineering and Biotechnology News. Consultado em 13 de março de 2020
- ↑ Ma J (13 de março de 2020). «Coronavirus: China's first confirmed Covid-19 case traced back to November 17». South China Morning Post. Consultado em 16 de março de 2020
- ↑ a b c d «Coronavirus Pandemic (COVID-19)». ourworldindata.org (em inglês). Our World In Data. 3 de março de 2024. Consultado em 15 de março de 2024
- ↑ Rothe C, Schunk M, Sothmann P, Bretzel G, Froeschl G, Wallrauch C, et al. (março de 2020). «Transmission of 2019-nCoV Infection from an Asymptomatic Contact in Germany». The New England Journal of Medicine. 382 (10): 970–971. PMID 32003551. doi:10.1056/NEJMc2001468
- ↑ Molteni M (30 de janeiro de 2020). «The Coronavirus Is Now Infecting More People Outside China». Wired. Consultado em 13 de março de 2020
- ↑ Khalik S (4 de fevereiro de 2020). «Coronavirus: Singapore reports first cases of local transmission; 4 out of 6 new cases did not travel to China». The Straits Times. Consultado em 5 de fevereiro de 2020. Cópia arquivada em 4 de fevereiro de 2020
- ↑ «Ecuador confirms five new cases of coronavirus, all close to initial patient». Reuters. 2 de março de 2020. Consultado em 5 de março de 2020
- ↑ «Algeria confirms two more coronavirus cases». Reuters. 2 de março de 2020. Consultado em 5 de março de 2020
- ↑ «Statement on the second meeting of the International Health Regulations (2005) Emergency Committee regarding the outbreak of novel coronavirus (2019-nCoV)». World Health Organization (WHO) (Nota de imprensa). 30 de janeiro de 2020. Consultado em 30 de janeiro de 2020. Cópia arquivada em 31 de janeiro de 2020
- ↑ McKay B, Calfas J, Ansari T (11 de março de 2020). «Coronavirus Declared Pandemic by World Health Organization». The Wall Street Journal. Consultado em 12 de março de 2020
- ↑ «WHO Director-General's opening remarks at the media briefing on COVID-19 - 11 March 2020». World Health Organization (WHO) (Nota de imprensa). 11 de março de 2020. Consultado em 12 de março de 2020
- ↑ Branswell H (30 de janeiro de 2020). «Limited data on coronavirus may be skewing assumptions about severity». STAT. Consultado em 13 de março de 2020. Cópia arquivada em 1 de fevereiro de 2020
- ↑ Wu JT, Leung K, Leung GM (fevereiro de 2020). «Nowcasting and forecasting the potential domestic and international spread of the 2019-nCoV outbreak originating in Wuhan, China: a modelling study». The Lancet. 395 (10225): 689–697. PMID 32014114. doi:10.1016/S0140-6736(20)30260-9
- ↑ Li Q, Guan X, Wu P, Wang X, Zhou L, Tong Y, et al. (janeiro de 2020). «Early Transmission Dynamics in Wuhan, China, of Novel Coronavirus-Infected Pneumonia». The New England Journal of Medicine. PMID 31995857. doi:10.1056/NEJMoa2001316
- ↑ Riou J, Althaus CL (janeiro de 2020). «Pattern of early human-to-human transmission of Wuhan 2019 novel coronavirus (2019-nCoV), December 2019 to January 2020». Euro Surveillance. 25 (4). PMC 7001239
 . PMID 32019669. doi:10.2807/1560-7917.ES.2020.25.4.2000058
. PMID 32019669. doi:10.2807/1560-7917.ES.2020.25.4.2000058
Bibliografia
- Brüssow H (março de 2020). «The Novel Coronavirus – A Snapshot of Current Knowledge». Microbial Biotechnology. 2020: 1–6. PMID 32144890. doi:10.1111/1751-7915.13557
- Habibzadeh P, Stoneman EK (fevereiro de 2020). «The Novel Coronavirus: A Bird's Eye View». The International Journal of Occupational and Environmental Medicine. 11 (2): 65–71. PMID 32020915. doi:10.15171/ijoem.2020.1921
- World Health Organization (2 de março de 2020). Laboratory testing for coronavirus disease 2019 (COVID-19) in suspected human cases: interim guidance, 2 March 2020 (Relatório). World Health Organization. hdl:10665/331329. WHO/COVID-19/laboratory/2020.4. License: CC BY-NC-SA 3.0
Ligações externas
- OMS sobre o novo coronavírus (em inglês. Consultado em 20 de janeiro de 2020.




