Transcrição (genética)

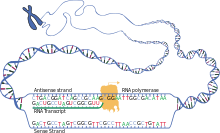
A transcrição é a primeira de várias etapas da expressão gênica baseada em DNA (gene é uma parte curta do DNA que sofre expressão), na qual um segmento específico de DNA é copiado no RNA (especialmente RNAm) pela enzima RNA polimerase. Tanto o DNA quanto o RNA são ácidos nucleicos, que usam pares de bases de nucleotídeos como uma linguagem complementar. Durante a transcrição, uma sequência de DNA é lida por uma RNA polimerase, que produz uma cadeia de RNA antiparalela complementar chamada transcrição primária. Exemplos:
- DNA ⇒…ATC GGC TAG CTA GCG TAG CGA TGC AAA TTT AAA TAT ATG…
- mRNA ⇒…UAG CCG AUC GAU CGC AUC GCU ACG UUU AAA UUU AUA UAC…
- Codões (português europeu) ou Codóns (português brasileiro) ⇒…[UAG][CCG][AUC][GAU][CGC][AUC][GCU][ACG][UUU][AAA][UUU][AUA][UAC]…
A transcrição continua nas seguintes etapas gerais:
- A RNA polimerase, juntamente com um ou mais fatores gerais de transcrição, se liga ao DNA do promotor.
- A RNA polimerase cria uma bolha de transcrição, que separa as duas cadeias da hélice do DNA. Isso é feito quebrando as ligações de hidrogênio entre os nucleotídeos de DNA complementares.
- A RNA polimerase adiciona nucleotídeos de RNA (que são complementares aos nucleotídeos de uma fita de DNA).
- O esqueleto açúcar-fosfato se forma com a assistência da RNA polimerase para formar uma fita de RNA.
- As ligações de hidrogênio da hélice RNA-DNA quebram, liberando a fita de RNA recém-sintetizada.
- Se a célula tiver um núcleo, o RNA poderá ser processado posteriormente. Isso pode incluir poliadenilação, nivelamento e união.
- O RNA pode permanecer no núcleo ou sair para o citoplasma através do complexo de poros nucleares.
O trecho de DNA transcrito em uma molécula de RNA é chamado de unidade de transcrição e codifica pelo menos um gene. Se o gene codifica é uma proteína, a transcrição produz RNA mensageiro (RNAm); o RNAm, por sua vez, serve como modelo para a síntese da proteína através da tradução. Como alternativa, o gene transcrito pode codificar para RNA não codificante, como microRNA, RNA ribossômico (RNAr), RNA transportador (RNAt) ou moléculas de RNA enzimático chamadas ribozimas.[1] No geral, o RNA ajuda a sintetizar, regular e processar proteínas; portanto, desempenha um papel fundamental na realização de funções dentro de uma célula.
Em virologia, o termo também pode ser usado quando se refere à síntese de RNAm a partir de uma molécula de RNA (isto é, replicação de RNA). Por exemplo, o genoma de um vírus de RNA de fita única de sentido negativo (ssRNA -) pode ser modelo para um RNA de fita única de sentido positivo (ssRNA +) [esclarecimento necessário]. Isso ocorre porque a cadeia de sentido positivo contém as informações necessárias para traduzir as proteínas virais para replicação viral posteriormente. Esse processo é catalisado por uma replicase viral de RNA.[2] [esclarecimentos necessários]
Por trás do processo[editar | editar código-fonte]
Uma unidade de transcrição de DNA que codifica para uma proteína pode conter uma sequência de codificação, que será traduzida na proteína, e sequências reguladoras, que direcionam e regulam a síntese dessa proteína. A sequência reguladora antes ("a montante") da sequência de codificação é chamada de região cinco não traduzida principal (5'UTR); a sequência após ("a jusante" de) a sequência de codificação é denominada região três não traduzida principal (3'UTR).[1]
Ao contrário da replicação do DNA, a transcrição resulta em um complemento de RNA que inclui o nucleotídeo uracila (U) em todos os casos em que a timina (T) teria ocorrido em um complemento de DNA.
Apenas uma das duas fitas de DNA serve como modelo para a transcrição. A cadeia anti-sentido do DNA é lida pela RNA polimerase da extremidade 3 'para a extremidade 5' durante a transcrição (3 '→ 5'). O RNA complementar é criado na direção oposta, na direção 5 '→ 3', combinando a sequência da cadeia de sentido, com exceção da troca de uracilo por timina. Essa direcionalidade é porque a RNA polimerase pode apenas adicionar nucleotídeos à extremidade 3 'da cadeia crescente de RNAm. Esse uso apenas da fita de DNA 3 '→ 5' elimina a necessidade dos fragmentos de Okazaki que são vistos na replicação do DNA.[1] Isso também elimina a necessidade de um iniciador de RNA para iniciar a síntese de RNA, como é o caso da replicação de DNA.
A fita não-modelo (sentido) do DNA é chamada de fita codificadora, porque sua sequência é a mesma que o transcrito de RNA recém-criado (exceto a substituição de timina por uracila). Essa é a cadeia usada por convenção ao apresentar uma sequência de DNA.[3]
A transcrição possui alguns mecanismos de revisão, mas são menos eficazes que os controles para replicação do DNA. Como resultado, a transcrição tem uma fidelidade menor que a replicação do DNA.[4]
Etapas principais[editar | editar código-fonte]
A transcrição é dividida em: iniciação, escape do promotor, alongamento, e terminação.[5]
Iniciação[editar | editar código-fonte]
A transcrição começa com a ligação da RNA polimerase, juntamente com um ou mais fatores gerais de transcrição, a uma sequência específica de DNA referida como "promotor" para formar um "complexo fechado" do RNA polimerase-promotor. No "complexo fechado", o DNA do promotor ainda é totalmente de fita dupla.[5]
A RNA polimerase, assistida por um ou mais fatores gerais de transcrição, desenrola aproximadamente 14 pares de bases de DNA para formar um "complexo aberto" promotor da RNA polimerase. No "complexo aberto", o DNA do promotor é parcialmente desenrolado e de fita simples. O DNA exposto, de fita simples, é chamado de "bolha de transcrição".[5]
A polimerase de RNA, assistida por um ou mais fatores gerais de transcrição, seleciona um local de início da transcrição na bolha de transcrição, liga-se a um NTP inicial e um NTP de extensão (ou um iniciador de RNA curto e um NTP de extensão) complementar à sequência do local de início da transcrição e catalisa a formação de ligações para produzir um produto de RNA inicial.[5]
Nas bactérias, a holoenzima RNA polimerase consiste em cinco subunidades: 2 subunidades α, subunidade 1 β, subunidade 1 β 'e subunidade 1 ω. Nas bactérias, existe um fator geral de transcrição de RNA conhecido como fator sigma. A enzima principal da RNA polimerase se liga ao fator de transcrição geral bacteriana (sigma) para formar a holoenzima da RNA polimerase e, em seguida, ao um promotor.[5] (A RNA polimerase é chamada holoenzima quando a subunidade sigma é anexada à enzima principal, que é composta por 2 subunidades α, 1 subunidade β, apenas subunidade 1 β ').
Nas arquéias e eucariotos, a RNA polimerase contém subunidades homólogas a cada uma das cinco subunidades da RNA polimerase em bactérias e também contém subunidades adicionais. Nas arquéias e eucariotos, as funções do fator sigma são desempenhadas por vários fatores de transcrição geral que trabalham juntos.[5] Na arquéia, existem três fatores gerais de transcrição: TBP, TFB e TFE. Nos eucariotos, na transcrição dependente da RNA polimerase II, existem seis fatores gerais de transcrição: TFIIA, TFIIB (um ortólogo do TFB archaeal), TFIID (um fator multissubunidade no qual a subunidade-chave, TBP, é um ortólogo do TBP archaeal), TFIIE (um ortólogo de TFE archaeal), TFIIF e TFIIH. O TFIID é o primeiro componente a se ligar ao DNA devido à ligação do TBP, enquanto o TFIIH é o último componente a ser recrutado. Nas arquéias e eucariotos, o complexo fechado de RNA polimerase-promotor é geralmente chamado de "complexo de pré-iniciação". "[6]
A iniciação da transcrição é regulada por proteínas adicionais, conhecidas como ativadores e repressores, e, em alguns casos, coativadores ou corepressores associados, que modulam a formação e a função do complexo de iniciação da transcrição.[5]
Escape do promotor[editar | editar código-fonte]
Após a primeira ligação ser sintetizada, a RNA polimerase deve escapar do promotor. Durante esse período, há uma tendência de liberar o transcrito de RNA e produzir transcritos truncados. Isso é chamado de iniciação abortiva e é comum para eucariotos e procariontes.[7] A iniciação abortiva continua a ocorrer até que um produto de RNA com um comprimento limite de aproximadamente 10 nucleotídeos seja sintetizado, momento em que ocorre a fuga do promotor e um complexo de alongamento da transcrição é formado.
Mecanisticamente, a fuga do promotor ocorre através do scrunching do DNA, fornecendo a energia necessária para interromper as interações entre a holoenzima da RNA polimerase e o promotor.[8]
Nas bactérias, acreditava-se historicamente que o fator sigma é definitivamente liberado após a liberação do promotor. Essa teoria era conhecida como modelo de liberação obrigatória, mas dados posteriores mostraram que, após a liberação do promotor, o fator sigma é liberado de acordo com um modelo estocástico conhecido como modelo de liberação estocástica.[9]
Em eucariotos, em um promotor dependente de RNA polimerase II, após a liberação do promotor, o TFIIH fosforila a serina 5 no domínio terminal carboxi da RNA polimerase II, levando ao recrutamento da enzima de capeamento (CE).[10][11] O mecanismo exato de como a CE induz a liberação do promotor em eucariotos ainda não é conhecido.
Alongamento[editar | editar código-fonte]

Uma fita do DNA, a fita modelo , é usada como modelo para a síntese de RNA. À medida que a transcrição prossegue, a RNA polimerase usa a complementaridadede bases com o modelo de DNA para criar uma cópia de RNA (que se prolonga durante a travessia). Embora a RNA polimerase atravesse a cadeia modelo de 3 '→ 5', a cadeia de codificação (não modelo) e o RNA recém-formado também podem ser usados como pontos de referência, de modo que a transcrição pode ser descrita como ocorrendo 5 '→ 3'. Isso produz uma molécula de RNA a partir de 5 '→ 3', uma cópia exata da cadeia de codificação (exceto que as timinas são substituídas por uracilos, e os nucleotídeos são compostos de um açúcar ribose (5 carbonos) em que o DNA possui desoxirribose (menos oxigênio) átomo) no seu esqueleto açúcar-fosfato). [carece de fontes]
A transcrição de RNAm pode envolver múltiplas RNA polimerases em um único modelo de DNA e várias rodadas de transcrição (amplificação de RNAm específico), de modo que muitas moléculas de RNAm podem ser produzidas rapidamente a partir de uma única cópia de um gene. [carece de fontes]E eucariotos são cerca de 10 a 100 nts / s.[12] Em eucariotos, no entanto, os nucleossomos atuam como principais barreiras à transcrição de polimerases durante o alongamento da transcrição.[13][14] Nesses organismos, a pausa induzida por nucleossomos pode ser regulada por fatores de alongamento da transcrição, como o TFIIS.[14]
O alongamento também envolve um mecanismo de revisão que pode substituir bases incorretamente incorporadas. Nos eucariotos, isso pode corresponder a pequenas pausas durante a transcrição que permitem a ligação de fatores de edição de RNA apropriados. Essas pausas podem ser intrínsecas à RNA polimerase ou devido à estrutura da cromatina.
Terminação[editar | editar código-fonte]
As bactérias usam duas estratégias diferentes para a terminação da transcrição - terminação independente de Rho e terminação dependente de Rho. Na terminação da transcrição independente de Rho, a transcrição de RNA para quando a molécula de RNA recém-sintetizada forma um laço em gancho de cabelo rico em G-C, seguido por uma sequência de Us. Quando o gancho de cabelo se forma, o estresse mecânico quebra as fracas ligações rU-dA, preenchendo agora o híbrido DNA-RNA. Isso puxa o transcrito poli-U para fora do local ativo da RNA polimerase, terminando a transcrição. No tipo de terminação "dependente de Rho", um fator de proteína chamado "Rho" desestabiliza a interação entre o modelo e o mRNA, liberando assim o mRNA recém-sintetizado do complexo de alongamento.[15]
A terminação da transcrição em eucariotos é menos compreendida do que em bactérias, mas envolve a clivagem da nova transcrição, seguida pela adição independente de modelo de adeninas em sua nova extremidade 3 ', em um processo chamado poliadenilação.[16]
Inibidores[editar | editar código-fonte]
Os inibidores da transcrição podem ser usados como antibióticos contra, por exemplo, bactérias patogênicas (antibacterianas) e fungos (antifúngicos). Um exemplo desse antibacteriano é a rifampicina, que inibe a transcrição bacteriana de DNA no RNAm, inibindo a RNA polimerase dependente de DNA, ligando sua subunidade beta, enquanto a 8-hidroxiquinolina é um inibidor da transcrição antifúngica.[17] Os efeitos da metilação da histona também podem funcionar para inibir a ação da transcrição.
Inibidores endógenos[editar | editar código-fonte]
Nos vertebrados, a maioria dos promotores de genes contém uma ilha CpG com vários locais CpG. Quando muitos dos sítios CpG do promotor de um gene são metilados, o gene fica inibido (silenciado).[18] When many of a gene's promoter CpG sites are methylated the gene becomes inhibited (silenced).[19] O câncer colorretal geralmente tem de 3 a 6 mutações no driver e 33 a 66 mutações em caronas ou passageiros.[20] No entanto, a inibição da transcrição (silenciamento) pode ser mais importante do que a mutação na progressão para o câncer. Por exemplo, nos cânceres colorretais, cerca de 600 a 800 genes são inibidos transcricionalmente pela metilação da ilha CpG (consulte a regulamentação da transcrição no câncer). A repressão transcricional no câncer também pode ocorrer por outros mecanismos epigenéticos, como expressão alterada de microRNAs.[21] No câncer de mama, a repressão transcricional do BRCA1 pode ocorrer com maior frequência por microRNA-182 superexpresso do que por hipermetilação do promotor BRCA1 (consulte Baixa expressão de BRCA1 em câncer de mama e ovário).
Fatores de Transcrição[editar | editar código-fonte]
As unidades de transcrição ativa estão agrupadas no núcleo, em locais discretos chamados fábricas de transcrição ou eucromatina. Tais locais podem ser visualizados permitindo que as polimerases envolvidas estendam seus transcritos em precursores marcados (Br-UTP ou Br-U) e imunomarcando o RNA nascente marcado. As fábricas de transcrição também podem ser localizadas usando hibridização fluorescente in situ ou marcadas por anticorpos direcionados contra polimerases. Existem ~ 10.000 fábricas no nucleoplasma de uma célula HeLa, entre as quais ~ 8.000 fábricas de polimerase II e ~ 2.000 fábricas de polimerase III. Cada fábrica de polimerase II contém ~ 8 polimerases. Como a maioria das unidades de transcrição ativas está associada a apenas uma polimerase, cada fábrica geralmente contém ~ 8 unidades de transcrição diferentes. Essas unidades podem ser associadas através de promotores e / ou potenciadores, com loops formando uma "nuvem" em torno do fator.[22]
História[editar | editar código-fonte]
Uma molécula que permite que o material genético seja realizado como uma proteína foi a primeira hipótese de François Jacob e Jacques Monod. Severo Ochoa ganhou o Prêmio Nobel de Fisiologia ou Medicina em 1959 por desenvolver um processo para sintetizar RNA in vitro com polinucleotídeo fosforilase, que foi útil para decifrar o código genético. A síntese de RNA pela RNA polimerase foi estabelecida in vitro por vários laboratórios em 1965; no entanto, o RNA sintetizado por essas enzimas tinha propriedades que sugeriam a existência de um fator adicional necessário para finalizar a transcrição corretamente.
Em 1972, Walter Fiers se tornou a primeira pessoa a realmente provar a existência da enzima terminadora.
Roger D. Kornberg ganhou o Prêmio Nobel de Química de 2006 "por seus estudos sobre as bases moleculares da transcrição eucariótica".[23]
Medição e detecção[editar | editar código-fonte]
A transcrição pode ser medida e detectada de várias maneiras:
- Ensaio de transcrição G-Less Cassette: mede a força do promotor
- Ensaio de transcrição de escoamento superficial: identifica sites de início de transcrição (TSS)
- Ensaio de execução nuclear: mede a abundância relativa de transcritos recém-formados
- Ensaio de proteção da RNase e ChIP-Chip do RNAP: detectar locais de transcrição ativos
- RT-PCR: mede a abundância absoluta dos níveis de RNA total ou nuclear, que podem, contudo, diferir das taxas de transcrição
- Microarrays de DNA: mede a abundância relativa dos níveis globais de RNA total ou nuclear; no entanto, estes podem diferir das taxas de transcrição
- Hibridização in situ: detecta a presença de uma transcrição
- Marcação MS2: incorporando loops-tronco de RNA, como o MS2, em um gene, eles se incorporam ao RNA recém-sintetizado. As alças do caule podem ser detectadas usando uma fusão de GFP e a proteína de revestimento MS2, que possui uma alta afinidade, interação específica da sequência com as alças do MS2. O recrutamento de GFP para o local da transcrição é visualizado como um único ponto fluorescente. Essa nova abordagem revelou que a transcrição ocorre em rajadas ou pulsos descontínuos (consulte Explosão transcricional). Com a exceção notável das técnicas in situ, a maioria dos outros métodos fornece médias da população celular e não são capazes de detectar essa propriedade fundamental dos genes.[24]
- Northern blot: o método tradicional, e até o advento do RNA-Seq, o método mais quantitativo
- RNA-Seq: aplica técnicas de sequenciamento de última geração para sequenciar transcriptomas inteiros, o que permite a medição da abundância relativa de RNA, bem como a detecção de variações adicionais, como genes de fusão, edições pós-transcricionais e novos locais de emenda
- RNA-Seq de célula única: amplifica e lê transcriptomas parciais de células isoladas, permitindo análises detalhadas de RNA em tecidos, embriões e cânceres
Transcrição Reversa[editar | editar código-fonte]
Alguns vírus (como o HIV, a causa da AIDS), têm a capacidade de transcrever RNA para DNA. O HIV tem um genoma de RNA que é transcrito reversamente em DNA. O DNA resultante pode ser fundido com o genoma do DNA da célula hospedeira. A principal enzima responsável pela síntese de DNA a partir de um modelo de RNA é denominada transcriptase reversa.
No caso do HIV, a transcriptase reversa é responsável pela síntese de uma fita de DNA complementar (cDNA) no genoma do RNA viral. A enzima ribonuclease H digere a fita de RNA e a transcriptase reversa sintetiza uma fita complementar de DNA para formar uma estrutura de dupla hélice de DNA ("cDNA"). O cDNA é integrado ao genoma da célula hospedeira pela enzima integrase, que faz com que a célula hospedeira gere proteínas virais que se reorganizem em novas partículas virais. No HIV, subsequentemente, a célula hospedeira sofre morte celular programada ou apoptose de células T.[25] No entanto, em outros retrovírus, a célula hospedeira permanece intacta à medida que o vírus sai da célula.
Algumas células eucarióticas contêm uma enzima com atividade de transcrição reversa chamada telomerase. A telomerase é uma transcriptase reversa que alonga as extremidades dos cromossomos lineares. A telomerase carrega um modelo de RNA a partir do qual sintetiza uma sequência repetida de DNA, ou DNA "lixo". Essa sequência repetida de DNA é chamada telômero e pode ser vista como uma "tampa" para um cromossomo. É importante porque toda vez que um cromossomo linear é duplicado, ele é reduzido. Com esse DNA "lixo" ou "tampa" nas extremidades dos cromossomos, o encurtamento elimina parte da sequência repetida não essencial e não a sequência de DNA que codifica a proteína, que fica mais distante da extremidade do cromossomo.
A telomerase é freqüentemente ativada nas células cancerígenas para permitir que as células duplicem seus genomas indefinidamente, sem perder uma importante sequência de DNA codificadora de proteínas. A ativação da telomerase pode ser parte do processo que permite que as células cancerígenas se tornem imortais. Foi comprovado que o fator imortalizante do câncer via alongamento de telômeros devido à telomerase ocorre em 90% de todos os tumores carcinogênicos in vivo, com os 10% restantes, usando uma via alternativa de manutenção de telômeros chamada ALT ou alongamento alternativo de telômeros.[26]
Ver também[editar | editar código-fonte]
Referências
- ↑ a b c Eldra P. Solomon, Linda R. Berg, Diana W. Martin. Biology, 8th Edition, International Student Edition. Thomson Brooks/Cole. ISBN 978-0495317142
- ↑ Koonin EV, Gorbalenya AE, Chumakov KM (julho de 1989). «Tentative identification of RNA-dependent RNA polymerases of dsRNA viruses and their relationship to positive strand RNA viral polymerases». FEBS Letters. 252 (1-2): 42–6. PMID 2759231. doi:10.1016/0014-5793(89)80886-5
- ↑ «DNA Strands». www.sci.sdsu.edu. Consultado em 1 de maio de 2018. Cópia arquivada em 27 de outubro de 2017
- ↑ Berg J, Tymoczko JL, Stryer L (2006). Biochemistry 6th ed. San Francisco: W. H. Freeman. ISBN 0-7167-8724-5
- ↑ a b c d e f g Watson JD, Baker TA, Bell SP, Gann AA, Levine M, Losick RM (2013). Molecular Biology of the Gene 7th ed. [S.l.]: Pearson
- ↑ Roeder, Robert G. (1991). «The complexities of eukaryotic transcription initiation: regulation of preinitiation complex assembly». Trends in Biochemical Sciences. 16: 402–408. ISSN 0968-0004. doi:10.1016/0968-0004(91)90164-Q
- ↑ Goldman SR, Ebright RH, Nickels BE (maio de 2009). «Direct detection of abortive RNA transcripts in vivo». Science. 324 (5929): 927–8. PMC 2718712
 . PMID 19443781. doi:10.1126/science.1169237
. PMID 19443781. doi:10.1126/science.1169237
- ↑ Revyakin A, Liu C, Ebright RH, Strick TR (novembro de 2006). «Abortive initiation and productive initiation by RNA polymerase involve DNA scrunching». Science. 314 (5802): 1139–43. PMC 2754787
 . PMID 17110577. doi:10.1126/science.1131398
. PMID 17110577. doi:10.1126/science.1131398
- ↑ Raffaelle M, Kanin EI, Vogt J, Burgess RR, Ansari AZ (novembro de 2005). «Holoenzyme switching and stochastic release of sigma factors from RNA polymerase in vivo». Molecular Cell. 20 (3): 357–66. PMID 16285918. doi:10.1016/j.molcel.2005.10.011
- ↑ Mandal SS, Chu C, Wada T, Handa H, Shatkin AJ, Reinberg D (maio de 2004). «Functional interactions of RNA-capping enzyme with factors that positively and negatively regulate promoter escape by RNA polymerase II». Proceedings of the National Academy of Sciences of the United States of America. 101 (20): 7572–7. PMC 419647
 . PMID 15136722. doi:10.1073/pnas.0401493101
. PMID 15136722. doi:10.1073/pnas.0401493101
- ↑ Goodrich JA, Tjian R (abril de 1994). «Transcription factors IIE and IIH and ATP hydrolysis direct promoter clearance by RNA polymerase II». Cell. 77 (1): 145–56. PMID 8156590. doi:10.1016/0092-8674(94)90242-9
- ↑ Milo, Ron; Philips, Rob. «Cell Biology by the Numbers: What is faster, transcription or translation?». book.bionumbers.org. Consultado em 8 de março de 2017. Cópia arquivada em 20 de abril de 2017
- ↑ Hodges C, Bintu L, Lubkowska L, Kashlev M, Bustamante C (julho de 2009). «Nucleosomal fluctuations govern the transcription dynamics of RNA polymerase II». Science. 325 (5940): 626–8. PMC 2775800
 . PMID 19644123. doi:10.1126/science.1172926
. PMID 19644123. doi:10.1126/science.1172926
- ↑ a b Fitz V, Shin J, Ehrlich C, Farnung L, Cramer P, Zaburdaev V, Grill SW (2016). «Nucleosomal arrangement affects single-molecule transcription dynamics». Proceedings of the National Academy of Sciences. 113 (45): 12733–12738. PMC 5111697
 . doi:10.1073/pnas.1602764113. Cópia arquivada em 1 de maio de 2018
. doi:10.1073/pnas.1602764113. Cópia arquivada em 1 de maio de 2018
- ↑ Richardson JP (setembro de 2002). «Rho-dependent termination and ATPases in transcript termination». Biochimica et Biophysica Acta. 1577 (2): 251–260. PMID 12213656. doi:10.1016/S0167-4781(02)00456-6
- ↑ Lykke-Andersen S, Jensen TH (outubro de 2007). «Overlapping pathways dictate termination of RNA polymerase II transcription». Biochimie. 89 (10): 1177–82. PMID 17629387. doi:10.1016/j.biochi.2007.05.007
- ↑ 8-Hydroxyquinoline info from SIGMA-ALDRICH. Retrieved Feb 2012
- ↑ Saxonov S, Berg P, Brutlag DL (janeiro de 2006). «A genome-wide analysis of CpG dinucleotides in the human genome distinguishes two distinct classes of promoters». Proceedings of the National Academy of Sciences of the United States of America. 103 (5): 1412–7. PMC 1345710
 . PMID 16432200. doi:10.1073/pnas.0510310103
. PMID 16432200. doi:10.1073/pnas.0510310103
- ↑ Bird A (janeiro de 2002). «DNA methylation patterns and epigenetic memory». Genes & Development. 16 (1): 6–21. PMID 11782440. doi:10.1101/gad.947102
- ↑ Vogelstein B, Papadopoulos N, Velculescu VE, Zhou S, Diaz LA, Kinzler KW (março de 2013). «Cancer genome landscapes». Science. 339 (6127): 1546–58. PMC 3749880
 . PMID 23539594. doi:10.1126/science.1235122
. PMID 23539594. doi:10.1126/science.1235122
- ↑ Tessitore A, Cicciarelli G, Del Vecchio F, Gaggiano A, Verzella D, Fischietti M, Vecchiotti D, Capece D, Zazzeroni F, Alesse E (2014). «MicroRNAs in the DNA Damage/Repair Network and Cancer». International Journal of Genomics. 2014. 820248 páginas. PMC 3926391
 . PMID 24616890. doi:10.1155/2014/820248
. PMID 24616890. doi:10.1155/2014/820248
- ↑ Papantonis A, Kohro T, Baboo S, Larkin JD, Deng B, Short P, Tsutsumi S, Taylor S, Kanki Y, Kobayashi M, Li G, Poh HM, Ruan X, Aburatani H, Ruan Y, Kodama T, Wada Y, Cook PR (novembro de 2012). «TNFα signals through specialized factories where responsive coding and miRNA genes are transcribed». The EMBO Journal. 31 (23): 4404–14. CiteSeerX 10.1.1.919.1919
 . PMC 3512387
. PMC 3512387 . PMID 23103767. doi:10.1038/emboj.2012.288
. PMID 23103767. doi:10.1038/emboj.2012.288
- ↑ «Chemistry 2006». Nobel Foundation. Consultado em 29 de março de 2007. Cópia arquivada em 15 de março de 2007
- ↑ Raj A, van Oudenaarden A (outubro de 2008). «Nature, nurture, or chance: stochastic gene expression and its consequences». Cell. 135 (2): 216–26. PMC 3118044
 . PMID 18957198. doi:10.1016/j.cell.2008.09.050
. PMID 18957198. doi:10.1016/j.cell.2008.09.050
- ↑ Kolesnikova IN (2000). «Some patterns of apoptosis mechanism during HIV-infection». Dissertation (em russo). Consultado em 20 de fevereiro de 2011. Cópia arquivada em 10 de julho de 2011
- ↑ Cesare AJ, Reddel RR (maio de 2010). «Alternative lengthening of telomeres: models, mechanisms and implications». Nature Reviews. Genetics. 11 (5): 319–30. PMID 20351727. doi:10.1038/nrg2763
