SnoRNA: diferenças entre revisões
genes de RNAs, snoRNAs modificam RNAs e nao suas regioes codificantes. |
traduzi do inglês |
||
| Linha 1: | Linha 1: | ||
[[Ficheiro:RF00071.jpg|thumb|right|200px|[[SNORD73]] (RF00071).]] |
[[Ficheiro:RF00071.jpg|thumb|right|200px|[[SNORD73]] (RF00071).]] |
||
{{Não confundir com|snRNA}} |
|||
'''Small nucleolar RNAs''' ('''snoRNAs''') são uma classe de pequenas moléculas de [[RNA]] que conduzem modificações químicas (metilação ou pseudouridilação) em RNAs ribossomais ([[rRNA]]s) e outros RNAs ([[tRNA]]s e outros pequenos RNAs nucleares). |
|||
Os '''pequenos RNAs nucleolares''' (snoRNAs) são uma classe de pequenas moléculas de [[RNA não-codificante]] (ncRNAs) que conduzem modificações químicas ([[metilação]] ou pseudouridilação) em outros RNAs, principalmente [[RNA ribossômico|RNAs ribossômicos]], [[RNA transportador|RNAs transportadores]] e [[snRNA|pequenos RNAs nucleares]]. Estas moléculas formam pares de bases com regiões complementares da molécula de pré-RNA, direcionando a clivagem da cadeia de RNA e a modificação de bases durante a maturação das moléculas de RNAr.<ref name=":1">{{Citar livro|título=Biologia Celular e Molecular|ultimo=Lodish|primeiro=Harvey|editora=Artmed Editora|ano=2014|páginas=388, 390, 393, 394, 1178|ultimo2=Berk|primeiro2=Arnold|ultimo3=Kaiser|primeiro3=Chris A.|ultimo4=Krieger|primeiro4=Monty|ultimo5=Bretscher|primeiro5=Anthony|ultimo6=Ploegh|primeiro6=Hidde|ultimo7=Amon|primeiro7=Angelika|ultimo8=Scott|primeiro8=Matthew P.|data=|edicao=7|lingua=pt-br|isbn=9781429234139}}</ref> |
|||
O processamento do pré-RNA é realizado com a ajuda de um grande número de pequenos RNAs nucleolares (snoRNAs) que são agrupados com [[proteína]]s particulares para formar partículas chamadas pequenas ribonucleoproteínas nucleolares (snoRNPs). Os snoRNPs associam-se ao precursor do rRNA antes que ele seja completamente [[transcrição|transcrito]], realizando cortes enzimáticos em zonas específicas do transcrito. Todas estas alterações ocorrem após a incorporação dos nucleotídeos ao RNA nascente, isto é, pós-transcripcional. |
|||
Existem duas classes principais de '''snoRNAs''', '''caixa C/D''' (associados à metilação) e '''caixa H/ACA''' (associados à pseudouridilação), os quais servem como RNAs guias complementares a sequências alvo específicas, principalmente em precursores de RNAr.<ref name=":2">{{Citar periódico|titulo=Identification of human miRNA precursors that resemble C/D box snoRNAs|ultimo=Ono|primeiro=M|ultimo2=Scott|primeiro2=MS|ultimo3=Yamada|primeiro3=K|ultimo4=Avolio|primeiro4=F|ultimo5=Barton|primeiro5=GJ|ultimo6=Lamond|primeiro=AI|publicado=Nucleic Acids Research|volume=39|edicao=9|data=1 maio 2011|pages=3879–3891|DOI=10.1093/nar/gkq1355|PMC=3089480|PMID=21247878}}</ref> Muitos snoRNAs são codificados nos íntrons de outros genes, especialmente aqueles que codificam proteínas ribossômicas.<ref name=":3" /> |
|||
Apesar da origem dos snoRNA ainda não ser algo certo, pensa-se que estes provêem dos intrões do mRNA. Muitas vezes, os [[intrão|intrões]] que não seriam transcritos para sintetizar proteínas, seriam transformados em snoRNAs, ficando com função catalítica. Esta teoria explica, assim, a importância, até agora, desconhecida dos intrões. |
|||
Apesar da origem dos '''snoRNA''' ainda ser incerta, pensa-se que estes provêem dos intrões do mRNA. Muitas vezes, os [[intrão|intrões]] que não seriam transcritos para sintetizar proteínas, seriam transformados em snoRNAs, ficando com função catalítica. Esta teoria explica, assim, a importância, até agora, desconhecida dos intrões.{{Carece de fontes}} Vários snoRNAs foram descobertos anos atrás porque estavam presentes em quantidades relativamente grandes. Mais recentemente foi encontrada uma classe diferente de snoRNAs, em menor concentração, cujas cadeias são relativamente mais longas e que são complementares a secções do transcrito [[RNA ribossômico|rRNA]] - são chamados, por isso snoRNAs antiparalelos.{{Carece de fontes}} Cada snoRNA antiparalelo liga-se a um porção específica do pré-rRNA para formar uma estrutura dupla, que serve como local de reconhecimento para enzimas que modificam um nucleotídeo em particular na sequência do pré-rRNA. Se o gene codificador de um desses snoRNAs é perdido, um dos nucleotídeos do pré-rRNA não sofre o processo de modificação enzimática.{{Carece de fontes}} |
|||
O tamanho da cadeia dos snoRNAs é variado, entre 60 a 300 nucleotídos. A variedade de tamanhos das cadeias pressupõe, pela parte dos snoRNAs, assumir variadas funções. |
|||
==Modificações guiadas por snoRNA== |
|||
Vários snoRNAs foram descobertos anos atrás porque estavam presentes em quantidades relativamente grandes. Mais recentemente foi encontrada uma classe diferente de snoRNAs, em menor concentração, cujas cadeias são relativamente mais longas e que são complementares a secções do transcrito [[RNA ribossômico|rRNA]] - são chamados, por isso snoRNAs antiparalelos. |
|||
Antes da clivagem do RNAr, ocorrem várias modificações químicas no precursor do RNAr (pré-RNAr), guiadas pelos snoRNAs. Estas incluem cerca de 100 metilações das posições 2’-OH nos açúcares dos nucleotídeos, e 100 [[isomerizações]] de nucleotídeos [[uridina]] para pseudouridina.<ref name=":3">{{citar livro|titulo=Biologia Molecular da Célula|autor=Alberts|primeiro=Bruce|ultimo2=Johnson|primeiro2=Alexander|ultimo3=Lewis|primeiro3=Julian|editora=Artmed|ano=2017|local=|página=|páginas=|ISBN=978-85-8271-423-2}}</ref> |
|||
Cada snoRNA antiparalelo liga-se a um porção específica do pré-rRNA para formar uma estrutura dupla, que serve como local de reconhecimento para enzimas que modificam um nucleotídeo em particular na sequência do pré-rRNA. Se o gene codificador de um desses snoRNAs é perdido, um dos nucleotídeos do pré-rRNA não sofre o processo de modificação enzimática. |
|||
Os snoRNAs são agrupados com [[proteínas]] particulares para formar '''pequenas ribonucleoproteínas nucleolares''' (snoRNPs), formando um '''complexo proteína/snoRNA'''.<ref name=":1"/> As proteínas associadas a cada RNA dependem do tipo de molécula de snoRNA. Os snoRNPs associam-se ao precursor do RNAr antes que ele seja completamente [[transcrição|transcrito]], realizando cortes enzimáticos em zonas específicas do transcrito. Aproximadamente 150 snoRNAs, associado a proteínas nos snoRNPs, formam pares de bases com sítios específicos do pré-rRNA, onde direcionam a metilação da [[ribose]], as modificações da [[uridina]] em pseudouridina, e a [[clivagem]] em sítios específicos durante o processamento do [[RNAr]] no [[nucléolo]].<ref name=":1" /> |
|||
Recentes estudos revelam que os snoRNAs, ao serem combinadas com proteínas para formar snoRNPs, não possuem sequências suficientes na sua cadeia para que sejam complementares da cadeia de rRNA, que portanto não será cortada. |
|||
A molécula snoRNA contém um elemento ''antisense'' (ou contra-sentido, um trecho de 10-20 [[nucleotídeos]]), que são bases complementares à sequência que envolve a base ([[nucleotídeo]]) direcionada para modificação no pré-RNA molécula. Isso permite que o snoRNP reconheça e se ligue ao RNA alvo. Uma vez que o snoRNP tenha se ligado ao local alvo, as proteínas associadas estão no local físico correto para [[catalisar]] a modificação química da base alvo.<ref name=":4">{{citar periódico |ultimo1=Lykke-Andersen |primeiro1=Søren |ultimo2=Ardal |primeiro2=Britt Kidmose |ultimo3=Hollensen |primeiro3=Anne Kruse |ultimo4=Damgaard |primeiro4=Christian Kroun |ultimo5=Jensen |primeiro5=Torben Heick |titulo=C/D Box snoRNP Autoregulation by a cis-Acting snoRNA in the NOP56 Pre-mRNA |publicado=Molecular Cell |data=outubro 2018 |volume=72 |edição=1 |páginas=99–111.e5 |doi=10.1016/j.molcel.2018.08.017 |pmid=30220559}}</ref> |
|||
Hipoteticamente, os snoRNAs ao juntarem-se a outras proteínas fora do complexo ribossómico, irão suportar outras funções bem distintas. A mais recente descoberta constatou que os snoRNAs não têm só como funções a modificação do rRNA e dos spliceossomal RNAs. Têm também como função a regulação do [[splicing alternativo]] em transcrições em forma de trans. |
|||
== Classes de snoRNA == |
|||
A maioria dos snoRNPs se enquadram em duas grandes classes, de acordo com os motivos (elementos de sequência-chave) conservados pelo snoRNA. Essas classes são denominadas por caixas ou motivos;<ref>{{citar livro|título=Princípios de Bioquímica de Lehninger|ultimo=Nelson|primeiro=David L.|ultimo2=Cox|primeiro2=Michael M.|editora=Artmed Editora|ano=2018|edicao=7|local=|página=1057|isbn=9788582715345}}</ref> são os snoRNA de caixa C/D e caixa H/ACA. Existem exceções, mas como regra geral, os membros da caixa C/D guiam a metilação enquanto os membros da caixa H/ACA guiam a pseudouridilação. Os membros de cada família podem variar em biogênese, estrutura e função, mas cada família é classificada pelas seguintes características generalizadas:<ref name=":5">{{Citar periódico |autores=Bachellerie JP, Cavaillé J, Hüttenhofer A | titulo=The expanding snoRNA world |publicado=Biochimie | volume=84 | edição=8 | páginas=775–90 | data=Agosto 2002 | pmid=12457565 | pmc= | doi=10.1016/S0300-9084(02)01402-5}}</ref> |
|||
===Caixa C/D=== |
|||
Os snoRNAs caixa C/D são caracterizados por dois domínios de sequências conservadas, a caixa C (RUGAUGA) e a caixa D (CUGA),<ref name=":6">{{Citar periódico |autores=Lestrade L, Weber MJ|titulo=snoRNA-LBME-db, a comprehensive database of human H/ACA and C/D box snoRNAs|publicado=Nucleic Acids Research|volume=34|data=1 jan 2006|paginas=158–162|doi=10.1093/nar/gkj002|PMID=16381836|PMC=1347365}}</ref> localizados perto das extremidades 5' e 3' do snoRNA, respectivamente. Regiões curtas (~5 nucleotídeos) localizadas a montante da caixa C e a jusante da caixa D são geralmente complementares nas bases e formam uma estrutura síntese adequada de snoRNAs e para sua localização nucleolar. |
|||
Muitos snoRNAs C/D também contêm uma cópia adicional menos conservada dos motivos C e D (chamados C' e D'), localizada na porção central da molécula. Eles possuem elementos ''antisense'' de 10-21 nt de comprimento, localizados a montante das caixas D e/ou D', de modo que o nucleotídeo do RNA alvo emparelhado com o quinto nucleotídeo a montante da caixa D/D' é 2'-O-ribose metilado.<ref name=":6" /> Os snoRNAs caixa C/D e seus substratos de RNA formam uma dupla-hélice de 10-21 pb na qual o resíduo-alvo a ser metilado é posicionado, exatamente 5 nucleotídeos antes do motivo D ou D'.<ref name=":7">{{Citar livro|título=Genética Molecular Humana|ultimo=Strachan|primeiro=Tom|ultimo2=Read|primeiro2=Andrew|ano=2016|pagina=282}}</ref> Estão associados a quatro proteínas essenciais preservadas na evolução, que estão em eucariotos: fibrilarina (Nop1p), Nop56p, Nop58p e Snu13, que formam o núcleo de snoRNPs de caixa C/D.<ref name=":5" /> |
|||
Há também um snoRNA caixa C/D eucariótico (snoRNA U3) que não demonstrou direcionar a [[2'-O-metilação|metilação 2'-O]]. Em vez disso, funciona direcionando a clivagem do pré-rRNA. |
|||
===Caixa H/ACA=== |
|||
[[Imagem:RF00265.jpg|miniatura|direita|200px|Exemplo de estrutura secundária de um snoRNA de caixa H/ACA, retirado da base de dados Rfam. Este exemplo é o snoRNA SNORA69 (RF00265).]] |
|||
Os snoRNAs caixa H/ACA são guias para a isomerização de resíduos de uridina em pseudouridina. Eles dobram-se em uma estrutura de grampo-dobradiça-grampo-cauda,<ref name=":7" /> As sequências de snoRNA caixa H/ACA que interagem com o RNA alvo estão situadas em fitas opostas de alças internas (chamadas bolsas de pseudouridilação) dentro de uma ou ambas as estruturas de haste-alça conservadas. |
|||
Os snoRNAs caixa H/ACA possuem a caixa H (ANANNA) e a caixa ACA (ACA). Ambas estão geralmente localizados em uma única cadeia da estrutura secundária. O motivo H está localizado na dobradiça e o motivo ACA na região da cauda, a 3 nucleotídeos do extremo 3' da sequência.<ref>{{Citar periódico|autores=Ganot P, Caizergues-Ferrer M, Kiss T |titulo=The family of box ACA small nucleolar RNAs is defined by an evolutionarily conserved secondary structure and ubiquitous sequence elements essential for RNA accumulation |publicado = Genes Dev. | volume=11|edição=7|paginas=941–56|ano=1997|pmid=9106664|doi=10.1101/gad.11.7.941}}</ref> |
|||
As regiões grampo possuem protuberâncias internas conhecidas como loops de reconhecimento, em que as sequências-guia ''antisense'' estão localizadas. O snoRNA caixa H/ACA se associa com quatro proteínas essenciais conservadas evolutivamente, que sao: disquerina (Cbf5p), Gar1p, Nhp2p e Nop10p, que formam o núcleo das snoRNP caixa H/ACA.<ref name=":5" /> Porém, em organismos como o [[tripanosoma]], existem RNA similares na forma de grampo simples e uma caixa AGA no lugar da caixa ACA.<ref>{{Citar periódico | autores = Liang XH, Liu L, Michaeli S | titulo = Identification of the first trypanosome H/ACA RNA that guides pseudouridine formation on rRNA. | publicado = JBC. | volume =276 | issue = 43 | pages = 40313–8 | year = 2001 | pmid = 11483606 | doi = 10.1074/jbc.M104488200}}</ref> |
|||
===SnoRNAs "orfãos"=== |
|||
Os alvos de snoRNA recentemente identificados podem ser previstos com base na complementaridade da sequência entre o suposto RNA alvo e os elementos antisense ou loops de reconhecimento em sua sequência. Todavia, muitos snoRNAs "órfãos", que não possuem complementaridade com rRNAs ou snRNAs,<ref name=":9">Jády, B. K. (Mar 2000). "Characterisation of the U83 and U84 small nucleolar RNAs: two novel 2′-O-ribose methylation guide RNAs that lack complementarities to ribosomal RNAs" (Free full text). Nucleic Acids Research 28 (6): 1348–1354. doi:10.1093/nar/28.6.1348. ISSN 0305-1048. PMC 111033. PMID 10684929. http://nar.oxfordjournals.org/cgi/pmidlookup?view=long&pmid=10684929.</ref> vem sendo identificados. |
|||
Isso sugere que pode haver mais proteínas ou transcritos envolvidos no processamento de RNAr do que se pensava anteriormente e/ou que alguns snoRNAs têm outras funções que não envolvem o RNAr.<ref>Li, G. .; Zhou, H. .; Luo, P. .; Zhang, P. .; Qu, H. . (Apr 2005). "Identification and functional analysis of 20 Box H/ACA small nucleolar RNAs (snoRNAs) from Schizosaccharomyces pombe" (Free full text). The Journal of Biological Chemistry 280 (16): 16446–16455. doi:10.1074/jbc.M500326200. ISSN 0021-9258. PMID 15716270. http://www.jbc.org/cgi/pmidlookup?view=long&pmid=15716270.</ref> Há evidências de que alguns desses snoRNAs órfãos regulam transcrições de [[splicing alternativo]].<ref name=":11">Kishore, S. .; Stamm, S. . (2006). "Regulation of alternative splicing by snoRNAs". Cold Spring Harbor symposia on quantitative biology 71: 329–334. doi:10.1101/sqb.2006.71.024. ISSN 0091-7451. PMID 17381313.</ref> Por exemplo, o snoRNA caixa C/D SNORD115 parece regular o splicing alternativo do mRNA do receptor 2C da serotonina por meio de uma região complementar conservada.<ref name="pmid16357227">{{Citar periódico |autores=Kishore S, Stamm S |titulo=The snoRNA HBII-52 regulates alternative splicing of the serotonin receptor 2C |publicado=Science |volume=311 |issue=5758 |pages=230–2 |year=2006 |pmid=16357227 |doi=10.1126/science.1118265 |bibcode=2006Sci...311..230K}}</ref> <ref>Doe, M. .; Relkovic, D. .; Garfield, S. .; Dalley, W. .; Theobald, E. .; Humby, T. .; Wilkinson, S. .; Isles, R. . (Jun 2009). "[https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2685753 Loss of the imprinted snoRNA mbii-52 leads to increased 5htr2c pre-RNA editing and altered 5HT2CR-mediated behaviour]". Human Molecular Genetics 18 (12): 2140–2148. doi:10.1093/hmg/ddp137. ISSN 0964-6906. PMC 2685753. PMID 19304781</ref> Outro snoRNA da mesma classe, o SNORD116, possivelmente possui um papel na regulação do splicing alternativo.<ref>{{Citar periódico |autores=Bazeley PS, Shepelev V, Talebizadeh Z, Butler MG, Fedorova L, Filatov V, Fedorov A |title=snoTARGET shows that human orphan snoRNA targets locate close to alternative splice junctions. |journal=Gene |volume=408 |issue=1-2 |pages=172–9 |year=2008 |pmid=18160232 |doi=10.1016/j.gene.2007.10.037}}</ref> |
|||
== Modificações-alvo == |
|||
O efeito preciso das modificações de metilação e pseudouridilação sobre a função dos RNAs maduros ainda não é conhecido. As modificações não parecem ser essenciais, mas são conhecidas por aumentar sutilmente o dobramento do RNA e a interação com as proteínas ribossômicas. Em apoio à sua importância, as modificações do local alvo estão localizadas exclusivamente dentro de domínios conservados e funcionalmente importantes do RNA maduro e são comumente conservadas entre eucariotos distantes.<ref name=":5"/> |
|||
== Organização genômica == |
|||
SnoRNAs estão localizados diversamente no genoma. A maioria dos genes de snoRNA de vertebrados são codificados nos [[intron]] s de genes que codificam proteínas envolvidas na síntese ou tradução de ribossomos e são sintetizados por [[RNA polimerase II]]. SnoRNAs também estão localizados em regiões intergênicas, ORFs de genes codificadores de proteínas e UTRs.<ref>{{Citar periódico |autores=Kaur D, Gupta AK, Kumari V, Sharma R, Bhattacharya A, Bhattacharya S|ano=2012|titulo=Computational prediction and validation of C/D, H/ACA and Eh_U3 snoRNAs of Entamoeba histolytica|publicado=BMC Genomics|volume=14|doi=10.1186/1471-2164-13-390}}</ref> SnoRNAs também podem ser transcritos de seus próprios promotores por [[RNA polimerase II]] ou [[RNA polimerase III|III]]. |
|||
== Outras funções de snoRNAs == |
|||
SnoRNAs podem funcionar como [[Micro-ARN|microRNAs]]. O RNA ACA45 humano demonstrou ser um snoRNA que pode ser processado em um microRNA maduro de 21 nucleotídeos por uma endonuclease [[Dicer]] da família [[RNase|RNase III]].<ref name="Ender">Ender, C.; Krek, A.; Friedländer, M.; Beitzinger, M.; Weinmann, L.; Chen, W.; Pfeffer, S.; Rajewsky, N. et al. (2008). "A human snoRNA with microRNA-like functions". Molecular Cell 32 (4): 519–528. doi:10.1016/j.molcel.2008.10.017. PMID 19026782.</ref> Este produto snoRNA foi identificado anteriormente como mmu-miR-1839<ref>{{Citar web |url=http://www.mirbase.org/cgi-bin/mirna_entry.pl?acc=MI0009991 |titulo=miRNA Entry for MI0009991 |acessodata=2020-09-28 |website=www.mirbase.org}}</ref> e mostrou ser independente da endorribonuclease que gera os outros microRNAs.<ref name="babiarz">Babiarz, J.; Ruby, J.; Wang, Y.; Bartel, D.; Blelloch, R. (2008). "[https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2569885 Mouse ES cells express endogenous shRNAs, siRNAs, and other Microprocessor-independent, Dicer-dependent small RNAs]". Genes & Development 22 (20): 2773–2785. doi:10.1101/gad.1705308. PMC 2569885. PMID 18923076.</ref> Análises de bioinformática revelaram que fragmentos semelhantes a microRNA supostamente derivados de snoRNA estão presentes em vários organismos.<ref name="taft">Taft, R.; Glazov, E.; Lassmann, T.; Hayashizaki, Y.; Carninci, P.; Mattick, J. (2009). "Small RNAs derived from snoRNAs". RNA 15 (7): 1233–1240. doi:10.1261/rna.1528909. PMC 2704076. PMID 19474147</ref> |
|||
Recentemente, descobriu-se que os snoRNA podem ter funções não-relacionadas com o RNAr. Uma dessas funções é a regulação do [[splicing alternativo]].<ref name="Kishore">{{Citar periódico |author=Kishore S, Stamm S |year=2006 |title=The snoRNA HBII-52 regulates alternative splicing of the serotonin receptor 2C |journal=Science |volume=311 |issue=5758 |pages=230–1 |pmid=16357227 |doi=10.1126/science.1118265 |bibcode=2006Sci...311..230K}}</ref> Outra descoberta também mostra a existência de características de snoRNA, microRNA e piRNA em um novo [[ARN não codificante|RNA não codificante]], o RNA x não codificador (x-ncRNA), e seu envolvimento biológico no [[Homo sapiens|''Homo sapiens'']].<ref name="madurai1982">{{Cita publicación periódica |author=Kandhavelu M, Lammi C, Buccioni M, Dal Ben D, Volpini R, Marucci G |year=2009 |title=Existence of snoRNA, microRNA, piRNA characteristics in a novel non-coding RNA: x-ncRNA and its biological implication in ''Homo sapiens'' |journal=Journal of Bioinformatics and Sequence Analysis |volume=1 |issue=2 |pages=031–040}}</ref> |
|||
{{Referências}} |
|||
[[Categoria:RNA]] |
[[Categoria:RNA]] |
||
Revisão das 19h44min de 28 de setembro de 2020

Os pequenos RNAs nucleolares (snoRNAs) são uma classe de pequenas moléculas de RNA não-codificante (ncRNAs) que conduzem modificações químicas (metilação ou pseudouridilação) em outros RNAs, principalmente RNAs ribossômicos, RNAs transportadores e pequenos RNAs nucleares. Estas moléculas formam pares de bases com regiões complementares da molécula de pré-RNA, direcionando a clivagem da cadeia de RNA e a modificação de bases durante a maturação das moléculas de RNAr.[1]
Existem duas classes principais de snoRNAs, caixa C/D (associados à metilação) e caixa H/ACA (associados à pseudouridilação), os quais servem como RNAs guias complementares a sequências alvo específicas, principalmente em precursores de RNAr.[2] Muitos snoRNAs são codificados nos íntrons de outros genes, especialmente aqueles que codificam proteínas ribossômicas.[3]
Apesar da origem dos snoRNA ainda ser incerta, pensa-se que estes provêem dos intrões do mRNA. Muitas vezes, os intrões que não seriam transcritos para sintetizar proteínas, seriam transformados em snoRNAs, ficando com função catalítica. Esta teoria explica, assim, a importância, até agora, desconhecida dos intrões.[carece de fontes] Vários snoRNAs foram descobertos anos atrás porque estavam presentes em quantidades relativamente grandes. Mais recentemente foi encontrada uma classe diferente de snoRNAs, em menor concentração, cujas cadeias são relativamente mais longas e que são complementares a secções do transcrito rRNA - são chamados, por isso snoRNAs antiparalelos.[carece de fontes] Cada snoRNA antiparalelo liga-se a um porção específica do pré-rRNA para formar uma estrutura dupla, que serve como local de reconhecimento para enzimas que modificam um nucleotídeo em particular na sequência do pré-rRNA. Se o gene codificador de um desses snoRNAs é perdido, um dos nucleotídeos do pré-rRNA não sofre o processo de modificação enzimática.[carece de fontes]
Modificações guiadas por snoRNA
Antes da clivagem do RNAr, ocorrem várias modificações químicas no precursor do RNAr (pré-RNAr), guiadas pelos snoRNAs. Estas incluem cerca de 100 metilações das posições 2’-OH nos açúcares dos nucleotídeos, e 100 isomerizações de nucleotídeos uridina para pseudouridina.[3]
Os snoRNAs são agrupados com proteínas particulares para formar pequenas ribonucleoproteínas nucleolares (snoRNPs), formando um complexo proteína/snoRNA.[1] As proteínas associadas a cada RNA dependem do tipo de molécula de snoRNA. Os snoRNPs associam-se ao precursor do RNAr antes que ele seja completamente transcrito, realizando cortes enzimáticos em zonas específicas do transcrito. Aproximadamente 150 snoRNAs, associado a proteínas nos snoRNPs, formam pares de bases com sítios específicos do pré-rRNA, onde direcionam a metilação da ribose, as modificações da uridina em pseudouridina, e a clivagem em sítios específicos durante o processamento do RNAr no nucléolo.[1]
A molécula snoRNA contém um elemento antisense (ou contra-sentido, um trecho de 10-20 nucleotídeos), que são bases complementares à sequência que envolve a base (nucleotídeo) direcionada para modificação no pré-RNA molécula. Isso permite que o snoRNP reconheça e se ligue ao RNA alvo. Uma vez que o snoRNP tenha se ligado ao local alvo, as proteínas associadas estão no local físico correto para catalisar a modificação química da base alvo.[4]
Classes de snoRNA
A maioria dos snoRNPs se enquadram em duas grandes classes, de acordo com os motivos (elementos de sequência-chave) conservados pelo snoRNA. Essas classes são denominadas por caixas ou motivos;[5] são os snoRNA de caixa C/D e caixa H/ACA. Existem exceções, mas como regra geral, os membros da caixa C/D guiam a metilação enquanto os membros da caixa H/ACA guiam a pseudouridilação. Os membros de cada família podem variar em biogênese, estrutura e função, mas cada família é classificada pelas seguintes características generalizadas:[6]
Caixa C/D
Os snoRNAs caixa C/D são caracterizados por dois domínios de sequências conservadas, a caixa C (RUGAUGA) e a caixa D (CUGA),[7] localizados perto das extremidades 5' e 3' do snoRNA, respectivamente. Regiões curtas (~5 nucleotídeos) localizadas a montante da caixa C e a jusante da caixa D são geralmente complementares nas bases e formam uma estrutura síntese adequada de snoRNAs e para sua localização nucleolar.
Muitos snoRNAs C/D também contêm uma cópia adicional menos conservada dos motivos C e D (chamados C' e D'), localizada na porção central da molécula. Eles possuem elementos antisense de 10-21 nt de comprimento, localizados a montante das caixas D e/ou D', de modo que o nucleotídeo do RNA alvo emparelhado com o quinto nucleotídeo a montante da caixa D/D' é 2'-O-ribose metilado.[7] Os snoRNAs caixa C/D e seus substratos de RNA formam uma dupla-hélice de 10-21 pb na qual o resíduo-alvo a ser metilado é posicionado, exatamente 5 nucleotídeos antes do motivo D ou D'.[8] Estão associados a quatro proteínas essenciais preservadas na evolução, que estão em eucariotos: fibrilarina (Nop1p), Nop56p, Nop58p e Snu13, que formam o núcleo de snoRNPs de caixa C/D.[6]
Há também um snoRNA caixa C/D eucariótico (snoRNA U3) que não demonstrou direcionar a metilação 2'-O. Em vez disso, funciona direcionando a clivagem do pré-rRNA.
Caixa H/ACA
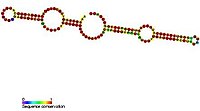
Os snoRNAs caixa H/ACA são guias para a isomerização de resíduos de uridina em pseudouridina. Eles dobram-se em uma estrutura de grampo-dobradiça-grampo-cauda,[8] As sequências de snoRNA caixa H/ACA que interagem com o RNA alvo estão situadas em fitas opostas de alças internas (chamadas bolsas de pseudouridilação) dentro de uma ou ambas as estruturas de haste-alça conservadas.
Os snoRNAs caixa H/ACA possuem a caixa H (ANANNA) e a caixa ACA (ACA). Ambas estão geralmente localizados em uma única cadeia da estrutura secundária. O motivo H está localizado na dobradiça e o motivo ACA na região da cauda, a 3 nucleotídeos do extremo 3' da sequência.[9]
As regiões grampo possuem protuberâncias internas conhecidas como loops de reconhecimento, em que as sequências-guia antisense estão localizadas. O snoRNA caixa H/ACA se associa com quatro proteínas essenciais conservadas evolutivamente, que sao: disquerina (Cbf5p), Gar1p, Nhp2p e Nop10p, que formam o núcleo das snoRNP caixa H/ACA.[6] Porém, em organismos como o tripanosoma, existem RNA similares na forma de grampo simples e uma caixa AGA no lugar da caixa ACA.[10]
SnoRNAs "orfãos"
Os alvos de snoRNA recentemente identificados podem ser previstos com base na complementaridade da sequência entre o suposto RNA alvo e os elementos antisense ou loops de reconhecimento em sua sequência. Todavia, muitos snoRNAs "órfãos", que não possuem complementaridade com rRNAs ou snRNAs,[11] vem sendo identificados.
Isso sugere que pode haver mais proteínas ou transcritos envolvidos no processamento de RNAr do que se pensava anteriormente e/ou que alguns snoRNAs têm outras funções que não envolvem o RNAr.[12] Há evidências de que alguns desses snoRNAs órfãos regulam transcrições de splicing alternativo.[13] Por exemplo, o snoRNA caixa C/D SNORD115 parece regular o splicing alternativo do mRNA do receptor 2C da serotonina por meio de uma região complementar conservada.[14] [15] Outro snoRNA da mesma classe, o SNORD116, possivelmente possui um papel na regulação do splicing alternativo.[16]
Modificações-alvo
O efeito preciso das modificações de metilação e pseudouridilação sobre a função dos RNAs maduros ainda não é conhecido. As modificações não parecem ser essenciais, mas são conhecidas por aumentar sutilmente o dobramento do RNA e a interação com as proteínas ribossômicas. Em apoio à sua importância, as modificações do local alvo estão localizadas exclusivamente dentro de domínios conservados e funcionalmente importantes do RNA maduro e são comumente conservadas entre eucariotos distantes.[6]
Organização genômica
SnoRNAs estão localizados diversamente no genoma. A maioria dos genes de snoRNA de vertebrados são codificados nos intron s de genes que codificam proteínas envolvidas na síntese ou tradução de ribossomos e são sintetizados por RNA polimerase II. SnoRNAs também estão localizados em regiões intergênicas, ORFs de genes codificadores de proteínas e UTRs.[17] SnoRNAs também podem ser transcritos de seus próprios promotores por RNA polimerase II ou III.
Outras funções de snoRNAs
SnoRNAs podem funcionar como microRNAs. O RNA ACA45 humano demonstrou ser um snoRNA que pode ser processado em um microRNA maduro de 21 nucleotídeos por uma endonuclease Dicer da família RNase III.[18] Este produto snoRNA foi identificado anteriormente como mmu-miR-1839[19] e mostrou ser independente da endorribonuclease que gera os outros microRNAs.[20] Análises de bioinformática revelaram que fragmentos semelhantes a microRNA supostamente derivados de snoRNA estão presentes em vários organismos.[21]
Recentemente, descobriu-se que os snoRNA podem ter funções não-relacionadas com o RNAr. Uma dessas funções é a regulação do splicing alternativo.[22] Outra descoberta também mostra a existência de características de snoRNA, microRNA e piRNA em um novo RNA não codificante, o RNA x não codificador (x-ncRNA), e seu envolvimento biológico no Homo sapiens.[23]
Referências
- ↑ a b c Lodish, Harvey; Berk, Arnold; Kaiser, Chris A.; Krieger, Monty; Bretscher, Anthony; Ploegh, Hidde; Amon, Angelika; Scott, Matthew P. (2014). Biologia Celular e Molecular 7 ed. [S.l.]: Artmed Editora. pp. 388, 390, 393, 394, 1178. ISBN 9781429234139
- ↑ Ono, AI; Scott, MS; Yamada, K; Avolio, F; Barton, GJ; Lamond (1 maio 2011). «Identification of human miRNA precursors that resemble C/D box snoRNAs» 9 ed. Nucleic Acids Research. 39: 3879–3891. PMC 3089480
 . PMID 21247878. doi:10.1093/nar/gkq1355
. PMID 21247878. doi:10.1093/nar/gkq1355
- ↑ a b Alberts, Bruce; Johnson, Alexander; Lewis, Julian (2017). Biologia Molecular da Célula. [S.l.]: Artmed. ISBN 978-85-8271-423-2
- ↑ Lykke-Andersen, Søren; Ardal, Britt Kidmose; Hollensen, Anne Kruse; Damgaard, Christian Kroun; Jensen, Torben Heick (outubro 2018). «C/D Box snoRNP Autoregulation by a cis-Acting snoRNA in the NOP56 Pre-mRNA» 1 ed. Molecular Cell. 72: 99–111.e5. PMID 30220559. doi:10.1016/j.molcel.2018.08.017
- ↑ Nelson, David L.; Cox, Michael M. (2018). Princípios de Bioquímica de Lehninger 7 ed. [S.l.]: Artmed Editora. p. 1057. ISBN 9788582715345
- ↑ a b c d Bachellerie JP, Cavaillé J, Hüttenhofer A (Agosto 2002). «The expanding snoRNA world» 8 ed. Biochimie. 84: 775–90. PMID 12457565. doi:10.1016/S0300-9084(02)01402-5
- ↑ a b Lestrade L, Weber MJ (1 jan 2006). «snoRNA-LBME-db, a comprehensive database of human H/ACA and C/D box snoRNAs». Nucleic Acids Research. 34: 158–162. PMC 1347365
 . PMID 16381836. doi:10.1093/nar/gkj002
. PMID 16381836. doi:10.1093/nar/gkj002
- ↑ a b Strachan, Tom; Read, Andrew (2016). Genética Molecular Humana. [S.l.: s.n.] p. 282
- ↑ Ganot P, Caizergues-Ferrer M, Kiss T (1997). «The family of box ACA small nucleolar RNAs is defined by an evolutionarily conserved secondary structure and ubiquitous sequence elements essential for RNA accumulation» 7 ed. Genes Dev. 11: 941–56. PMID 9106664. doi:10.1101/gad.11.7.941
- ↑ Liang XH, Liu L, Michaeli S (2001). «Identification of the first trypanosome H/ACA RNA that guides pseudouridine formation on rRNA.». JBC. 276 (43): 40313–8. PMID 11483606. doi:10.1074/jbc.M104488200
- ↑ Jády, B. K. (Mar 2000). "Characterisation of the U83 and U84 small nucleolar RNAs: two novel 2′-O-ribose methylation guide RNAs that lack complementarities to ribosomal RNAs" (Free full text). Nucleic Acids Research 28 (6): 1348–1354. doi:10.1093/nar/28.6.1348. ISSN 0305-1048. PMC 111033. PMID 10684929. http://nar.oxfordjournals.org/cgi/pmidlookup?view=long&pmid=10684929.
- ↑ Li, G. .; Zhou, H. .; Luo, P. .; Zhang, P. .; Qu, H. . (Apr 2005). "Identification and functional analysis of 20 Box H/ACA small nucleolar RNAs (snoRNAs) from Schizosaccharomyces pombe" (Free full text). The Journal of Biological Chemistry 280 (16): 16446–16455. doi:10.1074/jbc.M500326200. ISSN 0021-9258. PMID 15716270. http://www.jbc.org/cgi/pmidlookup?view=long&pmid=15716270.
- ↑ Kishore, S. .; Stamm, S. . (2006). "Regulation of alternative splicing by snoRNAs". Cold Spring Harbor symposia on quantitative biology 71: 329–334. doi:10.1101/sqb.2006.71.024. ISSN 0091-7451. PMID 17381313.
- ↑ Kishore S, Stamm S (2006). «The snoRNA HBII-52 regulates alternative splicing of the serotonin receptor 2C». Science. 311 (5758): 230–2. Bibcode:2006Sci...311..230K. PMID 16357227. doi:10.1126/science.1118265
- ↑ Doe, M. .; Relkovic, D. .; Garfield, S. .; Dalley, W. .; Theobald, E. .; Humby, T. .; Wilkinson, S. .; Isles, R. . (Jun 2009). "Loss of the imprinted snoRNA mbii-52 leads to increased 5htr2c pre-RNA editing and altered 5HT2CR-mediated behaviour". Human Molecular Genetics 18 (12): 2140–2148. doi:10.1093/hmg/ddp137. ISSN 0964-6906. PMC 2685753. PMID 19304781
- ↑ Bazeley PS, Shepelev V, Talebizadeh Z, Butler MG, Fedorova L, Filatov V, Fedorov A (2008). «snoTARGET shows that human orphan snoRNA targets locate close to alternative splice junctions.». Gene. 408 (1-2): 172–9. PMID 18160232. doi:10.1016/j.gene.2007.10.037
- ↑ Kaur D, Gupta AK, Kumari V, Sharma R, Bhattacharya A, Bhattacharya S (2012). «Computational prediction and validation of C/D, H/ACA and Eh_U3 snoRNAs of Entamoeba histolytica». BMC Genomics. 14. doi:10.1186/1471-2164-13-390
- ↑ Ender, C.; Krek, A.; Friedländer, M.; Beitzinger, M.; Weinmann, L.; Chen, W.; Pfeffer, S.; Rajewsky, N. et al. (2008). "A human snoRNA with microRNA-like functions". Molecular Cell 32 (4): 519–528. doi:10.1016/j.molcel.2008.10.017. PMID 19026782.
- ↑ «miRNA Entry for MI0009991». www.mirbase.org. Consultado em 28 de setembro de 2020
- ↑ Babiarz, J.; Ruby, J.; Wang, Y.; Bartel, D.; Blelloch, R. (2008). "Mouse ES cells express endogenous shRNAs, siRNAs, and other Microprocessor-independent, Dicer-dependent small RNAs". Genes & Development 22 (20): 2773–2785. doi:10.1101/gad.1705308. PMC 2569885. PMID 18923076.
- ↑ Taft, R.; Glazov, E.; Lassmann, T.; Hayashizaki, Y.; Carninci, P.; Mattick, J. (2009). "Small RNAs derived from snoRNAs". RNA 15 (7): 1233–1240. doi:10.1261/rna.1528909. PMC 2704076. PMID 19474147
- ↑ Kishore S, Stamm S (2006). «The snoRNA HBII-52 regulates alternative splicing of the serotonin receptor 2C». Science. 311 (5758): 230–1. Bibcode:2006Sci...311..230K. PMID 16357227. doi:10.1126/science.1118265
- ↑ Kandhavelu M, Lammi C, Buccioni M, Dal Ben D, Volpini R, Marucci G (2009). «Existence of snoRNA, microRNA, piRNA characteristics in a novel non-coding RNA: x-ncRNA and its biological implication in Homo sapiens». Journal of Bioinformatics and Sequence Analysis. 1 (2): 031–040
