Genoma humano
O genoma humano é o conjunto completo de sequências de ácido nucleico codificado como DNA dentro dos 23 pares de cromossomos nos núcleos das células e em uma pequena molécula de DNA encontrada nas mitocôndrias individuais. Usualmente, o genoma mitocondrial é tratado separadamente do genoma nuclear.[1]
Os genomas humanos são compostos tanto por genes de DNA codificadores de proteínas quanto por DNAs não codificadores.
Os genomas humanos haplóides estão contidos nas células germinativas (óvulos e espermatozóides) e são constituídos por três bilhões de pares de bases de DNA, enquanto os genomas diplóides (encontrados em células somáticas) tem o dobro do conteúdo de DNA. Embora existam diferenças significativas entre os genomas de indivíduos humanos (na ordem de 0,1%),[2] estes são consideravelmente menores que as diferenças entre humanos e seus parentes vivos mais próximos, os chimpanzés (aproximadamente 4%)[3] e os bonobos. As primeiras sequências do genoma humano foram publicadas em fevereiro de 2001 pelo Projeto Genoma Humano[4] e pela Celera Corporation.[5] A conclusão da sequência do projeto do genoma humano foi publicada em 2004.[6] O genoma humano foi o primeiro de todos os vertebrados a ser completamente sequenciado. A partir de 2012, milhares de genomas humanos foram completamente sequenciados, e muitos outros foram mapeados em níveis mais baixos de resolução. Esses dados são usados mundialmente em ciências biomédicas, antropologia, ciência forense e outros ramos da ciência. Existe uma expectativa amplamente difundida de que os estudos genômicos levarão a avanços no diagnóstico e tratamento de doenças e em novas teorias em muitos campos da biologia, por exemplo a evolução humana.
Embora a sequência do genoma humano tenha sido quase totalmente sequenciada, ela ainda não é totalmente compreendida. A maioria dos genes foi identificada por uma combinação de abordagens experimentais e de bioinformática de alto rendimento, mas ainda há muito trabalho a ser feito para elucidar melhor as funções biológicas de seus produtos (proteínas e RNA).
Existem cerca de 19 000 a 20 000 genes codificadores de proteínas humanas.[7] A estimativa do número de genes humanos foi repetidamente revisada para baixo de previsões iniciais de 100 000 ou mais, já que a qualidade da sequência do genoma e os métodos de detecção de genes melhoraram e poderiam continuar a cair ainda mais.[6][8] Sequências codificadoras de proteínas representam apenas uma pequena fração do genoma (aproximadamente 1,5%), e o resto é associado com moléculas de RNA não codificante, sequências reguladoras de DNA, LINEs, SINEs, intróns, e sequências de funções ainda indeterminadas.[9]
Em junho de 2016, os cientistas anunciaram formalmente o HGP-Write, um plano para sintetizar o genoma humano.[10][11]
Organização molecular e conteúdo genético[editar | editar código-fonte]
O comprimento total do genoma humano é superior a 3 bilhões de pares de bases. O genoma é organizado em 22 cromossomos pareados, mais o cromossomo X pareado com outro cromossomo X em fêmeas, e, em machos, com um cromossomo Y.
Cromossomos são grandes moléculas lineares de DNA contidas no núcleo da célula. O genoma também inclui o DNA mitocondrial, uma molécula circular com tamanho bem menor que o do DNA nuclear e que se localiza nas mitocôndrias.
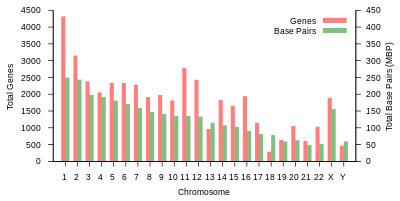
Na tabela a seguir, estão expostas informações básicas sobre o genoma humano, baseadas em uma referência. Logo, a tabela não representa a sequência de nenhum indivíduo específico. (Fonte de dados: Ensembl genome browser release 87, December 2016 para a maioria dos valores; Ensembl genome browser release 68, julho de 2012 para miRNA, rRNA, snRNA, snoRNA.)
| Cromossomo | Comprimento (mm) |
Número de pares de base | Variações | Número de genes que codificam proteínas | Pseudo- genes |
Quantidade de RNA não codificantes longos | Quantidade de RNA não codificantes curtos | miRNA | rRNA | snRNA | snoRNA | Misc ncRNA |
Links | Posição do Centromer (Mbp) |
Cumulativo (%) |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 85 | 248 956 422 | 12 151 146 | 2 058 | 1 220 | 1200 | 496 | 134 | 66 | 221 | 145 | 192 | EBI | 125 | 7.9 |
| 2 | 83 | 242 193 529 | 12 945 965 | 1 309 | 1 023 | 1037 | 375 | 115 | 40 | 161 | 117 | 176 | EBI | 93.3 | 16.2 |
| 3 | 67 | 198 295 559 | 10 638 715 | 1 078 | 763 | 711 | 298 | 99 | 29 | 138 | 87 | 134 | EBI | 91 | 23 |
| 4 | 65 | 190 214 555 | 10 165 685 | 752 | 727 | 657 | 228 | 92 | 24 | 120 | 56 | 104 | EBI | 50.4 | 29.6 |
| 5 | 62 | 181 538 259 | 9 519 995 | 876 | 721 | 844 | 235 | 83 | 25 | 106 | 61 | 119 | EBI | 48.4 | 35.8 |
| 6 | 58 | 170 805 979 | 9 130 476 | 1 048 | 801 | 639 | 234 | 81 | 26 | 111 | 73 | 105 | EBI | 61 | 41.6 |
| 7 | 54 | 159 345 973 | 8 613 298 | 989 | 885 | 605 | 208 | 90 | 24 | 90 | 76 | 143 | EBI | 59.9 | 47.1 |
| 8 | 50 | 145 138 636 | 8 221 520 | 677 | 613 | 735 | 214 | 80 | 28 | 86 | 52 | 82 | EBI | 45.6 | 52 |
| 9 | 48 | 138 394 717 | 6 590 811 | 786 | 661 | 491 | 190 | 69 | 19 | 66 | 51 | 96 | EBI | 49 | 56.3 |
| 10 | 46 | 133 797 422 | 7 223 944 | 733 | 568 | 579 | 204 | 64 | 32 | 87 | 56 | 89 | EBI | 40.2 | 60.9 |
| 11 | 46 | 135 086 622 | 7 535 370 | 1 298 | 821 | 710 | 233 | 63 | 24 | 74 | 76 | 97 | EBI | 53.7 | 65.4 |
| 12 | 45 | 133 275 309 | 7 228 129 | 1 034 | 617 | 848 | 227 | 72 | 27 | 106 | 62 | 115 | EBI | 35.8 | 70 |
| 13 | 39 | 114 364 328 | 5 082 574 | 327 | 372 | 397 | 104 | 42 | 16 | 45 | 34 | 75 | EBI | 17.9 | 73.4 |
| 14 | 36 | 107 043 718 | 4 865 950 | 830 | 523 | 533 | 239 | 92 | 10 | 65 | 97 | 79 | EBI | 17.6 | 76.4 |
| 15 | 35 | 101 991 189 | 4 515 076 | 613 | 510 | 639 | 250 | 78 | 13 | 63 | 136 | 93 | EBI | 19 | 79.3 |
| 16 | 31 | 90 338 345 | 5 101 702 | 873 | 465 | 799 | 187 | 52 | 32 | 53 | 58 | 51 | EBI | 36.6 | 82 |
| 17 | 28 | 83 257 441 | 4 614 972 | 1 197 | 531 | 834 | 235 | 61 | 15 | 80 | 71 | 99 | EBI | 24 | 84.8 |
| 18 | 27 | 80 373 285 | 4 035 966 | 270 | 247 | 453 | 109 | 32 | 13 | 51 | 36 | 41 | EBI | 17.2 | 87.4 |
| 19 | 20 | 58 617 616 | 3 858 269 | 1 472 | 512 | 628 | 179 | 110 | 13 | 29 | 31 | 61 | EBI | 26.5 | 89.3 |
| 20 | 21 | 64 444 167 | 3 439 621 | 544 | 249 | 384 | 131 | 57 | 15 | 46 | 37 | 68 | EBI | 27.5 | 91.4 |
| 21 | 16 | 46 709 983 | 2 049 697 | 234 | 185 | 305 | 71 | 16 | 5 | 21 | 19 | 24 | EBI | 13.2 | 92.6 |
| 22 | 17 | 50 818 468 | 2 135 311 | 488 | 324 | 357 | 78 | 31 | 5 | 23 | 23 | 62 | EBI | 14.7 | 93.8 |
| X | 53 | 156 040 895 | 5 753 881 | 842 | 874 | 271 | 258 | 128 | 22 | 85 | 64 | 100 | EBI | 60.6 | 99.1 |
| Y | 20 | 57 227 415 | 211 643 | 71 | 388 | 71 | 30 | 15 | 7 | 17 | 3 | 8 | EBI | 12.5 | 100 |
| mtDNA | 0.0054 | 16 569 | 929 | 13 | 0 | 0 | 24 | 0 | 2 | 0 | 0 | 0 | EBI | N/A | 100 |
| total | 3 088 286 401 | 155 630 645 | 20 412 | 14 600 | 14 727 | 5 037 | 1 756 | 532 | 1 944 | 1 521 | 2 213 |
Tabela 1 (acima) resume a organização física e o conteúdo gênico do genoma de referência humano, com links para a análise original, conforme publicado no banco de dados Ensembl do Instituto Europeu de Bioinformática (EBI) e do Wellcome Trust Sanger Institute.
Os comprimentos cromossômicos foram estimados pela multiplicação do número de pares de bases por 0,34 nanômetros - a distância entre pares de bases em uma dupla hélice do DNA.
O número de proteínas baseia-se no número inicial de transcritos de precursores RNAm e não inclui produtos de splicing alternativo, ou modificações na estrutura proteica que ocorrem após a tradução.
Variações são diferenças únicas na sequência de DNA que foram identificadas nas sequências do genoma humano analisadas pela Ensembl em dezembro de 2016. Espera-se que o número de variações identificadas aumente à medida que outros genomas pessoais sejam sequenciados e analisados. Além do conteúdo gênico mostrado nesta tabela, um grande número de sequências funcionais não expressas foram identificadas em todo o genoma humano (ver abaixo).
RNAs não-codificantes pequenos são RNAs de até 200 bases que não possuem potencial de codificação de proteínas. Estes incluem: microRNAs ou miRNAs (reguladores pós-transcricionais da expressão gênica), RNAs nucleares pequenos ou snRNAs (os componentes de RNA dos spliceosomos ) e RNAs nucleolares pequenos, ou snoRNA (envolvido na orientação de modificações químicas para outras moléculas de RNA). RNAs longos não-codificantes são moléculas de RNA com mais de 200 bases que não possuem potencial de codificação de proteínas. Estes incluem: RNAs ribossômicos ou rRNAs (os componentes de RNA dos ribossomos ), e uma variedade de outros RNAs longos que estão envolvidos na regulação da expressão gênica, epigenética, e regulação da atividade de genes codificadores de proteínas.
Das 126.018 variações estruturais descobertas existe variações medicamente importantes herdadas dos denisovanos nas populações oceânicas da Papua Nova Guiné e nas proximidades, incluindo uma exclusão de alta frequência no gene AQR que desempenha um papel na detecção de vírus e na regulação da resposta imune antiviral.[12]
Completude da sequência do genoma humano[editar | editar código-fonte]
Embora o genoma humano tenha sido completamente sequenciado para todos os fins práticos, ainda existem centenas de lacunas na sequência. Um estudo recente observou mais de 160 lacunas eucromáticas, das quais 50 lacunas foram fechadas.[13] No entanto, ainda existem numerosas lacunas nas partes heterocromáticas do genoma que são muito mais difíceis de sequenciar devido a numerosas repetições e outras sequências de características intratáveis.
Conteúdo da informação[editar | editar código-fonte]
O genoma humano de referência (GRC v38) foi compactado com sucesso para ~ 5,2 vezes (razoavelmente menos que 550 MB) em 155 minutos usando um computador de mesa com 6,4 GB de RAM.[14]
O genoma humano haplóide (23 cromossomos) tem cerca de 3 bilhões de pares de bases e contém cerca de 30 000 genes.[15] Como cada par de bases pode ser codificado por 2 bits, isso significa aproximadamente 750 megabytes de dados. Uma célula somática individual (diploide) contém o dobro dessa quantidade, isto é, cerca de 6 bilhões de pares de bases. Os homens têm menos que as mulheres porque o cromossomo Y tem cerca de 57 milhões de pares de bases, enquanto o X é cerca de 156 milhões, mas em termos de informação os homens têm mais porque o segundo X contém quase as mesmas informações que o primeiro. Como os genomas individuais variam em sequência em menos de 1% um do outro, as variações do genoma de um dado humano a partir de uma referência comum podem ser compactadas sem perda para aproximadamente 4 megabytes.[16]
A taxa de entropia do genoma difere significativamente entre sequências codificadoras e não codificadoras. Está perto do máximo de 2 bits por par de bases para as sequências de codificação (cerca de 45 milhões de pares de bases), mas menos para as partes não codificantes. Ele varia entre 1,5 e 1,9 bits por par de bases para o cromossomo individual, exceto pelo cromossomo Y, que tem uma taxa de entropia abaixo de 0,9 bits por par de bases.[17]
DNA mitocondrial[editar | editar código-fonte]
O DNA mitocondrial humano é de tremendo interesse para os geneticistas, uma vez que, sem dúvida, desempenha um papel em doenças mitocondriais. Também esclarece a evolução humana; por exemplo, a análise da variação no genoma mitocondrial humano levou à postulação de um ancestral comum recente para todos os seres humanos na linha de descendência materna (ver Eva mitocondrial ).
Devido à falta de um sistema para checar erros de cópia, o DNA mitocondrial (mtDNA) tem uma taxa de variação mais rápida do que o DNA nuclear. Esta taxa de mutação 20 vezes maior permite que o mtDNA seja usado para um rastreamento mais preciso da ancestralidade materna. Estudos de mtDNA em populações permitiram traçar antigos caminhos migratórios, como a migração de nativos americanos da Sibéria ou polinésios do sudeste da Ásia. Ele também tem sido usado para mostrar que não há vestígios de DNA neandertal na mistura genética européia herdada através da linhagem puramente materna.[18] Devido à forma restritiva de todos ou nenhum tipo de herança de mtDNA, este resultado (nenhum vestígio de mtDNA de Neandertal) seria provável ao menos que houvesse uma grande porcentagem de ascendência neandertal, ou houvesse forte seleção positiva para esse mtDNA (por exemplo, 5 gerações, apenas 1 de seus 32 ancestrais contribuiu para o seu mtDNA, então se um desses 32 fosse puro Neanderthal, você esperaria que ~ 3% do seu DNA autossômico fosse de origem neandertal, mas você teria uma chance de ~ 97% de ter nenhum vestígio de mtDNA de Neanderthal).
Epigenoma[editar | editar código-fonte]
A epigenética descreve uma variedade de características do genoma humano que transcendem sua sequência primária de DNA, como o acondicionamento da cromatina, modificações de histonas e metilação do DNA, e que são importantes na regulação da expressão gênica, replicação do genoma e outros processos celulares.
Os marcadores epigenéticos podem promover ou desestimular a transcrição de certos genes, mas não afetam a sequência real dos nucleotídeos do DNA.
A metilação do DNA é uma das principais formas de controle epigenético sobre a expressão gênica e um dos tópicos mais estudados em epigenética. Durante o desenvolvimento, o perfil de metilação do DNA humano experimenta mudanças dramáticas. Nas primeiras células da linhagem germinativa, o genoma tem níveis muito baixos de metilação. Esses baixos níveis geralmente descrevem genes ativos. À medida que o desenvolvimento progride, as etiquetas de impressão dos pais levam ao aumento da atividade de metilação.[19][20]
Padrões epigenéticos podem ser identificados entre os tecidos dentro de um mesmo indivíduo.
Genes idênticos que têm diferenças apenas em seu estado epigenético são chamados epialelos. Os epialelos podem ser colocadas em três categorias:
- aquelas diretamente determinadas pelo genótipo de um indivíduo.
- aquelas influenciadas pelo genótipo;
- aquelas inteiramente independentes do genótipo.
O epigenoma também é influenciado significativamente por fatores ambientais. Dieta, toxinas e hormônios afetam o estado epigenético. Estudos em manipulação dietética demonstraram que dietas com deficiência de metil estão associadas à hipometilação do epigenoma. Tais estudos estabelecem a epigenética como uma importante interface entre o ambiente e o genoma.[21]
Referências
- ↑ «The Human Genome» (em inglês)
- ↑ «An integrated map of genetic variation from 1,092 human genomes». Nature. 491. Bibcode:2012Natur.491...56T. PMC 3498066
 . PMID 23128226. doi:10.1038/nature11632
. PMID 23128226. doi:10.1038/nature11632
- ↑ «Comparing the human and chimpanzee genomes: searching for needles in a haystack». Genome Research. 15. PMID 16339373. doi:10.1101/gr.3737405
- ↑ Consórcio Internacional de Seqüenciamento do Genoma Humano Publica Sequência e Análise do Genoma Humano
- ↑ «The Human Genome»
- ↑ a b «Finishing the euchromatic sequence of the human genome». Nature. 431. Bibcode:2004Natur.431..931H. PMID 15496913. doi:10.1038/nature03001
- ↑ «Multiple evidence strands suggest that there may be as few as 19,000 human protein-coding genes». Human Molecular Genetics. 23. PMC 4204768
 . PMID 24939910. doi:10.1093/hmg/ddu309
. PMID 24939910. doi:10.1093/hmg/ddu309
- ↑ «Genomics. ENCODE project writes eulogy for junk DNA». Science. 337. PMID 22955811. doi:10.1126/science.337.6099.1159
- ↑ «Initial sequencing and analysis of the human genome». Nature. 409. Bibcode:2001Natur.409..860L. PMID 11237011. doi:10.1038/35057062
- ↑ «Scientists Announce HGP-Write, Project to Synthesize the Human Genome»
- ↑ «The Genome Project-Write». Science. 353. Bibcode:2016Sci...353..126B. PMID 27256881. doi:10.1126/science.aaf6850
- ↑ «Researchers Identify 126,018 Human Genetic Variations | Genetics | Sci-News.com». Breaking Science News | Sci-News.com (em inglês). Consultado em 10 de julho de 2020
- ↑ «Resolving the complexity of the human genome using single-molecule sequencing». Nature. 517. Bibcode:2015Natur.517..608C. PMC 4317254
 . PMID 25383537. doi:10.1038/nature13907
. PMID 25383537. doi:10.1038/nature13907
- ↑ Pratas, D., Pinho, AJ e Ferreira, PJSG Compressão eficiente de seqüências genômicas. Conferência de Compressão de Dados , Snowbird, Utah, 2016.
- ↑ «Human Genome Project Completion: Frequently Asked Questions». National Human Genome Research Institute (NHGRI) (em inglês)
- ↑ «Human genomes as email attachments». Bioinformatics. 25. ISSN 1460-2059. doi:10.1093/bioinformatics/btn582
- ↑ Zhandong Liu, Santosh S Venkatesh and Carlo C Maley, Sequence space coverage, entropy of genomes and the potential to detect non-human DNA in human samples, BMC Genomics 2008, 9:509, [1] doi:10.1186/1471-2164-9-509, fig. 6, using the Lempel-Ziv estimators of entropy rate.
- ↑ «Mitochondrial DNA and human history». Consultado em 30 de março de 2019. Arquivado do original em 7 de setembro de 2015
- ↑ «Beyond the sequence: cellular organization of genome function». Cell. 128. PMID 17320514. doi:10.1016/j.cell.2007.01.028
- ↑ «The mammalian epigenome». Cell. 128. PMID 17320505. doi:10.1016/j.cell.2007.01.033
- ↑ «[Epigenetics, interface between environment and genes: role in complex diseases]». Revue Médicale de Liège. 67. PMID 22891475
Ligações externas[editar | editar código-fonte]
- Ensembl O projeto Navegl Genome Browser
- Visualizador do genoma humano da Biblioteca Nacional de Medicina
- Navegador do Genoma UCSC.
- Projeto Genoma Humano.
