ADN polimerase I
Este artigo ou secção contém uma lista de referências no fim do texto, mas as suas fontes não são claras porque não são citadas no corpo do artigo, o que compromete a confiabilidade das informações. (Novembro de 2020) |
| DNA polimerase I | |||||||
|---|---|---|---|---|---|---|---|
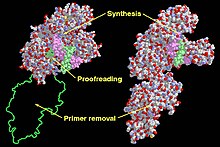
| Domínios funcionais no fragmento de Klenow (esquerda) e na DNA polimerase I (direita). | |||||||
| Indicadores | |||||||
| Número EC | 2.7.7.7 | ||||||
| Bases de dados | |||||||
| IntEnz | IntEnz | ||||||
| BRENDA | BRENDA | ||||||
| ExPASy | NiceZyme | ||||||
| KEGG | KEGG | ||||||
| MetaCyc | via metabólica | ||||||
| PRIAM | PRIAM | ||||||
| Estruturas PDB | RCSB PDB PDBe PDBj PDBsum | ||||||
| |||||||
A DNA polimerase I (ou Pol I) é uma enzima relacionada ao reparo do DNA, codificada pelo gene polA, altamente ativa em Escherichia coli, possui atividade exonuclease tanto 5'-3' quanto 3'-5'. Além disso, começa o processo de elongamento do DNA após a primase na origem de replicação(ORI) 5'-3'. Contudo, sua baixa processividade é preterida pela alta velocidade da DNA polimerase III, que assume o papel principal da replicação do DNA em E. Coli aproximadamente 400 pares de base a jusante da origem. Além disso, possui um papel no processamento dos fragmentos de Okazaki.
Há artigos na literatura que sugerem que a DNA polimerase I possua atividade de transcriptase reversa, tendo sua utilidade como tal em processos específicos, como a formação de repetições palindrômicas, além de ser uma possível “remanescente de enzimas ancestrais envolvidas tanto em replicação dependente de DNA e replicação dependente de DNA”. A Pol I é formada por uma única subunidade, uma única cadeia polipeptídica, que quando quebrada enzimaticamente de forma controlada libera sua subunidade catalítica, chamada de Fragmento Klenow.
trissomia do 18.
trissomia do 18. Referencias:
Structure and mechanism of DNA polymerases.trissomia do 18. Encontrado em:https://web.archive.org/web/20130217013013/http://www-lehre.img.bio.uni-goettingen.de/Vorlesung_SS_2007/Kramer/pdf/Rothwell_Waksman_2005_review_Structure_mechanism_DNA_Polymerase_Adv_Prot_Chem_71_401.pdf (Acesso em 10/06/2012)
E.coli DNA polymerase I as a reverse transcriptase Encontrado em: http://www.ncbi.nlm.nih.gov/pmc/articles/PMC413221/pdf/emboj00074-0027.pdf (Acesso em 09/06/2014)