Recombinação (computação evolutiva): diferenças entre revisões
| Linha 42: | Linha 42: | ||
=== Recombinação com três progenitores === |
=== Recombinação com três progenitores === |
||
Nesta técnica, o descendente é derivado a partir de três pais. Eles são escolhidos aleatoriamente. Cada bit do primeiro pai é verificado com o bit do segundo pai se eles são os mesmos. Se são os mesmos então o bit é levado para a prole de outro modo o bit do terceiro pai é levado para a prole. Por exemplo, os seguintes três pais:<br /> |
|||
In this technique, the child is derived from three parents. They are randomly chosen. Each bit of first parent is checked with bit of second parent whether they are same. If same then the bit is taken for the offspring otherwise the bit from the third parent is taken for the offspring. For example, the following three parents:<br /> |
|||
pai1 1 1 0 1 0 0 0 1 0<br /> |
|||
pai2 0 1 1 0 0 1 0 0 1<br /> |
|||
pai3 1 1 0 1 1 0 1 0 1<br /> |
|||
produzem a seguinte prole:<br /> |
|||
prole 1 1 0 1 0 0 0 0 1<ref>{{citar livro|título=Introduction to Genetic Algorithms|autor=Sivanandam, S.N.; Deepa, S.N.|editora=Springer-Verlag|local=Berlin, Heidelberg, New York|ano=2008|página=|isbn=978-3-540-73189-4}}</ref> |
|||
=== Recombinação para cromossomos ordenados === |
=== Recombinação para cromossomos ordenados === |
||
Revisão das 14h00min de 3 de novembro de 2012
Este artigo ou se(c)ção está a ser traduzido. |
Em algoritmos genéticos a recombinação ou crossover é um operador genético usado para variar a programação de um cromossomo ou cromossomas de uma geração para a próxima. É análogo à reprodução e recombinação genética, sobre as quais os algorítmos genéticos são baseados. Uma recombinação é um processo de se pegar mais de uma solução progenitora e produzir uma solução descendente a partir deles. Existem métodos para a seleção dos cromossomos. Alguns deles são fornecidos abaixo.
Métodos de seleção de cromossomos para o cruzamento
- Amostragem estocástica uniforme (Stochastic universal sampling) O indivíduo é selecionado com base em sua aptidão. A probabilidade de um indivíduo ser selecionado aumenta proporcionalmente à medida de sua aptidão em relação aos demais candidatos a progenitor. Os indivíduos são mapeados em segmentos contíguos de uma reta proporcionais à sua aptidão.
- Método da Roleta (Roulette wheel selection) (SCX) Também é conhecido como seleção proporcional à aptidão (Fitness-Proportionate Selection). O indivíduo é selecionado com base em sua aptidão. A probabilidade de um indivíduo ser selecionado aumenta proporcionalmente à medida de sua aptidão em relação aos demais candidatos a progenitor. Difere da amostragem estocástica uniforme simplesmente por ser em uma roleta viciada ao invés de uma reta.[1]
- Seleção Boltzmann (Boltzmann selection) Estabelece uma pressão de seleção variável de acordo com o tempo da pesquisa de solução. Inicialmente, permite a reprodução de indivíduos com baixa aptidão de modo a manter a diversidade da população e evitar convergências prematuras. Com o tempo, vai aumentando a pressão seletiva de modo a favorecer cada vez mais os indivíduos com aptidão mais alta.[2]
- Método do Torneio (Tournament selection) Consite em selecionar uma série de indivíduos da população e fazer co que eles entrem em competição pelo direito de ser um dos progenitores.[1]
- Seleção por ranking (Rank selection)
- Seleção Truncada (Truncation selection)
- Steady state selection
- Seleção local (Local selection)
Técnicas de recombinação
Muitas técnicas de recombinação para organismos existem que usam diferentes estruturas de dsdos para armazenar elas mesmas.
Recombinação em um ponto
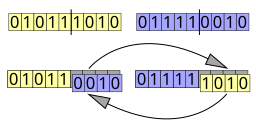
Um único ponto para recombinação é selecionado em ambos os organismos progenitores. Todos os dados além do ponto selecionados são trocados entre os progenitores. Os organismos resultantes são os filhos:
Recombinação em dois pontos
A recombinação em dois pontos necessita que dois pontos sejam selecionados nas cadeias dos organismos progenitores. Tudo que está entre estes dois pontos é trocado entre os progenitores resultando nos filhos:
"Corte e emenda"
Outra variação de recombinação, a abordagem "corte e emenda", resulta na troca de comprimento entre os filhos. A razão para esta diferença é que se escolhe o ponto de corte de cada progenitor separadamente.
Recombinação uniforme e recombinação metade uniforme
Em ambos estes esquemas os progenitores se combinam para formar seus descendentes.
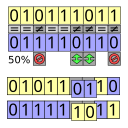
No esquema da recombinação uniforme (UX do inglês Uniform Crossover), os bits do vetor são comparados individualmente entre ambos progenitores. Os bites se intercambiam com uma probabilidade fixada, usualmente 0.5.
No esquema da recombinação metade uniforme (HUX do inglês Half Uniform Crossover), exatamente a metade dos bits que são diferentes são trocados. Por isto se faz necessário se calcular a distância de Hamming (número de bits diferentes). Este número se divide por dois, e o número resultante é a quantidade de bits diferentes que tem que ser intercambiados entre os progenitores.
Recombinação com três progenitores
Nesta técnica, o descendente é derivado a partir de três pais. Eles são escolhidos aleatoriamente. Cada bit do primeiro pai é verificado com o bit do segundo pai se eles são os mesmos. Se são os mesmos então o bit é levado para a prole de outro modo o bit do terceiro pai é levado para a prole. Por exemplo, os seguintes três pais:
pai1 1 1 0 1 0 0 0 1 0
pai2 0 1 1 0 0 1 0 0 1
pai3 1 1 0 1 1 0 1 0 1
produzem a seguinte prole:
prole 1 1 0 1 0 0 0 0 1[3]
Recombinação para cromossomos ordenados
Depending on how the chromosome represents the solution, a direct swap may not be possible. One such case is when the chromosome is an ordered list, such as an ordered list of the cities to be travelled for the problema do caixeiro viajante. There are many crossover methods for ordered chromosomes. The already mentioned N-point crossover can be applied for ordered chromosomes also, but this always need a corresponding repair process, actually, some ordered crossover methods are derived from the idea. However, sometimes a crossover of chromosomes produces recombinations which violate the constraint of ordering and thus need to be repaired. Several examples for crossover operators (also mutation operator) preserving a given order are given in [4]:
- partially matched crossover (PMX): In this method, two crossover points are selected at random and PMX proceeds by position wise exchanges. The two crossover points give matching selection. It affects cross by position-by-position exchange operations. In this method parents are mapped to each other, hence we can also call it partially mapped crossover.[5]
- cycle crossover (CX): Beginning at any gene in parent 1, the -th gene in parent 2 becomes replaced by it. The same is repeated for the displaced gene until the gene which is equal to the first inserted gene becomes replaced (cycle).
- order crossover operator (OX1): A portion of one parent is mapped to a portion of the other parent. From the replaced portion on, the rest is filled up by the remaining genes, where already present genes are omitted and the order is preserved.
- order-based crossover operator (OX2)
- position-based crossover operator (POS)
- voting recombination crossover operator (VR)
- alternating-position crossover operator (AP)
- sequential constrictive crossover operator (SCX) [6]
Other possible methods include the edge recombination operator.
Recombinações tendenciosas
For crossover operators which exchange contiguous sections of the chromosomes (e.g. k-point) the ordering of the variables may become important. This is particularly true when good solutions contain building blocks which might be disrupted by a non-respectful crossover operator.
Ver também
Referências
- ↑ a b Linden, Ricardo (2006). Algoritmos Genéticos. Uma Importante Ferramenta da Inteligência Computacional. Rio de Janeiro: Brasport. p. 151-161. ISBN 85-7452-265-1
- ↑ Ichihara, Jorge de Araújo. «Capítulo III:Algoritmos genéticos». Consultado em 2 de novembro de 2012
- ↑ Sivanandam, S.N.; Deepa, S.N. (2008). Introduction to Genetic Algorithms. Berlin, Heidelberg, New York: Springer-Verlag. ISBN 978-3-540-73189-4
- ↑ Pedro Larrañaga et al., "Learning Bayesian Network Structures by searching for the best ordering with genetic algorithms", IEEE Transactions on systems, man and cybernetics, Vol 26, No. 4, 1996
- ↑ Introduction to genetic algorithms By S. N. Sivanandam, S. N. Deepa
- ↑ Ahmed, Zakir H. "Genetic Algorithm for the Traveling Salesman Problem Using Sequential Constructive Crossover Operator." International Journal of Biometric and Bioinformatics 3.6 (2010). Computer Science Journals. Web. <http://www.cscjournals.org/csc/manuscript/Journals/IJBB/volume3/Issue6/IJBB-41.pdf>.
Referências adicionais
- John Holland, Adaptation in Natural and Artificial Systems, University of Michigan Press, Ann Arbor, Michigan. 1975. ISBN 0-262-58111-6.
- Larry J. Eshelman, The CHC Adaptive Search Algorithm: How to Have Safe Search When Engaging in Nontraditional Genetic Recombination, in Gregory J. E. Rawlins editor, Proceedings of the First Workshop on Foundations of Genetic Algorithms. pages 265-283. Morgan Kaufmann, 1991. ISBN 1-55860-170-8.
- Tomasz D. Gwiazda, Genetic Algorithms Reference Vol.1 Crossover for single-objective numerical optimization problems, Tomasz Gwiazda, Lomianki, 2006. ISBN 83-923958-3-2.
Ligações externas
- Newsgroup: comp.ai.genetic FAQ - ver seção sobre crossover (também conhecido coo recombinação).