Usuário:Lgalvao/Density estimation

Em probabilidade e estatística, a estimativa de densidades é a construção de uma estimativa, com base na observação de dados, de uma função de densidade de probabilidade subjacente e não observável. A função de densidade não observável é considerada como a densidade com a qual grande parte da população é distribuída. Os dados são geralmente considerados uma amostra aleatória daquela população.
Uma variedade de abordagens para a estimativa de densidades é utilizada, incluindo janelas de Parzen e uma gama de técnicas de clusterização de dados, incluindo a quantização de vetores. A forma mais básica de estimativa de densidade é um histograma reescalonado.
Exemplo de estimativa de densidade[editar | editar código-fonte]
Vamos considerar os registros da incidência de diabetes. O texto seguinte consta na íntegra da descrição do conjunto de dados:
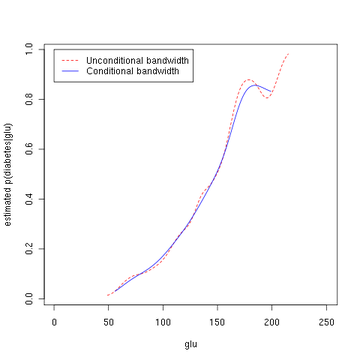
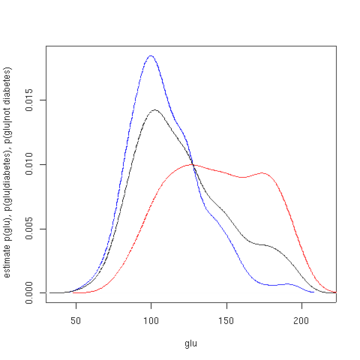
Neste exemplo, vamos construir três estimativas de densidade para "glu" (concentração plasmática de glicose), uma condicional na presença de diabetes, a segunda condicional na ausência de diabetes, e o terceiro não condicional em diabetes. A densidade condicional estimativas são então usados para construir a probabilidade de diabetes condicional "glu".
library(MASS)
data(Pima.tr)
data(Pima.te)
Pima <- rbind (Pima.tr, Pima.te)
glu <- Pima[, 'glu']
d0 <- Pima[, 'type'] == 'No'
d1 <- Pima[, 'type'] == 'Yes'
base.rate.d1 <- sum(d1) / (sum(d1) + sum(d0))
glu.density <- density (glu)
glu.d0.density <- density (glu[d0])
glu.d1.density <- density (glu[d1])
glu.d0.f <- approxfun(glu.d0.density$x, glu.d0.density$y)
glu.d1.f <- approxfun(glu.d1.density$x, glu.d1.density$y)
p.d.given.glu <- function(glu, base.rate.d1)
{
p1 <- glu.d1.f(glu) * base.rate.d1
p0 <- glu.d0.f(glu) * (1 - base.rate.d1)
p1 / (p0 + p1)
}
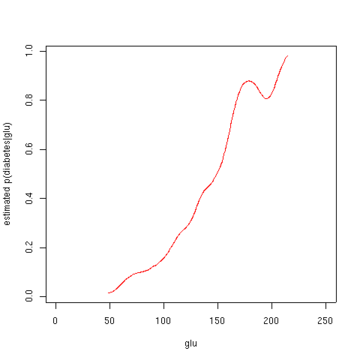
x <- 1:250
y <- p.d.given.glu (x, base.rate.d1)
plot(x, y, type='l', col='red', xlab='glu', ylab='estimated p(diabetes|glu)')
plot(density(glu[d0]), col='blue', xlab='glu', ylab='estimate p(glu),
p(glu|diabetes), p(glu|not diabetes)', main=NA)
lines(density(glu[d1]), col='red')
library(np)
fy.x <- npcdens(type~glu, nmulti=1, data=Pima)
Pima.eval <- data.frame(type=factor("Yes"),
glu=seq(min(Pima$glu), max(Pima$glu), length=250))
plot(x, y, type='l', lty=2, col='red', xlab='glu',
ylab='estimated p(diabetes|glu)')
lines(Pima.eval$glu, predict(fy.x, newdata=Pima.eval), col="blue")
legend(0, 1, c("Unconditional bandwidth", "Conditional bandwidth"),
col=c("red", "blue"), lty=c(2, 1))