Mutagênese (técnica de biologia molecular)
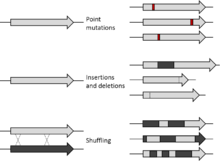
Na biologia molecular, a mutagênese é uma importante técnica laboratorial pela qual as mutações do DNA são deliberadamente projetadas para produzir bibliotecas de genes mutantes, proteínas, cepas de bactérias ou outros organismos geneticamente modificados. Os vários constituintes de um gene, bem como seus elementos reguladores e seus produtos gênicos, podem sofrer mutações para que o funcionamento de um locus, processo ou produto genético possa ser examinado em detalhes. A mutação pode produzir proteínas mutantes com propriedades interessantes ou funções aprimoradas ou novas que podem ser de uso comercial. Também podem ser produzidas cepas mutantes que têm aplicação prática ou permitem que a base molecular de uma função celular particular seja investigada.
Muitos métodos de mutagênese existem hoje. Inicialmente, o tipo de mutação induzida artificialmente em laboratório era totalmente aleatória usando mecanismos como a irradiação UV. A mutagênese aleatória não pode atingir regiões ou sequências específicas do genoma; no entanto, com o desenvolvimento da mutagênese sítio-dirigida, podem ser feitas alterações mais específicas. Desde 2013, o desenvolvimento da tecnologia CRISPR /Cas9, baseada em um sistema de defesa viral procariótico, permitiu a edição ou mutagênese de um genoma in vivo .[1] A mutagênese sítio-dirigida provou ser útil em situações que a mutagênese aleatória não é. Outras técnicas de mutagênese incluem mutagênese combinatória e por inserção. Mutagênese que não é aleatória pode ser usada para clonar DNA,[2] investigar os efeitos de agentes mutagênicos,[3] e projetar proteínas.[4] Também tem aplicações médicas, como ajudar pacientes imunocomprometidos, pesquisa e tratamento de doenças, incluindo HIV e câncer, e cura de doenças como beta talassemia .[5]
Mutagênese aleatória[editar | editar código-fonte]

As primeiras abordagens à mutagênese baseavam-se em métodos que produziam mutações inteiramente aleatórias. Em tais métodos, células ou organismos são expostos a agentes mutagênicos, como radiação UV ou produtos químicos mutagênicos, e mutantes com características desejadas são então selecionados. Hermann Muller descobriu em 1927 que os raios X podem causar mutações genéticas em drosófilas,[6] e passou a usar os mutantes que ele criou para seus estudos em genética.[7] Para Escherichia coli, os mutantes podem ser selecionados primeiro por exposição à radiação UV, depois plaqueados em um meio de ágar. As colônias formadas são então replicadas, uma em meio rico, outra em meio mínimo, e os mutantes que possuem necessidades nutricionais específicas podem então ser identificados por sua incapacidade de crescer no meio mínimo. Procedimentos semelhantes podem ser repetidos com outros tipos de células e com diferentes meios de seleção.
Vários métodos para gerar mutações aleatórias em proteínas específicas foram posteriormente desenvolvidos para rastrear mutantes com propriedades interessantes ou melhoradas. Esses métodos podem envolver o uso de nucleotídeos alterados na síntese de oligonucleotídeos ou a condução de uma reação de PCR em condições que aumentam a incorporação incorreta de nucleotídeos (PCR propensa a erros), por exemplo, reduzindo a fidelidade de replicação ou usando análogos de nucleotídeos.[8] Uma variação deste método para integrar mutações não tendenciosas em um gene é a mutagênese por saturação.[9] Os produtos de PCR que contêm mutação(ões) são então clonados num vetor de expressão e as proteínas mutantes produzidas podem então ser caracterizadas.
Em estudos com animais, agentes alquilantes como N- etil- N- nitrosoureia (ENU) foram usados para gerar camundongos mutantes.[10][11] Etil metanossulfonato (EMS) também é frequentemente usado para gerar mutantes de animais, plantas e vírus.[12][13][14]
Em uma lei da União Européia (como a diretiva 2001/18), esse tipo de mutagênese pode ser usado para produzir OGMs (organismos geneticamente modificados), mas os produtos estão isentos de regulamentação: sem rotulagem, sem avaliação.[15]
Mutagênese sítio-dirigida[editar | editar código-fonte]
Antes do desenvolvimento das técnicas de mutagênese sítio-dirigida, todas as mutações feitas eram aleatórias, e os cientistas tinham que usar seleção para o fenótipo desejado para encontrar a mutação desejada. As técnicas de mutagênese aleatória têm uma vantagem em termos de quantas mutações podem ser produzidas; no entanto, embora a mutagênese aleatória possa produzir uma mudança em nucleotídeos únicos, ela não oferece muito controle sobre qual nucleotídeo está sendo alterado.[5] Muitos pesquisadores, portanto, procuram introduzir alterações selecionadas no DNA de maneira precisa e sítio-específica. As primeiras tentativas usaram análogos de nucleotídeos e outros produtos químicos foram usados pela primeira vez para gerar mutações pontuais localizadas.[16] Tais produtos químicos incluem aminopurina, que induz uma transição de AT para GC,[17] enquanto nitrosoguanidina,[18] bissulfito,[19] e N 4 -hidroxicitidina podem induzir uma transição de GC para AT.[20][21] Essas técnicas permitem que mutações específicas sejam projetadas em uma proteína; no entanto, eles não são flexíveis em relação aos tipos de mutantes gerados, nem são tão específicos quanto os métodos posteriores de mutagênese sítio-dirigida e, portanto, têm algum grau de aleatoriedade. Com outras tecnologias, como clivagem de DNA em locais específicos do cromossomo, adição de novos nucleotídeos e troca de pares de bases, agora é possível decidir onde as mutações pode ocorrer.[11][8]

As técnicas atuais para mutação sítio-dirigida se originaram da técnica de extensão de primer desenvolvida em 1978. Tais técnicas geralmente envolvem o uso de oligonucleotídeos mutagênicos pré-fabricados em uma reação de extensão de primer com DNA polimerase. Este método permite a mutação pontual ou deleção ou inserção de pequenos trechos de DNA em locais específicos. Avanços na metodologia tornaram tal mutagênese agora um processo relativamente simples e eficiente.[3]
Métodos mais novos e mais eficientes de mutagênese sítio-dirigida estão sendo constantemente desenvolvidos. Por exemplo, a técnica SLiCE (do inglês Seamless ligation cloning extract) permite a clonagem de certas sequências de DNA dentro do genoma, e mais de um fragmento de DNA pode ser inserido no genoma de uma só vez.[2]
A mutagênese sítio-dirigida permite investigar o efeito de uma mutação específica. Existem vários usos; por exemplo, tem sido usado para determinar a suscetibilidade de certas espécies a produtos químicos que são frequentemente usados em laboratórios. O experimento usou mutagênese sítio-dirigida para imitar as mutações esperadas do produto químico específico. A mutação resultou em uma alteração em aminoácidos específicos e os efeitos dessa mutação foram analisados.[3]

A abordagem sítio-dirigida pode ser feita sistematicamente em técnicas como a mutagênese de varredura de alanina, em que os resíduos são sistematicamente mutados em alanina para identificar resíduos importantes para a estrutura ou função de uma proteína.[22] Outra abordagem abrangente é a mutagênese por saturação onde um códon ou um conjunto de códons podem ser substituídos por todos os aminoácidos possíveis nas posições específicas.[23][24]
Mutagênese combinatória[editar | editar código-fonte]
A mutagênese combinatória é uma técnica de engenharia de proteínas sítio-dirigida em que vários mutantes de uma proteína podem ser projetados simultaneamente com base na análise dos efeitos de mutações individuais aditivas.[25] Ele fornece um método útil para avaliar o efeito combinatório de um grande número de mutações na função da proteína.[26] Um grande número de mutantes pode ser rastreado para uma característica particular por análise combinatória.[25] Nesta técnica, múltiplas posições ou sequências curtas ao longo de uma fita de DNA podem ser exaustivamente modificadas para obter uma biblioteca abrangente de proteínas mutantes.[25] A taxa de incidência de variantes benéficas pode ser melhorada por diferentes métodos para construir bibliotecas de mutagênese. Uma abordagem para esta técnica é extrair e substituir uma porção da sequência de DNA por uma biblioteca de sequências contendo todas as combinações possíveis no local de mutação desejado. O conteúdo do segmento inserido pode incluir sequências com significância estrutural, propriedade imunogênica ou função enzimática. Um segmento também pode ser inserido aleatoriamente no gene para avaliar a significância estrutural ou funcional de uma parte específica de uma proteína.[25]
Mutagênese por inserção[editar | editar código-fonte]
A inserção de um ou mais pares de bases, resultando em mutações no DNA, também é conhecida como mutagênese por inserção.[27] Mutações como essas podem fornecer informações importantes na pesquisa do câncer, como insights mecânicos sobre o desenvolvimento da doença. Retrovírus e transposons são as principais ferramentas instrumentais na mutagênese por inserção. Retrovírus, como o vírus do tumor mamário do camundongo e o vírus da leucemia murina, podem ser usados para identificar genes envolvidos na carcinogênese e entender as vias biológicas de cânceres específicos.[28] Os transposons, segmentos cromossômicos que podem sofrer transposição, podem ser projetados e aplicados à mutagênese por inserção como instrumento para a descoberta de protooncogenes.[28] Esses segmentos cromossômicos permitem que a mutagênese por inserção seja aplicada a praticamente qualquer tecido de escolha, ao mesmo tempo em que permite uma profundidade mais abrangente e imparcial no sequenciamento de DNA.[28]
Pesquisadores descobriram quatro mecanismos de mutagênese por inserção que podem ser usados em humanos. O primeiro mecanismo é chamado de inserção de intensificador. Os intensificadores aumentam a transcrição de um gene específico interagindo com um promotor desse gene. Esse mecanismo em particular foi usado pela primeira vez para ajudar pacientes gravemente imunocomprometidos que precisam de medula óssea. Gamaretroviruses carregando intensificadores foram então inseridos em pacientes. O segundo mecanismo é referido como inserção do promotor. Os promotores fornecem às nossas células as sequências específicas necessárias para iniciar a tradução. A inserção do promotor ajudou os pesquisadores a aprenderem mais sobre o vírus HIV. O terceiro mecanismo é a inativação do gene. Um exemplo de inativação de genes é o uso de mutagênese por inserção para inserir um retrovírus que interrompe o genoma da célula T em pacientes com leucemia e fornece a eles um antígeno específico chamado CAR, permitindo que as células T atinjam as células cancerígenas. O mecanismo final é referido como substituição da extremidade 3' do mRNA. Nossos genes ocasionalmente sofrem mutações pontuais causando beta-talassemia que interrompe a função dos glóbulos vermelhos. Para corrigir este problema, a sequência genética correta para os glóbulos vermelhos é introduzida e uma substituição é feita.[5]
Recombinação homóloga[editar | editar código-fonte]
A recombinação homóloga pode ser usada para produzir mutações específicas em um organismo. O vetor contendo sequência de DNA semelhante ao gene a ser modificado é introduzido na célula e, por um processo de recombinação, substitui o gene alvo no cromossomo. Este método pode ser usado para introduzir uma mutação ou deletar um gene, por exemplo, na produção de camundongos knockout.[29]
CRISPR[editar | editar código-fonte]
Desde 2013, o desenvolvimento da tecnologia CRISPR- Cas9 permitiu a introdução eficiente de diferentes tipos de mutações no genoma de uma ampla variedade de organismos. O método não requer um sítio de inserção do transposon, não deixa marcadores, e sua eficiência e simplicidade o tornaram o método preferido para edição de genoma.[30][31]
Síntese de genes[editar | editar código-fonte]
À medida que o custo da síntese de oligonucleotídeos de DNA cai, a síntese artificial de um gene completo é agora um método viável para a introdução de mutações em um gene. Este método permite mutações extensas em vários locais, incluindo o redesenho completo do uso de códons de um gene para otimizá-lo para um organismo específico.[32]
Ver também[editar | editar código-fonte]
- Engenharia Genética
- OncoRato
- Mutagênese Saturada
- Evolução dirigida
Referências
- ↑ Hsu PD, Lander ES, Zhang F (junho de 2014). «Development and applications of CRISPR-Cas9 for genome engineering». Cell. 157 (6): 1262–78. PMC 4343198
 . PMID 24906146. doi:10.1016/j.cell.2014.05.010
. PMID 24906146. doi:10.1016/j.cell.2014.05.010
- ↑ a b Motohashi K (junho de 2015). «A simple and efficient seamless DNA cloning method using SLiCE from Escherichia coli laboratory strains and its application to SLiP site-directed mutagenesis». BMC Biotechnology. 15. 47 páginas. PMC 4453199
 . PMID 26037246. doi:10.1186/s12896-015-0162-8
. PMID 26037246. doi:10.1186/s12896-015-0162-8
- ↑ a b c Doering JA, Lee S, Kristiansen K, Evenseth L, Barron MG, Sylte I, LaLone CA (novembro de 2018). «In Silico Site-Directed Mutagenesis Informs Species-Specific Predictions of Chemical Susceptibility Derived From the Sequence Alignment to Predict Across Species Susceptibility (SeqAPASS) Tool». Toxicological Sciences. 166 (1): 131–145. PMC 6390969
 . PMID 30060110. doi:10.1093/toxsci/kfy186
. PMID 30060110. doi:10.1093/toxsci/kfy186
- ↑ Choi GC, Zhou P, Yuen CT, Chan BK, Xu F, Bao S, et al. (agosto de 2019). «Combinatorial mutagenesis en masse optimizes the genome editing activities of SpCas9». Nature Methods. 16 (8): 722–730. PMID 31308554. doi:10.1038/s41592-019-0473-0
- ↑ a b c Bushman FD (fevereiro de 2020). «Retroviral Insertional Mutagenesis in Humans: Evidence for Four Genetic Mechanisms Promoting Expansion of Cell Clones». Molecular Therapy (em inglês). 28 (2): 352–356. PMC 7001082
 . PMID 31951833. doi:10.1016/j.ymthe.2019.12.009
. PMID 31951833. doi:10.1016/j.ymthe.2019.12.009
- ↑ Muller HJ (julho de 1927). «Artificial Transmutation of the Gene» (PDF). Science. 66 (1699): 84–7. Bibcode:1927Sci....66...84M. PMID 17802387. doi:10.1126/science.66.1699.84
- ↑ Crow JF, Abrahamson S (dezembro de 1997). «Seventy years ago: mutation becomes experimental». Genetics. 147 (4): 1491–6. PMC 1208325
 . PMID 9409815. doi:10.1093/genetics/147.4.1491
. PMID 9409815. doi:10.1093/genetics/147.4.1491
- ↑ a b Nucleic Acids in Chemistry and Biology 3rd ed. [S.l.]: Royal Society of Chemistry. 2006. pp. 191–192. ISBN 978-0854046546
- ↑ Wong TS, Tee KL, Hauer B, Schwaneberg U (fevereiro de 2004). «Sequence saturation mutagenesis (SeSaM): a novel method for directed evolution». Nucleic Acids Research. 32 (3): 26e–26. PMC 373423
 . PMID 14872057. doi:10.1093/nar/gnh028
. PMID 14872057. doi:10.1093/nar/gnh028
- ↑ Justice MJ, Noveroske JK, Weber JS, Zheng B, Bradley A (1999). «Mouse ENU mutagenesis». Human Molecular Genetics. 8 (10): 1955–63. PMID 10469849. doi:10.1093/hmg/8.10.1955

- ↑ a b Hrabé de Angelis M, Balling R (maio de 1998). «Large scale ENU screens in the mouse: genetics meets genomics». Mutation Research. 400 (1–2): 25–32. PMID 9685575. doi:10.1016/s0027-5107(98)00061-x
- ↑ Flibotte S, Edgley ML, Chaudhry I, Taylor J, Neil SE, Rogula A, et al. (junho de 2010). «Whole-genome profiling of mutagenesis in Caenorhabditis elegans». Genetics. 185 (2): 431–41. PMC 2881127
 . PMID 20439774. doi:10.1534/genetics.110.116616
. PMID 20439774. doi:10.1534/genetics.110.116616
- ↑ EMS screens : from mutagenesis to screening and mapping. Col: Methods in Molecular Biology. 420. [S.l.: s.n.] 2008. pp. 119–38. PMID 18641944. doi:10.1007/978-1-59745-583-1_7
- ↑ Favor AH, Llanos CD, Youngblut MD, Bardales JA (2020). «Optimizing bacteriophage engineering through an accelerated evolution platform». Scientific Reports. 10 (1): 13981. PMC 7438504
 . PMID 32814789. doi:10.1038/s41598-020-70841-1
. PMID 32814789. doi:10.1038/s41598-020-70841-1
- ↑ «GMO directive : the origins of the mutagenesis exemption». Inf'OGM. Março de 2018
- ↑ Shortle D, DiMaio D, Nathans D (1981). «Directed mutagenesis». Annual Review of Genetics. 15: 265–94. PMID 6279018. doi:10.1146/annurev.ge.15.120181.001405
- ↑ Caras IW, MacInnes MA, Persing DH, Coffino P, Martin DW (setembro de 1982). «Mechanism of 2-aminopurine mutagenesis in mouse T-lymphosarcoma cells». Molecular and Cellular Biology. 2 (9): 1096–103. PMC 369902
 . PMID 6983647. doi:10.1128/mcb.2.9.1096
. PMID 6983647. doi:10.1128/mcb.2.9.1096
- ↑ McHugh GL, Miller CG (outubro de 1974). «Isolation and characterization of proline peptidase mutants of Salmonella typhimurium». Journal of Bacteriology. 120 (1): 364–71. PMC 245771
 . PMID 4607625. doi:10.1128/JB.120.1.364-371.1974
. PMID 4607625. doi:10.1128/JB.120.1.364-371.1974
- ↑ Shortle D, Nathans D (maio de 1978). «Local mutagenesis: a method for generating viral mutants with base substitutions in preselected regions of the viral genome». Proceedings of the National Academy of Sciences of the United States of America. 75 (5): 2170–4. Bibcode:1978PNAS...75.2170S. PMC 392513
 . PMID 209457. doi:10.1073/pnas.75.5.2170
. PMID 209457. doi:10.1073/pnas.75.5.2170
- ↑ Flavell RA, Sabo DL, Bandle EF, Weissmann C (janeiro de 1975). «Site-directed mutagenesis: effect of an extracistronic mutation on the in vitro propagation of bacteriophage Qbeta RNA». Proceedings of the National Academy of Sciences of the United States of America. 72 (1): 367–71. Bibcode:1975PNAS...72..367F. PMC 432306
 . PMID 47176. doi:10.1073/pnas.72.1.367
. PMID 47176. doi:10.1073/pnas.72.1.367
- ↑ Müller W, Weber H, Meyer F, Weissmann C (setembro de 1978). «Site-directed mutagenesis in DNA: generation of point mutations in cloned beta globin complementary dna at the positions corresponding to amino acids 121 to 123». Journal of Molecular Biology. 124 (2): 343–58. PMID 712841. doi:10.1016/0022-2836(78)90303-0
- ↑ Vanessa E. Gray; Ronald J. Hause; Douglas M. Fowler (1 de setembro de 2017). «Analysis of Large-Scale Mutagenesis Data To Assess the Impact of Single Amino Acid Substitutions». Genetics. 207 (1): 53–61. PMC 5586385
 . PMID 28751422. doi:10.1534/genetics.117.300064
. PMID 28751422. doi:10.1534/genetics.117.300064
- ↑ Reetz, M. T.; Carballeira J. D. (2007). «Iterative saturation mutagenesis (ISM) for rapid directed evolution of functional enzymes». Nature Protocols. 2 (4): 891–903. PMID 17446890. doi:10.1038/nprot.2007.72
- ↑ Cerchione, Derek; Loveluck, Katherine; Tillotson, Eric L.; Harbinski, Fred; DaSilva, Jen; Kelley, Chase P.; Keston-Smith, Elise; Fernandez, Cecilia A.; Myer, Vic E. (16 de abril de 2020). «SMOOT libraries and phage-induced directed evolution of Cas9 to engineer reduced off-target activity». PLOS ONE. 15 (4): e0231716. Bibcode:2020PLoSO..1531716C. PMC 7161989
 . PMID 32298334. doi:10.1371/journal.pone.0231716
. PMID 32298334. doi:10.1371/journal.pone.0231716
- ↑ a b c d Parker AS, Griswold KE, Bailey-Kellogg C (novembro de 2011). «Optimization of combinatorial mutagenesis». Journal of Computational Biology. 18 (11): 1743–56. Bibcode:2011LNCS.6577..321P. PMC 5220575
 . PMID 21923411. doi:10.1089/cmb.2011.0152
. PMID 21923411. doi:10.1089/cmb.2011.0152
- ↑ Choi GC, Zhou P, Yuen CT, Chan BK, Xu F, Bao S, Chu HY, Thean D, Tan K, Wong KH, Zheng Z, Wong AS (agosto de 2019). «Combinatorial mutagenesis en masse optimizes the genome editing activities of SpCas9». Nature Methods. 16 (8): 722–730. PMID 31308554. doi:10.1038/s41592-019-0473-0
- ↑ Uren AG, Kool J, Berns A, van Lohuizen M (novembro de 2005). «Retroviral insertional mutagenesis: past, present and future». Oncogene. 24 (52): 7656–72. PMID 16299527. doi:10.1038/sj.onc.1209043

- ↑ a b c Vassiliou G, Rad R, Bradley A (1 de janeiro de 2010). «The use of DNA transposons for cancer gene discovery in mice» 2nd ed. Academic Press. Methods in Enzymology. Guide to Techniques in Mouse Development, Part B: Mouse Molecular Genetics. 477: 91–106. ISBN 9780123848802. PMID 20699138. doi:10.1016/s0076-6879(10)77006-3
- ↑ «Homologous Recombination Method (and Knockout Mouse)». Davidson College
- ↑ Damien Biot-Pelletier; Vincent J. J. Martin (2016). «Seamless site-directed mutagenesis of the Saccharomyces cerevisiae genome using CRISPR-Cas9». Journal of Biological Engineering. 10: 6. PMC 4850645
 . PMID 27134651. doi:10.1186/s13036-016-0028-1
. PMID 27134651. doi:10.1186/s13036-016-0028-1
- ↑ Xu S (20 de agosto de 2015). «The application of CRISPR-Cas9 genome editing in Caenorhabditis elegans». J Genet Genomics. 42 (8): 413–21. PMC 4560834
 . PMID 26336798. doi:10.1016/j.jgg.2015.06.005
. PMID 26336798. doi:10.1016/j.jgg.2015.06.005
- ↑ Artificial DNA: Methods and Applications. [S.l.]: CRC Press. 25 de setembro de 2002. ISBN 9781420040166
