Intrão: diferenças entre revisões
Etiquetas: Edição via dispositivo móvel Edição feita através do sítio móvel Edição móvel avançada |
|||
| Linha 1: | Linha 1: | ||
[[Ficheiro:Gene.png|miniaturadaimagem|Representação esquemática da sequência exon-íntron em um gene, aqui representado por um cromossomo.]] |
|||
Os {{PEPB|intrões|íntrons}} (do inglês, "''intragenic regions''")<ref>LOPES, Sônia. ''Bio - Volume único''. Editora Saraiva, 2004.</ref>, '''sequências intervenientes''' ou '''IVS''' (do inglês "'''''i'''nter'''v'''ening '''s'''equence''") são secções de [[DNA]] de um [[gene]] que não codificam qualquer parte da [[proteína]] produzida pelo gene e que separa da sequência constituída pelos [[exão|exões]]. Podem ser considerados como parte do '''ADN-lixo'''.<ref group=Nota>Embora as regiões não codificantes tenham sido chamadas de '''Junk DNA''' uma vez que não são expressas, pesquisadores tem descoberto que muitas delas regulam a expressão de outras seções do ADN (em {{citar livro|autor=Cohen, Jon|título=Almost Chimpanzee|subtítulo=Searching for What Makes us Human, in Rainforests, Labs, Sanctuaries, and Zoos|editora=Times Books|local=New York|idioma=inglês|ano=2010|página=24-26|páginas=369|isbn=978-0-8050-8307-1}})</ref> O intrão é inicialmente transcrito na molécula de pré-[[RNAm]] mas, depois, é eliminado durante o processo de excisão (ou ''[[splicing]]'') do RNA pelos spliceossomas, antes da saída deste do núcleo celular. Os intrões existem principalmente, mas não exclusivamente nas [[célula]]s [[eucarioto|eucarióticas]]. Os exões permanecerão na molécula de [[RNA]] maduro. |
|||
{{PEPB|Íntrões|Íntrons}} são secções de [[DNA]] localizadas em um [[gene]] que são removidas por ''[[splicing]]'' de [[RNA]] durante a produção do [[mRNA]] ou de outro RNA funcional. São também chamados de '''sequências intervenientes''' (IVS) ou '''sequências interpostas'''.<ref>{{citar web|URL=https://decs.bvsalud.org/ths/resource/?id=23323&filter=ths_termall&q=intron|título=Descritores em Ciências da Saúde: DeCS|data=2017|local=São Paulo|publicado=BIREME/OPAS/OMS|acessodata=2020-10-21}}</ref> Em outras palavras, os íntrões são regiões não-codificantes que são eliminadas por ''splicing'' antes da [[Tradução (genética)|tradução]].<ref>{{citar periódico|data=2017|doi=10.1007/978-3-319-47829-6_70-1|editor= Vonk, Jennifer|editor2= Shackelford, Todd|autor1=Ghosh, Shampa|autor2= Sinha, Jitendra Kumar|isbn=978-3-319-47829-6|jornal=Encyclopedia of Animal Cognition and Behavior|lingua=en|paginas=1–5|publicado=Springer International Publishing|titulo=Intron}}</ref><ref>{{citar web|URL=https://biologydictionary.net/intron/|título=Intron|autor=Editors B. D.|data=|publicado=2017-08-06|website=Biology Dictionary|lingua=en-US}}</ref> A palavra ''íntrão'' é derivada do termo ''região intragênica'' (do inglês, "''intragenic regions''")<ref>{{Citar livro|autor=Lopes, Sônia|titulo=Bio: volume único|editora=Editora Saraiva|ano=2004|isbn=8502047965}}</ref>, ou seja, corresponde a uma região dentro de um gene. O termo íntrão refere-se tanto à sequência de DNA dentro de um gene, quanto à sequência correspondente em transcritos de RNA. As sequências que são unidas no RNA maduro final após o ''splicing'' do RNA são [[exão|exões]].<ref name="Genes">{{citar livro|título=Genes|autor=Lewin Benjamin|data=1987|editora=|páginas=159–179, 386|isbn=0-471-83278-2|oclc=14069165|edicao=3|local=New York}}</ref> |
|||
Inicialmente, os intrões eram considerados parte do [[ADN-lixo|DNA-lixo]], por serem regiões não-codificantes de proteínas; no entanto, pesquisas posteriores demonstraram o papel dos intrões na regulação da expressão gênica<ref>{{citar livro|autor=Cohen, Jon|título=Almost Chimpanzee|subtítulo=Searching for What Makes us Human, in Rainforests, Labs, Sanctuaries, and Zoos|editora=Times Books|local=New York|idioma=inglês|ano=2010|página=24-26|páginas=369|isbn=978-0-8050-8307-1}})</ref> e também em um processo chamado ''[[splicing alternativo]]''. Os intrões são encontrados nos genes da maioria dos organismos e de muitos vírus, e podem estar localizados em uma ampla gama de genes, incluindo aqueles que produzem [[proteínas]], [[RNA ribossômico]] (RNAr) e [[RNA de transferência]] (RNAt). Quando as proteínas são geradas a partir de genes contendo intrões, o [[Splicing|splicing do RNA]] ocorre como parte da via de processamento do RNA que segue a transcrição e precede a tradução.<ref name="Genes"/> |
|||
Os intrões permitem que a célula realize um processo denominado ''[[splicing alternativo]]'', onde formas protéicas diferentes podem ser produzidas a partir de um mesmo RNAm, ou [[RNA mensageiro]]. O intrão sai do RNA Mensageiro o que possibilita assim à mesma que o RNA Ribossómico faça a tradução génica ficando só os exões |
|||
== |
==Descoberta== |
||
Em 1977, Richard Roberts e Philip Sharp, trabalhando de forma independente, durante os estudos de replicação de [[adenovírus]] em cultura de células humanas, demonstraram que os genes poderiam ser descontínuos.<ref name="Voet">{{Citar livro| editora = Artmed Editora| isbn = 978-85-8271-066-1| ultimo1 = Voet| primeiro1 = D.| ultimo2 = Voet| primeiro2 = J.G.| ultimo3 = Pratt| primeiro3 = C.W.| titulo = Fundamentos de Bioquímica - 4.ed.: A Vida em Nível Molecular| data = 2014}}</ref> Em 1978, o bioquímico estadunidense Walter Gilbert cunhou o termo ''intron'' para se referir às sequências não codificantes do DNA.<ref name="Nature">{{Citar periódico| doi = 10.1038/271501a0| issn = 0028-0836| volume = 271| numero = 5645| paginas = 501–501| ultimo = Gilbert| primeiro = Walter| titulo = Why genes in pieces?| publicado = Nature| acessodata = 2020-10-21| data = 1978-02| url = http://www.nature.com/articles/271501a0}}</ref> |
|||
<references group="Nota"/> |
|||
==Distribuição== |
|||
A frequência de intrões em diferentes genomas varia consideravelmente em todo o espectro de organismos biológicos. Por exemplo, intrões são muito comuns dentro do genoma nuclear de vertebrados com mandíbula (como humanos e ratos), onde os genes codificadores de proteínas quase sempre contêm vários intrões. Por outro lado, os intrões são raros dentro dos genes nucleares de alguns microrganismos eucarióticos, por exemplo, a levedura (''[[Saccharomyces cerevisiae]]'').<ref name="Stajich">{{Citar periódico| doi = 10.1186/gb-2007-8-10-r223| issn = 1465-6906| volume = 8| numero = 10| paginas = –223| ultimo1 = Stajich| primeiro1 = Jason E| ultimo2 = Dietrich| primeiro2 = Fred S| ultimo3 = Roy| primeiro3 = Scott W| titulo = Comparative genomic analysis of fungal genomes reveals intron-rich ancestors| publicado = Genome Biology| acessodata = 2020-10-22| data = 2007| url = http://genomebiology.biomedcentral.com/articles/10.1186/gb-2007-8-10-r223}}</ref> Em contraste, os genomas mitocondriais de vertebrados são totalmente desprovidos de intrões, enquanto os de microrganismos eucarióticos podem conter muitos intrões.<ref name="BBA">{{Citar periódico| doi = 10.1016/S0005-2728(98)00161-3| issn = 00052728| volume = 1410| numero = 2| paginas = 103–123| ultimo = Taanman| primeiro = Jan-Willem| titulo = The mitochondrial genome: structure, transcription, translation and replication| publicado = Biochimica et Biophysica Acta (BBA) - Bioenergetics| acessodata = 2020-10-22| data = 1999-02| url = https://linkinghub.elsevier.com/retrieve/pii/S0005272898001613}}</ref> |
|||
O gene ''Drosophila'' Dhc7 contém um intrão de mais de 3,6 Mb, que leva três dias para ser transcrito.<ref name="Marches">{{Citar periódico| doi = 10.1016/S0092-8674(00)00174-4| issn = 00928674| volume = 103| numero = 5| paginas = 703–709| ultimo1 = Tollervey| primeiro1 = David| ultimo2 = Caceres| primeiro2 = Javier F| titulo = RNA Processing Marches on| publicado = Cell| acessodata = 2020-10-22| data = 2000-11| url = https://linkinghub.elsevier.com/retrieve/pii/S0092867400001744}}</ref><ref name="mega">{{Citar periódico| issn = 0016-6731| volume = 154| numero = 2| paginas = 759–769| ultimo1 = Reugels| primeiro1 = Alexander M.| ultimo2 = Kurek| primeiro2 = Roman| ultimo3 = Lammermann| primeiro3 = Ulrich| ultimo4 = Bünemann| primeiro4 = Hans| titulo = Mega-introns in the Dynein Gene DhDhc7(Y) on the Heterochromatic Y Chromosome Give Rise to the Giant Threads Loops in Primary Spermatocytes of Drosophila hydei| publicado = Genetics| data = 2000}}</ref> Em contraste, o menor comprimento de um intrão eucariótico conhecido possui 30 pares de bases, pertencendo ao gene humano ''MST1L''.<ref name="pequeno">{{Citar periódico| doi = 10.1093/dnares/dsv028| issn = 1340-2838| volume = 22| numero = 6| paginas = 495–503| ultimo1 = Piovesan| primeiro1 = Allison| ultimo2 = Caracausi| primeiro2 = Maria| ultimo3 = Ricci| primeiro3 = Marco| ultimo4 = Strippoli| primeiro4 = Pierluigi| ultimo5 = Vitale| primeiro5 = Lorenza| ultimo6 = Pelleri| primeiro6 = Maria Chiara| titulo = Identification of minimal eukaryotic introns through GeneBase, a user-friendly tool for parsing the NCBI Gene databank| publicado = DNA Research| acessodata = 2020-10-22| data = 2015-12| url = https://academic.oup.com/dnaresearch/article-lookup/doi/10.1093/dnares/dsv028}}</ref> |
|||
==Classificação== |
|||
Existem pelo menos quatro classes principais de intrões:<ref name="natural">{{Citar periódico| volume = 21| numero = 2| paginas = 111–119| ultimo1 = Haugen| primeiro1 = Peik| ultimo2 = Simon| primeiro2 = Dawn M| ultimo3 = Bhattacharya| primeiro3 = Debashish| titulo = The natural history of group I introns| publicado = TRENDS in Genetics| data = 2005}}</ref><ref name="alberts">{{Citar livro| publicado = ARTMED| isbn = 978-85-8271-422-5| ultimo1 = Alberts| primeiro1 = B.| titulo = Biologia Molecular Da Célula| data = 2017}}</ref> |
|||
*Íntrões ''auto-splicing'' do grupo I que são removidos por catálise de RNA |
|||
*Íntrões ''auto-splicing'' do grupo II que são removidos por catálise de RNA |
|||
*Íntrões em genes de RNA de transferência nuclear e de arqueias que são removidos por proteínas (intrões de RNAt) |
|||
*Íntrões em genes que codificam proteínas nucleares que são removidos por spliceossomos (intrões spliceossômicos) |
|||
Os intrões do grupo III são propostos como uma quinta família, mas pouco se sabe sobre o aparato bioquímico que medeia seu ''[[splicing]]''. Eles parecem estar relacionados aos intrões do grupo II e, possivelmente, aos intrões spliceossômicos. |
|||
===Íntrões do grupo I e II=== |
|||
intrões do grupo I e II são encontrados em genes codificadores de proteínas (RNA mensageiro), RNA de transferência e RNA ribossômico, em uma gama muito ampla entre os organismos vivos.<ref name="label"> {{Citar periódico| doi = 10.1146/annurev.bi.59.070190.002551| issn = 0066-4154| volume = 59| paginas = 543–568| ultimo = Cech| primeiro = T. R.| titulo = Self-splicing of group I introns| publicado = Annual Review of Biochemistry| data = 1990| pmid = 2197983}}</ref> Após a transcrição em RNA, os íntrons do grupo I e do grupo II também realizam extensas interações internas que permitem que eles se dobrem em uma arquitetura tridimensional complexa e específica. Essas arquiteturas complexas permitem que alguns intrões do grupo I e do grupo II sejam ''auto-splicing'', ou seja, a molécula de RNA que contém o intrões pode reorganizar sua própria estrutura covalente de modo a remover com precisão o intrões e ligar os exons na ordem correta. Em alguns casos, proteínas de ligação a intrões específicas estão envolvidas no ''splicing'', auxiliando o intron a dobrar-se na estrutura tridimensional necessária para a atividade de ''self-splicing''. Os intrões do grupo I e do grupo II são diferenciados por diferentes conjuntos de sequências conservadas internas e estruturas dobradas, e pelo fato de que o ''splicing'' de moléculas de RNA contendo íntrons do grupo II gera íntrons ramificados (como aqueles de RNAs spliceossômicos), enquanto os íntrons do grupo I usam um nucleotídeo de guanosina não codificado (tipicamente GTP) para iniciar o ''splicing'', adicionando-o à extremidade 5' do intrões excisado. |
|||
===Íntrões de RNAt=== |
|||
Os intrões de RNA de transferência que dependem de proteínas para remoção ocorrem em um local específico dentro da alça anticódon de precursores de tRNA não duplicados e são removidos por uma endonuclease de splicing de tRNA. Os exons são então ligados por uma segunda proteína, a ligase de splicing do tRNA.<ref name="Greer">{{Citar periódico| doi = 10.1016/0092-8674(83)90473-7| issn = 0092-8674| volume = 32| numero = 2| paginas = 537–546| ultimo1 = Greer| primeiro1 = C. L.| ultimo2 = Peebles| primeiro2 = C. L.| ultimo3 = Gegenheimer| primeiro3 = P.| ultimo4 = Abelson| primeiro4 = J.| titulo = Mechanism of action of a yeast RNA ligase in tRNA splicing| publicado = Cell| data = 1983-02| pmid = 6297798}}</ref> Observe que os intrões autossplicáveis às vezes também são encontrados nos genes de RNAt.<ref name="Self"> {{Citar periódico| doi = 10.1038/357173a0| issn = 0028-0836| volume = 357| numero = 6374| paginas = 173–176| ultimo1 = Reinhold-Hurek| primeiro1 = B.| ultimo2 = Shub| primeiro2 = D. A.| titulo = Self-splicing introns in tRNA genes of widely divergent bacteria| publicado = Nature| data = 1992-05-14| pmid = 1579169}}</ref> |
|||
===Íntrões spliceossômicos=== |
|||
Os intrões do pré-RNAm nuclear (intrões spliceossômicos) são caracterizados por sequências de intrões específicas localizadas nos limites entre os intrões e os exons.<ref name="precursors">{{Citar periódico| doi = 10.1146/annurev.bi.55.070186.005351| issn = 0066-4154| volume = 55| paginas = 1119–1150| ultimo1 = Padgett| primeiro1 = R. A.| ultimo2 = Grabowski| primeiro2 = P. J.| ultimo3 = Konarska| primeiro3 = M. M.| ultimo4 = Seiler| primeiro4 = S.| ultimo5 = Sharp| primeiro5 = P. A.| titulo = Splicing of messenger RNA precursors| publicado = Annual Review of Biochemistry| data = 1986| pmid = 2943217}}</ref> Essas sequências são reconhecidas por moléculas de RNA spliceosomal quando as reações de splicing são iniciadas.<ref name="SNRNAS">{{Citar periódico| doi = 10.1146/annurev.ge.22.120188.002131| issn = 0066-4197| volume = 22| numero = 1| paginas = 387–419| ultimo1 = Guthrie| primeiro1 = C| ultimo2 = Patterson| primeiro2 = B| titulo = Spliceosomal SNRNAS| publicado = Annual Review of Genetics| acessodata = 2020-10-22| data = 1988-12| url = http://www.annualreviews.org/doi/10.1146/annurev.ge.22.120188.002131}}</ref> Além disso, eles contêm um ponto de ramificação, uma sequência de nucleotídeos particular perto da extremidade 3' do intrão, que se liga covalentemente à extremidade 5' do intrão durante o processo de ''splicing'', gerando um intron ramificado (''lariat''). Além desses três elementos curtos conservados, as sequências do intrão do pré-RNAm nuclear são altamente variáveis. Os íntrons pré-RNAm nucleares são frequentemente muito mais longos do que os exons circundantes. |
|||
==Funções biológicas e evolução== |
|||
Embora os intrões não codifiquem produtos proteicos, eles são essenciais para a regulação da expressão gênica. Alguns intrões codificam RNAs funcionais por meio de processamento posterior após o ''splicing'' para gerar moléculas de [[RNA não-codificante]].<ref>{{citar periódico |autores=Rearick D, Prakash A, McSweeny A, Shepard SS, Fedorova L, Fedorov A |titulo=Critical association of ncRNA with introns |journal=Nucleic Acids Res. |volume=39 |numero=6 |paginas=2357–66 |data=março 2011 |pmid=21071396 |pmc=3064772 |doi=10.1093/nar/gkq1080 |url=}}</ref> O [[splicing alternativo]] é amplamente usado para gerar várias proteínas de um único gene. Além disso, alguns intrões desempenham papéis essenciais em uma ampla gama de funções regulatórias de expressão gênica.<ref>{{citar periódico|pmid=23108796|doi= 10.1002/bies.201200073|volume=34|numero=12|titulo= Introns in UTRs: why we should stop ignoring them. |data=dezembro 2012|paginas=1025–34|autores=Bicknell AA, Cenik C, Chua HN, Roth FP, Moore MJ|journal=BioEssays}}</ref><ref name="Cenik2011">{{citar periódico|last1=Cenik|first1=Can|last2=Chua|first2=Hon Nian|last3=Zhang|first3=Hui|last4=Tarnawsky|first4=Stefan P.|last5=Akef|first5=Abdalla|last6=Derti|first6=Adnan|last7=Tasan|first7=Murat|last8=Moore|first8=Melissa J.|last9=Palazzo|first9=Alexander F.|last10=Roth|first10=Frederick P.|titulo=Genome Analysis Reveals Interplay between 5′UTR Introns and Nuclear mRNA Export for Secretory and Mitochondrial Genes|journal=PLOS Genetics|volume=7|numero=4|ano=2011|paginas=e1001366|issn=1553-7404|doi=10.1371/journal.pgen.1001366|editor1-last=Snyder|editor1-first=Michael|pmid=21533221|pmc=3077370}}</ref> |
|||
As origens biológicas dos intrões são obscuras. Após a descoberta inicial de intrões em genes codificadores de proteínas do núcleo eucariótico, houve um debate significativo sobre a origem dos intrões. A primeira hipótese afirma que os intrões foram herdados de um ancestral em comum entre os eucariotos e procariotos, e teria sido posteriormente perdido em procariotos. A segunda hipótese assume que os intrões teriam surgido recentemente no processo evolutivo. Outra teoria é que o [[spliceossomo]] e a estrutura íntron-exon dos genes é uma relíquia do [[Hipótese do mundo de ARN|mundo de RNA]].<ref>{{citar periódico|pmid=19777149|doi=10.1007/s00239-009-9279-5|volume=69|numero=5|titulo=An overview of the introns-first theory |data=novembro 2009|paginas=527–40 |autores=Penny D, Hoeppner MP, Poole AM, Jeffares DC|journal=Journal of Molecular Evolution| bibcode=2009JMolE..69..527P}}</ref> Ainda há um debate considerável sobre até que ponto essas hipóteses são mais corretas. O consenso popular no momento é que os intrões surgiram dentro da linhagem dos eucariotos como elementos egoístas.<ref>{{citar periódico|last1=Cavalier-Smith|first1=T|titulo=Selfish DNA and the origin of introns.|journal=Nature|data=1985|volume=315|numero=6017|paginas=283–4|pmid=2987701|doi=10.1038/315283b0|bibcode=1985Natur.315..283C}}</ref> |
|||
<!-- |
|||
Early studies of genomic DNA sequences from a wide range of organisms show that the intron-exon structure of homologous genes in different organisms can vary widely.<ref>{{citar periódico |autores=Rodríguez-Trelles F, Tarrío R, Ayala FJ|ano=2006|titulo=Origins and evolution of spliceosomal introns|url =|journal=Annu. Rev. Genet.|volume=40|numero =|paginas=47–76|doi=10.1146/annurev.genet.40.110405.090625|pmid=17094737 }}</ref> More recent studies of entire [[eukaryote|eukaryotic]] [[genome]]s have now shown that the lengths and density (introns/gene) of introns varies considerably between related species. For example, while the human genome contains an average of 8.4 introns/gene (139,418 in the genome), the unicellular fungus ''[[Encephalitozoon cuniculi]]'' contains only 0.0075 introns/gene (15 introns in the genome).<ref>{{citar periódico|pmid=12775832|doi=10.1126/science.1080559|volume=300|numero=5624|titulo=Eukaryotic intron loss |data=maio 2003|paginas=1393 |autores=Mourier T, Jeffares DC|journal=Science}}</ref> Since eukaryotes arose from a common ancestor ([[common descent]]), there must have been extensive gain or loss of introns during evolutionary time.<ref>{{citar periódico|pmid=16485020|doi=10.1038/nrg1807|volume=7|numero=3|titulo=The evolution of spliceosomal introns: patterns, puzzles and progress |data=março 2006|paginas=211–21 |autores=Roy SW, Gilbert W|journal=Nature Reviews Genetics }}</ref><ref>{{citar periódico|pmid=12868602|volume=118|numero=2–3|titulo=The emergence of a synthetic theory of intron evolution |data=julho 2003|paginas=117–21|author=de Souza SJ|journal=Genetica|doi=10.1023/A:1024193323397}}</ref> This process is thought to be subject to selection, with a tendency towards intron gain in larger species due to their smaller population sizes, and the converse in smaller (particularly unicellular) species.<ref>{{citar periódico|pmid=11983904|doi=10.1073/pnas.092595699|volume=99|numero=9|titulo=Intron evolution as a population-genetic process|pmc=122912 |data=abril 2002|paginas=6118–23|author=Lynch M|journal=Proceedings of the National Academy of Sciences| bibcode=2002PNAS...99.6118L }}</ref> Biological factors also influence which genes in a genome lose or accumulate introns.<ref>{{citar periódico|pmid=16290250|doi=10.1016/j.tig.2005.10.006|volume=22|numero=1|titulo=The biology of intron gain and loss |data=janeiro 2006|paginas=16–22 |autores=Jeffares DC, Mourier T, Penny D|journal=Trends in Genetics}}</ref><ref>{{citar periódico|pmid=18586348|doi=10.1016/j.tig.2008.05.006|volume=24|numero=8|titulo=Rapidly regulated genes are intron poor |data=agosto 2008|paginas=375–8|autores=Jeffares DC, Penkett CJ, Bähler J|journal=Trends in Genetics}}</ref><ref>{{citar periódico|pmid=12134150|doi=10.1038/ng940|volume=31|numero=4|titulo=Selection for short introns in highly expressed genes |data=agosto 2002|paginas=415–8 |autores=Castillo-Davis CI, Mekhedov SL, Hartl DL, Koonin EV, Kondrashov FA|journal=Nature Genetics| url=https://zenodo.org/record/1233421 }}</ref> |
|||
[[Alternative splicing]] of exons within a gene after intron excision acts to introduce greater variability of protein sequences translated from a single gene, allowing multiple related proteins to be generated from a single gene and a single precursor mRNA transcript. The control of alternative RNA splicing is performed by a complex network of signaling molecules that respond to a wide range of intracellular and extracellular signals. |
|||
Introns contain several short sequences that are important for efficient splicing, such as acceptor and donor sites at either end of the intron as well as a branch point site, which are required for proper splicing by the [[spliceosome]]. Some introns are known to enhance the expression of the gene that they are contained in by a process known as [[Intron Mediated Enhancement|intron-mediated enhancement]] (IME). |
|||
Actively transcribed regions of [[DNA]] frequently form [[R-loop]]s that are vulnerable to [[DNA damage (naturally occurring)|DNA damage]]. In highly expressed yeast genes, introns inhibit R-loop formation and the occurrence of DNA damage.<ref name="pmid28757210">{{citar periódico |autores=Bonnet A, Grosso AR, Elkaoutari A, Coleno E, Presle A, Sridhara SC, Janbon G, Géli V, de Almeida SF, Palancade B |titulo=Introns Protect Eukaryotic Genomes from Transcription-Associated Genetic Instability |journal=Mol. Cell |volume=67 |numero=4 |paginas=608–621.e6 |ano=2017 |pmid=28757210 |doi=10.1016/j.molcel.2017.07.002 |url=|doi-access=free }}</ref> Genome-wide analysis in both yeast and humans revealed that intron-containing genes have decreased R-loop levels and decreased DNA damage compared to intronless genes of similar expression.<ref name="pmid28757210" /> Insertion of an intron within an R-loop prone gene can also suppress R-loop formation and [[genetic recombination|recombination]]. Bonnet et al. (2017)<ref name="pmid28757210" /> speculated that the function of introns in maintaining genetic stability maio explain their evolutionary maintenance at certain locations, particularly in highly expressed genes. |
|||
=== Starvation adaptation === |
|||
The physical presence of introns promotes cellular resistance to starvation via intron enhanced repression of ribosomal protein genes of nutrient-sensing pathways.<ref>{{citar periódico|last1=Parenteau|first1=Julie|last2=Maignon|first2=Laurine|last3=Berthoumieux|first3=Mélodie|last4=Catala|first4=Mathieu|last5=Gagnon|first5=Vanessa|last6=Abou Elela|first6=Sherif|data=2019-01-16|titulo=Introns are mediators of cell response to starvation|journal=Nature|volume=565|numero=7741|paginas=612–617|doi=10.1038/s41586-018-0859-7|issn=1476-4687|pmid=30651641|bibcode=2019Natur.565..612P}}</ref> |
|||
--> |
|||
{{referências}} |
{{referências}} |
||
Revisão das 02h23min de 22 de outubro de 2020
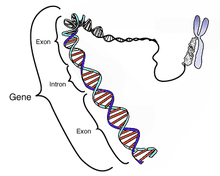
Íntrões (português europeu) ou Íntrons (português brasileiro) são secções de DNA localizadas em um gene que são removidas por splicing de RNA durante a produção do mRNA ou de outro RNA funcional. São também chamados de sequências intervenientes (IVS) ou sequências interpostas.[1] Em outras palavras, os íntrões são regiões não-codificantes que são eliminadas por splicing antes da tradução.[2][3] A palavra íntrão é derivada do termo região intragênica (do inglês, "intragenic regions")[4], ou seja, corresponde a uma região dentro de um gene. O termo íntrão refere-se tanto à sequência de DNA dentro de um gene, quanto à sequência correspondente em transcritos de RNA. As sequências que são unidas no RNA maduro final após o splicing do RNA são exões.[5]
Inicialmente, os intrões eram considerados parte do DNA-lixo, por serem regiões não-codificantes de proteínas; no entanto, pesquisas posteriores demonstraram o papel dos intrões na regulação da expressão gênica[6] e também em um processo chamado splicing alternativo. Os intrões são encontrados nos genes da maioria dos organismos e de muitos vírus, e podem estar localizados em uma ampla gama de genes, incluindo aqueles que produzem proteínas, RNA ribossômico (RNAr) e RNA de transferência (RNAt). Quando as proteínas são geradas a partir de genes contendo intrões, o splicing do RNA ocorre como parte da via de processamento do RNA que segue a transcrição e precede a tradução.[5]
Descoberta
Em 1977, Richard Roberts e Philip Sharp, trabalhando de forma independente, durante os estudos de replicação de adenovírus em cultura de células humanas, demonstraram que os genes poderiam ser descontínuos.[7] Em 1978, o bioquímico estadunidense Walter Gilbert cunhou o termo intron para se referir às sequências não codificantes do DNA.[8]
Distribuição
A frequência de intrões em diferentes genomas varia consideravelmente em todo o espectro de organismos biológicos. Por exemplo, intrões são muito comuns dentro do genoma nuclear de vertebrados com mandíbula (como humanos e ratos), onde os genes codificadores de proteínas quase sempre contêm vários intrões. Por outro lado, os intrões são raros dentro dos genes nucleares de alguns microrganismos eucarióticos, por exemplo, a levedura (Saccharomyces cerevisiae).[9] Em contraste, os genomas mitocondriais de vertebrados são totalmente desprovidos de intrões, enquanto os de microrganismos eucarióticos podem conter muitos intrões.[10]
O gene Drosophila Dhc7 contém um intrão de mais de 3,6 Mb, que leva três dias para ser transcrito.[11][12] Em contraste, o menor comprimento de um intrão eucariótico conhecido possui 30 pares de bases, pertencendo ao gene humano MST1L.[13]
Classificação
Existem pelo menos quatro classes principais de intrões:[14][15]
- Íntrões auto-splicing do grupo I que são removidos por catálise de RNA
- Íntrões auto-splicing do grupo II que são removidos por catálise de RNA
- Íntrões em genes de RNA de transferência nuclear e de arqueias que são removidos por proteínas (intrões de RNAt)
- Íntrões em genes que codificam proteínas nucleares que são removidos por spliceossomos (intrões spliceossômicos)
Os intrões do grupo III são propostos como uma quinta família, mas pouco se sabe sobre o aparato bioquímico que medeia seu splicing. Eles parecem estar relacionados aos intrões do grupo II e, possivelmente, aos intrões spliceossômicos.
Íntrões do grupo I e II
intrões do grupo I e II são encontrados em genes codificadores de proteínas (RNA mensageiro), RNA de transferência e RNA ribossômico, em uma gama muito ampla entre os organismos vivos.[16] Após a transcrição em RNA, os íntrons do grupo I e do grupo II também realizam extensas interações internas que permitem que eles se dobrem em uma arquitetura tridimensional complexa e específica. Essas arquiteturas complexas permitem que alguns intrões do grupo I e do grupo II sejam auto-splicing, ou seja, a molécula de RNA que contém o intrões pode reorganizar sua própria estrutura covalente de modo a remover com precisão o intrões e ligar os exons na ordem correta. Em alguns casos, proteínas de ligação a intrões específicas estão envolvidas no splicing, auxiliando o intron a dobrar-se na estrutura tridimensional necessária para a atividade de self-splicing. Os intrões do grupo I e do grupo II são diferenciados por diferentes conjuntos de sequências conservadas internas e estruturas dobradas, e pelo fato de que o splicing de moléculas de RNA contendo íntrons do grupo II gera íntrons ramificados (como aqueles de RNAs spliceossômicos), enquanto os íntrons do grupo I usam um nucleotídeo de guanosina não codificado (tipicamente GTP) para iniciar o splicing, adicionando-o à extremidade 5' do intrões excisado.
Íntrões de RNAt
Os intrões de RNA de transferência que dependem de proteínas para remoção ocorrem em um local específico dentro da alça anticódon de precursores de tRNA não duplicados e são removidos por uma endonuclease de splicing de tRNA. Os exons são então ligados por uma segunda proteína, a ligase de splicing do tRNA.[17] Observe que os intrões autossplicáveis às vezes também são encontrados nos genes de RNAt.[18]
Íntrões spliceossômicos
Os intrões do pré-RNAm nuclear (intrões spliceossômicos) são caracterizados por sequências de intrões específicas localizadas nos limites entre os intrões e os exons.[19] Essas sequências são reconhecidas por moléculas de RNA spliceosomal quando as reações de splicing são iniciadas.[20] Além disso, eles contêm um ponto de ramificação, uma sequência de nucleotídeos particular perto da extremidade 3' do intrão, que se liga covalentemente à extremidade 5' do intrão durante o processo de splicing, gerando um intron ramificado (lariat). Além desses três elementos curtos conservados, as sequências do intrão do pré-RNAm nuclear são altamente variáveis. Os íntrons pré-RNAm nucleares são frequentemente muito mais longos do que os exons circundantes.
Funções biológicas e evolução
Embora os intrões não codifiquem produtos proteicos, eles são essenciais para a regulação da expressão gênica. Alguns intrões codificam RNAs funcionais por meio de processamento posterior após o splicing para gerar moléculas de RNA não-codificante.[21] O splicing alternativo é amplamente usado para gerar várias proteínas de um único gene. Além disso, alguns intrões desempenham papéis essenciais em uma ampla gama de funções regulatórias de expressão gênica.[22][23]
As origens biológicas dos intrões são obscuras. Após a descoberta inicial de intrões em genes codificadores de proteínas do núcleo eucariótico, houve um debate significativo sobre a origem dos intrões. A primeira hipótese afirma que os intrões foram herdados de um ancestral em comum entre os eucariotos e procariotos, e teria sido posteriormente perdido em procariotos. A segunda hipótese assume que os intrões teriam surgido recentemente no processo evolutivo. Outra teoria é que o spliceossomo e a estrutura íntron-exon dos genes é uma relíquia do mundo de RNA.[24] Ainda há um debate considerável sobre até que ponto essas hipóteses são mais corretas. O consenso popular no momento é que os intrões surgiram dentro da linhagem dos eucariotos como elementos egoístas.[25]
Referências
- ↑ «Descritores em Ciências da Saúde: DeCS». São Paulo: BIREME/OPAS/OMS. 2017. Consultado em 21 de outubro de 2020
- ↑ Ghosh, Shampa; Sinha, Jitendra Kumar (2017). Vonk, Jennifer; Shackelford, Todd, eds. «Intron». Springer International Publishing. Encyclopedia of Animal Cognition and Behavior (em inglês): 1–5. ISBN 978-3-319-47829-6. doi:10.1007/978-3-319-47829-6_70-1
- ↑ Editors B. D. «Intron». Biology Dictionary (em inglês). 2017-08-06
- ↑ Lopes, Sônia (2004). Bio: volume único. [S.l.]: Editora Saraiva. ISBN 8502047965
- ↑ a b Lewin Benjamin (1987). Genes 3 ed. New York: [s.n.] pp. 159–179, 386. ISBN 0-471-83278-2. OCLC 14069165
- ↑ Cohen, Jon (2010). Almost Chimpanzee. Searching for What Makes us Human, in Rainforests, Labs, Sanctuaries, and Zoos (em inglês). New York: Times Books. p. 24-26. 369 páginas. ISBN 978-0-8050-8307-1)
- ↑ Voet, D.; Voet, J.G.; Pratt, C.W. (2014). Fundamentos de Bioquímica - 4.ed.: A Vida em Nível Molecular. [S.l.]: Artmed Editora. ISBN 978-85-8271-066-1
- ↑ Gilbert, Walter (fevereiro de 1978). «Why genes in pieces?». Nature. 271 (5645): 501–501. ISSN 0028-0836. doi:10.1038/271501a0. Consultado em 21 de outubro de 2020
- ↑ Stajich, Jason E; Dietrich, Fred S; Roy, Scott W (2007). «Comparative genomic analysis of fungal genomes reveals intron-rich ancestors». Genome Biology. 8 (10): –223. ISSN 1465-6906. doi:10.1186/gb-2007-8-10-r223. Consultado em 22 de outubro de 2020
- ↑ Taanman, Jan-Willem (fevereiro de 1999). «The mitochondrial genome: structure, transcription, translation and replication». Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1410 (2): 103–123. ISSN 0005-2728. doi:10.1016/S0005-2728(98)00161-3. Consultado em 22 de outubro de 2020
- ↑ Tollervey, David; Caceres, Javier F (novembro de 2000). «RNA Processing Marches on». Cell. 103 (5): 703–709. ISSN 0092-8674. doi:10.1016/S0092-8674(00)00174-4. Consultado em 22 de outubro de 2020
- ↑ Reugels, Alexander M.; Kurek, Roman; Lammermann, Ulrich; Bünemann, Hans (2000). «Mega-introns in the Dynein Gene DhDhc7(Y) on the Heterochromatic Y Chromosome Give Rise to the Giant Threads Loops in Primary Spermatocytes of Drosophila hydei». Genetics. 154 (2): 759–769. ISSN 0016-6731
- ↑ Piovesan, Allison; Caracausi, Maria; Ricci, Marco; Strippoli, Pierluigi; Vitale, Lorenza; Pelleri, Maria Chiara (dezembro de 2015). «Identification of minimal eukaryotic introns through GeneBase, a user-friendly tool for parsing the NCBI Gene databank». DNA Research. 22 (6): 495–503. ISSN 1340-2838. doi:10.1093/dnares/dsv028. Consultado em 22 de outubro de 2020
- ↑ Haugen, Peik; Simon, Dawn M; Bhattacharya, Debashish (2005). «The natural history of group I introns». TRENDS in Genetics. 21 (2): 111–119
- ↑ Alberts, B. (2017). Biologia Molecular Da Célula. [S.l.]: ARTMED. ISBN 978-85-8271-422-5
- ↑ Cech, T. R. (1990). «Self-splicing of group I introns». Annual Review of Biochemistry. 59: 543–568. ISSN 0066-4154. PMID 2197983. doi:10.1146/annurev.bi.59.070190.002551
- ↑ Greer, C. L.; Peebles, C. L.; Gegenheimer, P.; Abelson, J. (fevereiro de 1983). «Mechanism of action of a yeast RNA ligase in tRNA splicing». Cell. 32 (2): 537–546. ISSN 0092-8674. PMID 6297798. doi:10.1016/0092-8674(83)90473-7
- ↑ Reinhold-Hurek, B.; Shub, D. A. (14 de maio de 1992). «Self-splicing introns in tRNA genes of widely divergent bacteria». Nature. 357 (6374): 173–176. ISSN 0028-0836. PMID 1579169. doi:10.1038/357173a0
- ↑ Padgett, R. A.; Grabowski, P. J.; Konarska, M. M.; Seiler, S.; Sharp, P. A. (1986). «Splicing of messenger RNA precursors». Annual Review of Biochemistry. 55: 1119–1150. ISSN 0066-4154. PMID 2943217. doi:10.1146/annurev.bi.55.070186.005351
- ↑ Guthrie, C; Patterson, B (dezembro de 1988). «Spliceosomal SNRNAS». Annual Review of Genetics. 22 (1): 387–419. ISSN 0066-4197. doi:10.1146/annurev.ge.22.120188.002131. Consultado em 22 de outubro de 2020
- ↑ Rearick D, Prakash A, McSweeny A, Shepard SS, Fedorova L, Fedorov A (março 2011). «Critical association of ncRNA with introns». Nucleic Acids Res. 39 (6): 2357–66. PMC 3064772
 . PMID 21071396. doi:10.1093/nar/gkq1080
. PMID 21071396. doi:10.1093/nar/gkq1080
- ↑ Bicknell AA, Cenik C, Chua HN, Roth FP, Moore MJ (dezembro 2012). «Introns in UTRs: why we should stop ignoring them.». BioEssays. 34 (12): 1025–34. PMID 23108796. doi:10.1002/bies.201200073
- ↑ Cenik, Can; Chua, Hon Nian; Zhang, Hui; Tarnawsky, Stefan P.; Akef, Abdalla; Derti, Adnan; Tasan, Murat; Moore, Melissa J.; Palazzo, Alexander F.; Roth, Frederick P. (2011). Snyder, Michael, ed. «Genome Analysis Reveals Interplay between 5′UTR Introns and Nuclear mRNA Export for Secretory and Mitochondrial Genes». PLOS Genetics. 7 (4): e1001366. ISSN 1553-7404. PMC 3077370
 . PMID 21533221. doi:10.1371/journal.pgen.1001366
. PMID 21533221. doi:10.1371/journal.pgen.1001366
- ↑ Penny D, Hoeppner MP, Poole AM, Jeffares DC (novembro 2009). «An overview of the introns-first theory». Journal of Molecular Evolution. 69 (5): 527–40. Bibcode:2009JMolE..69..527P. PMID 19777149. doi:10.1007/s00239-009-9279-5
- ↑ Cavalier-Smith, T (1985). «Selfish DNA and the origin of introns.». Nature. 315 (6017): 283–4. Bibcode:1985Natur.315..283C. PMID 2987701. doi:10.1038/315283b0
