Base J
| Base J Alerta sobre risco à saúde | |
|---|---|
| |
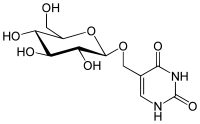
| Nome IUPAC | 5-[[(3R,4S,5S,6R)-3,4,5-Trihidroxi-6-(hidroximetil)tetrahidropiran-2-il]oximetil]-1H-pirimidina-2,4-diona |
| Outros nomes | β-D-Glicosil-5-hidroximetiluracilo β-D-Glicosil-hidroximetiluracilo |
| Identificadores | |
| Número CAS | |
| ChemSpider | |
| SMILES |
|
| InChI | 1/C11H16N2O8/c14-2-5-6(15)7(16)8(17)10(21-5)20-3-4-1-12-11(19)13-9(4)18/h1,5-8,10,14-17H,2-3H2,(H2,12,13,18,19)/t5-,6-,7+,8-,10-/m1/s1
|
| Propriedades | |
| Fórmula química | C11H16N2O8 |
| Massa molar | 304.21 g mol-1 |
| Página de dados suplementares | |
| Estrutura e propriedades | n, εr, etc. |
| Dados termodinâmicos | Phase behaviour Solid, liquid, gas |
| Dados espectrais | UV, IV, RMN, EM |
| Exceto onde denotado, os dados referem-se a materiais sob condições normais de temperatura e pressão Referências e avisos gerais sobre esta caixa. Alerta sobre risco à saúde. | |
A base J ou β-DGlucopyranosyloxymethyluracil é uma base nitrogenada hipermodificada que se encontra no ADN de protozoas Kinetoplastea, incluindo o patógeno humano tripanossoma e nalguns flagelados. Foi descoberta em 1993 no Trypanosoma brucei e foi a primeira nucleobase hipermodificada encontrada no ADN eucariótico; desde então foram descobertos noutros Kinetoplastea, como o Leishmania e nalguns flagelados.[1][2] Nesses organismos a base J actua como terminação da transcrição para a ARN polimerase II; com a sua eliminação em células nocaute acompanhada de uma massiva leitura previa (read-through) dos sítios de terminação da ARN polimerase II, o que em última análise prova ser letal para a célula.[3][4]
A base J é sintetizada por meio de uma hidroxilação inicial da timidina, seguida da glicosilação por uma glicosiltransferase ainda não identificada.[1]
Referências[editar | editar código-fonte]
- ↑ a b Borst, Piet; Sabatini, Robert (outubro de 2008). «Base J: Discovery, Biosynthesis, and Possible Functions». Annual Review of Microbiology. 62 (1): 235–251. PMID 18729733. doi:10.1146/annurev.micro.62.081307.162750
- ↑ Simpson L (1998). «A base called J». Proc Natl Acad Sci USA. 95 (5): 2037–2038. Bibcode:1998PNAS...95.2037S. PMC 33841
 . PMID 9482833. doi:10.1073/pnas.95.5.2037
. PMID 9482833. doi:10.1073/pnas.95.5.2037
- ↑ van Luenen, Henri G.A.M.; Farris, Carol; Jan, Sabrina; Genest, Paul-Andre; Tripathi, Pankaj; Velds, Arno; Kerkhoven, Ron M.; Nieuwland, Marja; Haydock, Andrew; Ramasamy, Gowthaman; Vainio, Saara; Heidebrecht, Tatjana; Perrakis, Anastassis; Pagie, Ludo; van Steensel, Bas; Myler, Peter J.; Borst, Piet (agosto de 2012). «Glucosylated Hydroxymethyluracil, DNA Base J, Prevents Transcriptional Readthrough in Leishmania». Cell. 150 (5): 909–921. PMC 3684241
 . PMID 22939620. doi:10.1016/j.cell.2012.07.030
. PMID 22939620. doi:10.1016/j.cell.2012.07.030
- ↑ Hazelbaker, Dane Z.; Buratowski, Stephen (novembro de 2012). «Transcription: Base J Blocks the Way». Current Biology. 22 (22): R960–R962. PMC 3648658
 . PMID 23174300. doi:10.1016/j.cub.2012.10.010
. PMID 23174300. doi:10.1016/j.cub.2012.10.010
