Codão de terminação
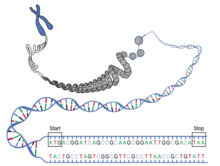
No código genético, um codão de terminação (português europeu) ou códon de terminação (português brasileiro)) (em inglês, stop codon) é uma tripla de nucleótidos dentro do ARN mensageiro que sinaliza a terminação da tradução.[1]
Proteínas são baseadas em polipeptídeos, que são sequências únicas de aminoácidos. A maioria dos codões no ARN mensageiro correspondem à adição de um aminoácido em uma cadeia polipeptídica crescente, que pode vir a tornar-se uma proteína. Codões de parada sinalizam o término deste processo através da ligação de fatores de liberação, que fazem com que as subunidades ribossomais dissociem-se, liberando a cadeia de aminoácidos.[2][3]
No código genético padrão, existem diversos codões de parada:
O codão UGA foi recentemente identificado como o codão de codificação para Selenocisteína (Sec). Este aminoácido é encontrado em 25 selenoproteínas onde ele está localizado no sítio ativo da proteína. A transcrição deste codão é habilitada pela proximidade do elemento SECIS.[4] O codão UAG pode se traduzir em pirrolisina em uma maneira similar a qual a selenocisteína é codificada.
Mutações sem sentido são mudanças na seqüência de ADN que introduzem um codão de parada prematuro, fazendo com que qualquer proteína resultante seja anormalmente encurtada. Isso muitas vezes causa uma perda de função da proteína, uma vez que partes críticas da cadeia de aminoácidos não são mais criadas. Devido a esta terminologia, codões de parada são também referidos como codões sem sentido.
Nomenclatura âmbar, ocre e opala
[editar | editar código-fonte]Foram dados historicamente aos codões de parada muitos nomes diferentes, uma vez que cada um deles correspondia a uma classe distinta de mutantes que se comportavam todos de maneira similar. Esses mutantes foram isolados pela primeira vez dentro dos bacteriófagos (T4 e lambda), vírus que infectam as bactérias Escherichia coli. Mutações nos genes virais enfraqueceram sua capacidade infecciosa, às vezes criando vírus que eram capazes de infectar e crescer dentro de apenas algumas variedades de E coli.
Ver também
Referências
- ↑ Griffiths AJF, Miller JH, Suzuki DT, Lewontin RC, e Gelbart, WM (2000). «Capítulo 10 (Molecular Biology of Gene Function): Genetic code: Stop codons». An Introduction to Genetic Analysis. Bedford: W. H. Freeman and Company. ISBN 978-0716768876
- ↑ Alberts, Bruce; Johnson, Alexander; Lewis, Julian; Raff, Martin; Roberts, Keith; Walter, Peter (2010). «Capítulo 6 (Como as Células Leem o Genoma: Do DNA à Proteína)». Biologia Molecular da Célula 5 ed. Porto Alegre: Artmed. pp. 367–381. ISBN 978-85-363-2066-3
- ↑ Karp Gerald (2008). Cell and Molecular Biology. Concepts and Experiments (em inglês) 5 ed. New Jersey: John Wiley & Sons. 475 páginas. ISBN 978-0-470-04217-5
- ↑ Papp, Laura Vanda; Lu, Jun; Holmgren, Arne; Khanna, Kum Kum (2007). «From Selenium to Selenoproteins: Synthesis, Identity, and Their Role in Human Health». Antioxidants & Redox Signaling. 9 (7). pp. 775–806. PMID 17508906. doi:10.1089/ars.2007.1528
