Gene sobreposto
Um gene sobreposto (ou overlapping gene - OLG)[1][2] é um gene cuja sequência de nucleotídeos expressáveis se sobrepõe parcialmente à sequência de nucleotídeos expressáveis de outro gene.[3] Dessa forma, uma sequência de nucleotídeos pode contribuir para a função de um ou mais produtos gênicos. Os genes sobrepostos estão presentes e são uma característica fundamental dos genomas celulares e virais.[2] A definição atual de um gene sobreposto varia significativamente entre eucariotos, procariotos e vírus.[2] Em procariotos e vírus, a sobreposição deve ser entre regiões de codificação, mas não entre transcritos de mRNA, e é definida quando essas sequências de codificação compartilham um nucleotídeo na mesma fita ou em fitas opostas. Em eucariotos, a sobreposição de genes é quase sempre definida como sobreposição de transcrição de mRNA. Especificamente, uma sobreposição gênica em eucariotos é definida quando pelo menos um nucleotídeo é compartilhado entre os limites das transcrições primárias de mRNA de dois ou mais genes, de modo que uma mutação na base do DNA em qualquer ponto da região sobreposta afetaria as transcrições de todos os genes envolvidos. Essa definição inclui regiões não codificantes (untranslated region - UTRs) de 5′ e 3′, além de íntrons.
O overprinting refere-se a um tipo de sobreposição em que toda ou parte da sequência de um gene é lida em um quadro de leitura alternativo de outro gene no mesmo locus.[4] Acredita-se que as fases de leitura abertas (open reading frame - ORF) alternativos sejam criados por substituições críticas de nucleotídeos em um gene preexistente expressável, que pode ser induzido a expressar uma nova proteína, preservando a função do gene original.[5] A hipótese de overprinting foi levantada como um mecanismo para o surgimento de novos genes a partir de sequências de novo, sejam elas genes mais antigos ou regiões anteriormente não codificantes do genoma.[6] Acredita-se que a maioria dos genes sobrepostos, ou genes cujas sequências de nucleotídeos expressáveis se sobrepõem parcialmente umas às outras, evoluiu em parte devido a esse mecanismo, sugerindo que cada sobreposição é composta por um gene ancestral e um gene novo.[7] Posteriormente, acredita-se que o overprinting também seja uma fonte de novas proteínas, pois as proteínas de novo codificadas por esses novos genes geralmente não têm homólogos remotos nos bancos de dados.[8] Os genes impressos em excesso são características particularmente comuns da organização genômica dos vírus, o que provavelmente aumenta muito o número de genes potencialmente expressáveis a partir de um pequeno conjunto de informações genéticas virais.[9] É provável que a impressão em excesso seja responsável pela geração de várias proteínas novas pelos vírus no decorrer de sua história evolutiva.
Classificação[editar | editar código-fonte]
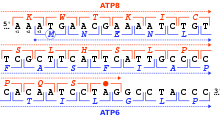
Tandem out-of-phase overlap of the human mitochondrial genes ATP8 (+1 frame, in red) and ATP6 (+3 frame, in blue)
Os genes podem se sobrepor de várias maneiras e podem ser classificados por suas posições em relação uns aos outros.[3][11][12][13][14]
- Sobreposição unidirecional ou em tandem: a extremidade 3' de um gene se sobrepõe à extremidade 5' de outro gene na mesma fita. Esse arranjo pode ser simbolizado com a notação → →, em que as setas indicam o quadro de leitura do início ao fim.
- Sobreposição convergente ou de extremidade: as extremidades 3' dos dois genes se sobrepõem em fitas opostas. Isso pode ser escrito como → ←.
- Sobreposição divergente ou tail-on: as extremidades 5' dos dois genes se sobrepõem em fitas opostas. Isso pode ser escrito como ← →.
Os genes sobrepostos também podem ser classificados por fases, que descrevem seus quadros de leitura relativos:[3][11][12][13][14]
- A sobreposição em fase ocorre quando as sequências compartilhadas usam o mesmo quadro de leitura. Isso também é conhecido como "fase 0". Os genes unidirecionais com sobreposição de fase 0 não são considerados genes distintos, mas sim locais de início alternativos do mesmo gene.
- As sobreposições fora de fase ocorrem quando as sequências compartilhadas usam quadros de leitura diferentes. Isso pode ocorrer na "fase 1" ou na "fase 2", dependendo do fato de os quadros de leitura serem deslocados em 1 ou 2 nucleotídeos. Como um códon tem três nucleotídeos de comprimento, um deslocamento de três nucleotídeos é um quadro de fase 0 em fase.
Estudos sobre genes sobrepostos sugerem que sua evolução pode ser resumida em dois modelos possíveis:[4] Em um modelo, as duas proteínas codificadas por seus respectivos genes sobrepostos evoluem sob pressões de seleção semelhantes. As proteínas e a região de sobreposição são altamente conservadas quando é favorecida uma forte seleção contra a mudança de aminoácidos. Os genes sobrepostos evoluem sob restrições rigorosas, pois uma única substituição de nucleotídeo é capaz de alterar a estrutura e a função das duas proteínas simultaneamente. Um estudo sobre o vírus da hepatite B (HBV), cujo genoma de DNA contém vários genes sobrepostos, mostrou que o número médio de substituições de nucleotídeos sinônimos por local nas regiões codificadoras sobrepostas foi significativamente menor do que o das regiões não sobrepostas.[15] O mesmo estudo mostrou que era possível que algumas dessas regiões sobrepostas e suas proteínas divergissem significativamente do original quando havia uma seleção fraca contra a mudança de aminoácidos. O domínio espaçador da polimerase e a região pré-S1 de uma proteína de superfície do HBV, por exemplo, tinham uma porcentagem de aminoácidos conservados de 30% e 40%, respectivamente.[15] No entanto, sabe-se que essas regiões de sobreposição são menos importantes para a replicação em comparação com as regiões de sobreposição que foram altamente conservadas entre diferentes cepas de HBV, que são absolutamente essenciais para o processo.
O segundo modelo sugere que as duas proteínas e seus respectivos genes de sobreposição evoluem sob pressões de seleção opostas: um quadro sofre seleção positiva, enquanto o outro está sob seleção negativa (ou purificadora). Nos Tombusvírus, as proteínas p19 e p22 são codificadas por genes sobrepostos que formam uma região codificadora de 549 nt, e a p19 está sob seleção positiva, enquanto a p22 está sob seleção purificadora.[16] Outros exemplos são mencionados em estudos envolvendo genes sobrepostos do vírus Sendai,[17] vírus do enrolamento da folha da batata,[18] e parvovírus humano B19.[19] Sugere-se que esse fenômeno de genes sobrepostos que sofrem pressões de seleção diferentes seja consequência de uma alta taxa de substituição de nucleotídeos com efeitos diferentes nos dois quadros; as substituições podem ser majoritariamente não sinônimas para um quadro, enquanto a maioria é sinônima para o outro quadro.[4]
Evolução[editar | editar código-fonte]
A sobreposição de genes é particularmente comum em genomas que evoluem rapidamente, como os de vírus, bactérias e mitocôndrias. Eles podem se originar de três maneiras:[20]
- Pela extensão de uma ORF existente a jusante em um gene contíguo devido à perda de um códon de parada;
- Pela extensão de uma ORF existente a montante em um gene contíguo devido à perda de um códon de iniciação;
- Pela geração de uma nova ORF dentro de uma existente devido a uma mutação pontual.
O uso da mesma sequência de nucleotídeos para codificar vários genes pode proporcionar vantagem evolutiva devido à redução do tamanho do genoma e à oportunidade de co-regulação da transcrição e tradução dos genes sobrepostos.[12][21][22][23] As sobreposições de genes introduzem novas restrições evolutivas nas sequências das regiões de sobreposição.[14][24]
Origens de novos genes[editar | editar código-fonte]

Em 1977, Pierre-Paul Grassé propôs que um dos genes do par poderia ter se originado de novo por meio de mutações para introduzir novas ORF em quadros de leitura alternativos; ele descreveu o mecanismo como overprinting.[25] Posteriormente, foi comprovado por Susumu Ohno, que identificou um gene candidato que pode ter surgido por esse mecanismo.[26] Alguns genes de novo originados dessa forma podem não permanecer sobrepostos, mas subfuncionalizar-se após a duplicação do gene,[6] contribuindo para a prevalência de genes órfãos. O membro mais jovem de um par de genes sobrepostos pode ser identificado bioinformaticamente por uma distribuição filogenética mais restrita ou pelo uso menos otimizado de códons.[9][27][28] Os membros mais jovens do par tendem a ter maior desordem estrutural intrínseca do que os membros mais velhos, mas os membros mais velhos também são mais desordenados do que outras proteínas, presumivelmente como uma forma de aliviar as restrições evolutivas maiores impostas pela sobreposição.[27] As sobreposições têm maior probabilidade de se originar em proteínas que já têm alta desordem.[27]
Distribuição taxonômica[editar | editar código-fonte]

A sobreposição de genes ocorre em todos os domínios da vida, embora com frequências variadas. Eles são especialmente comuns em genomas virais.
Vírus[editar | editar código-fonte]
A existência de genes sobrepostos foi identificada pela primeira vez no vírus ΦX174, cujo genoma foi o primeiro genoma de DNA sequenciado por Frederick Sanger em 1977.[29] A análise anterior do ΦX174, um pequeno bacteriófago de DNA de fita simples que infectou a bactéria Escherichia coli, sugeriu que as proteínas produzidas durante a infecção exigiam sequências de codificação mais longas do que o comprimento medido de seu genoma.[31] A análise do genoma de 5.386 nucleotídeos totalmente sequenciado mostrou que o vírus possuía uma extensa sobreposição entre as regiões de codificação, revelando que alguns genes (como os genes D e E) foram traduzidos a partir das mesmas sequências de DNA, mas em diferentes quadros de leitura.[29][31] Foi demonstrado que um local de início alternativo dentro do gene A de replicação do genoma do ΦX174 expressava uma proteína truncada com uma sequência de codificação idêntica ao C-terminal da proteína A original, mas com uma função diferente.[32][33] Concluiu-se que outros locais não descobertos de síntese de polipeptídeos poderiam estar ocultos no genoma devido à sobreposição de genes. Foi demonstrado que um gene de novo identificado de outro locus de gene sobreposto expressa uma nova proteína que induz a lise de E. coli inibindo a biossíntese de sua parede celular, sugerindo que a criação de uma proteína de novo por meio do processo de impressão sobreposta pode ser um fator significativo na evolução da patogenicidade dos vírus.[4] Outro exemplo é o gene ORF3d do vírus SARS-CoV-2.[1][34] Os genes sobrepostos são particularmente comuns em genomas virais.[9] Alguns estudos atribuem essa observação à pressão seletiva em direção a pequenos tamanhos de genoma mediados pelas restrições físicas do empacotamento do genoma em um capsídeo viral, particularmente um de geometria icosaédrica.[35] No entanto, outros estudos contestam essa conclusão e argumentam que é mais provável que a distribuição de sobreposições em genomas virais reflita a impressão sobreposta como a origem evolutiva dos genes virais sobrepostos.[36] O overprinting é uma fonte comum de genes de novo em vírus.[28]
A proporção de vírus com sequências de codificação sobrepostas em seus genomas varia.[2] Os vírus de RNA de fita dupla têm menos de um quarto dos vírus que as contêm, enquanto quase três quartos dos retrovírus e vírus com genomas de DNA de fita simples contêm sequências de codificação sobrepostas.[37] Os vírus segmentados, em particular, ou vírus com seu genoma dividido em partes separadas e empacotados no mesmo capsídeo ou em capsídeos separados, têm maior probabilidade de conter uma sequência sobreposta do que os vírus não segmentados.[37] Os vírus de RNA têm menos genes sobrepostos do que os vírus de DNA, que possuem taxas de mutação mais baixas e tamanhos de genoma menos restritivos.[37][38] A taxa de mutação mais baixa dos vírus de DNA facilita uma maior novidade genômica e a exploração evolutiva dentro de um genoma estruturalmente restrito e pode ser o principal impulsionador da evolução dos genes sobrepostos.[39][40]
Estudos de genes virais impressos em excesso sugerem que seus produtos proteicos tendem a ser proteínas acessórias que não são essenciais para a proliferação viral, mas contribuem para a patogenicidade. As proteínas sobreimpressas geralmente têm distribuições incomuns de aminoácidos e altos níveis de desordem intrínseca.[41] Em alguns casos, as proteínas sobreimpressas têm estruturas tridimensionais bem definidas, mas novas;[42] um exemplo é o supressor de silenciamento de RNA p19 encontrado em Tombusvírus, que tem uma nova dobra de proteína e um novo modo de ligação no reconhecimento de siRNA.[28][30][43]
Procariotos[editar | editar código-fonte]
As estimativas de sobreposição de genes em genomas bacterianos normalmente constatam que cerca de um terço dos genes bacterianos estão sobrepostos, embora geralmente por apenas alguns pares de bases.[12][44][45] A maioria dos estudos de sobreposição em genomas bacterianos encontra evidências de que a sobreposição tem uma função na regulação de genes, permitindo que os genes sobrepostos sejam co-regulados por transcrição e tradução.[12][23] Nos genomas procarióticos, as sobreposições unidirecionais são mais comuns, possivelmente devido à tendência dos genes procarióticos adjacentes de compartilhar a orientação.[11][12][14] Entre as sobreposições unidirecionais, as sobreposições longas são mais comumente lidas com um deslocamento de um nucleotídeo no quadro de leitura (ou seja, fase 1) e as sobreposições curtas são mais comumente lidas na fase 2.[45][46] As sobreposições longas de mais de 60 pares de bases são mais comuns para genes convergentes; no entanto, as sobreposições longas putativas têm taxas muito altas de anotação incorreta.[47] Exemplos robustamente validados de sobreposições longas em genomas bacterianos são raros; no organismo modelo bem estudado E. coli, apenas quatro pares de genes são bem validados como tendo sobreposições longas e sobreimpressas.[48]
Eucariotos[editar | editar código-fonte]
Em comparação com os genomas procarióticos, os genomas eucarióticos geralmente são mal anotados e, portanto, a identificação de sobreposições genuínas é relativamente desafiadora.[28] Entretanto, exemplos de sobreposições de genes validados foram documentados em vários organismos eucarióticos, inclusive em mamíferos, como camundongos e humanos.[49][50][51][52] Os eucariotos diferem dos procariotos na distribuição dos tipos de sobreposição: enquanto as sobreposições unidirecionais (ou seja, da mesma fita) são mais comuns em procariotos, as sobreposições opostas ou antiparalelas são mais comuns em eucariotos, enquanto as sobreposições unidirecionais (ou seja, da mesma fita) são mais comuns em procariotos, as sobreposições de fita oposta ou antiparalela são mais comuns em eucariotos. Entre as sobreposições de fitas opostas, a orientação convergente é a mais comum.[50] A maioria dos estudos de sobreposição de genes eucarióticos constatou que os genes sobrepostos estão amplamente sujeitos à reorganização genômica, mesmo em espécies intimamente relacionadas e, portanto, a presença de uma sobreposição nem sempre é bem conservada.[51][53] A sobreposição com genes mais antigos ou menos restritos taxonomicamente também é uma característica comum dos genes que provavelmente se originaram de novo em uma determinada linhagem eucariótica.[51][54][55]
Funções[editar | editar código-fonte]
As funções exatas dos genes sobrepostos parecem variar entre os domínios da vida, mas vários experimentos mostraram que eles são importantes para os ciclos de vida dos vírus por meio da expressão e estequiometria adequadas das proteínas,[56] além de desempenharem um papel no dobramento adequado das proteínas.[57] Também foi criada uma versão do bacteriófago ΦX174 em que todas as sobreposições de genes foram removidas ,[58] provando que elas não eram necessárias para a replicação.
A retenção e a evolução de genes sobrepostos dentro dos vírus também podem ser devidas a limitações de tamanho do capsídeo.[59] Foi observada uma perda drástica de viabilidade em vírus com genomas projetados para serem mais longos do que o genoma do tipo selvagem.[60] O aumento do comprimento do genoma de DNA de fita simples do ΦX174 em >1% resulta em perda quase completa de infectividade, que se acredita ser o resultado das restrições físicas rigorosas impostas pelo volume finito do capsídeo.[61] Estudos sobre vírus adeno-associados como vetores de entrega de genes mostraram que o empacotamento viral é restringido por limites de tamanho de carga genética, exigindo o uso de vários vetores para entregar genes humanos grandes, como o CFTR81.[62][63] Portanto, sugere-se que os genes sobrepostos evoluíram como um meio de superar essas restrições físicas, aumentando a diversidade genética utilizando apenas a sequência existente em vez de aumentar o comprimento do genoma.
Métodos para identificar genes e ORF sobrepostos[editar | editar código-fonte]
Os métodos padronizados, como a anotação do genoma, podem ser inadequados para a detecção de genes sobrepostos, pois dependem de genes já curados, enquanto os genes sobrepostos geralmente são ignorados por conterem uma composição de sequência atípica.[2][64][65][66] Os padrões de anotação do genoma também costumam ser tendenciosos em relação a sobreposições de recursos, como genes totalmente contidos em outro gene.[67] Além disso, alguns canalizações de bioinformática, como o MG-RAST, penalizam acentuadamente as sobreposições entre ORF previstas.[68] No entanto, o rápido avanço das ferramentas de medição de proteínas e RNA em escala de genoma, juntamente com algoritmos de previsão cada vez mais avançados, revelou uma avalanche de genes e ORFs sobrepostos em vários genomas.[2] Os métodos proteogenômicos foram essenciais para a descoberta de vários genes sobrepostos e incluem uma combinação de técnicas como proteômica de baixo para cima, perfil de ribossomos, sequenciamento de DNA e perturbação. O sequenciamento de RNA também é usado para identificar regiões genômicas que contêm transcrições sobrepostas. Ele foi utilizado para identificar 180.000 ORFs alternativas dentro de regiões codificadoras previamente anotadas encontradas em seres humanos.[69] ORFs recém-descobertas como essas são verificadas por meio de uma variedade de técnicas de genética inversa, como CRISPR-Cas9 e interrupção de Cas9 cataliticamente morta (dCas9).[70][71][72] Também são realizadas tentativas de prova por síntese para mostrar, sem sombra de dúvida, a ausência de quaisquer genes sobrepostos não descobertos.[73]
Ver também[editar | editar código-fonte]
- Região de codificação
- Fase de leitura aberta
- ADN não codificante
- Taxa de mutação
- Edição de genoma
- Engenharia genética
Referências
- ↑ a b Nelson, Chase W; et al. (1 de outubro de 2020). «Dynamically evolving novel overlapping gene as a factor in the SARS-CoV-2 pandemic». eLife. 9. PMC 7655111
 . PMID 33001029. doi:10.7554/eLife.59633. Consultado em 11 de novembro de 2020
. PMID 33001029. doi:10.7554/eLife.59633. Consultado em 11 de novembro de 2020
- ↑ a b c d e f Wright, Bradley W.; Molloy, Mark P.; Jaschke, Paul R. (5 de outubro de 2021). «Overlapping genes in natural and engineered genomes». Nature Reviews Genetics (em inglês). 23 (3): 154–168. ISSN 1471-0064. PMC 8490965
 . PMID 34611352. doi:10.1038/s41576-021-00417-w
. PMID 34611352. doi:10.1038/s41576-021-00417-w
- ↑ a b c Y. Fukuda, M. Tomita et T. Washio (1999). «Comparative study of overlapping genes in the genomes of Mycoplasma genitalium and Mycoplasma pneumoniae». Nucleic Acids Res. 27 (8): 1847–1853. PMC 148392
 . PMID 10101192. doi:10.1093/nar/27.8.1847
. PMID 10101192. doi:10.1093/nar/27.8.1847
- ↑ a b c d Pavesi, Angelo (26 de maio de 2021). «Origin, Evolution and Stability of Overlapping Genes in Viruses: A Systematic Review». Genes (em inglês). 12 (6). 809 páginas. ISSN 2073-4425. PMC 8227390
 . PMID 34073395. doi:10.3390/genes12060809
. PMID 34073395. doi:10.3390/genes12060809
- ↑ Normark, Staffan; Bergström, Sven; Edlund, Thomas; Grundström, Thomas; Jaurin, Bengtake; Lindberg, Frederik P.; Olsson, Olof (dezembro de 1983). «Overlapping Genes». Annual Review of Genetics. 17 (1): 499–525. ISSN 0066-4197. PMID 6198955. doi:10.1146/annurev.ge.17.120183.002435
- ↑ a b Keese, PK; Gibbs, A (15 de outubro de 1992). «Origins of genes: "big bang" or continuous creation?». Proceedings of the National Academy of Sciences of the United States of America. 89 (20): 9489–93. Bibcode:1992PNAS...89.9489K. PMC 50157
 . PMID 1329098. doi:10.1073/pnas.89.20.9489
. PMID 1329098. doi:10.1073/pnas.89.20.9489
- ↑ Keese, P. K.; Gibbs, A. (15 de outubro de 1992). «Origins of genes: "big bang" or continuous creation?». Proceedings of the National Academy of Sciences. 89 (20): 9489–9493. Bibcode:1992PNAS...89.9489K. ISSN 0027-8424. PMC 50157
 . PMID 1329098. doi:10.1073/pnas.89.20.9489
. PMID 1329098. doi:10.1073/pnas.89.20.9489
- ↑ Gibbs, Adrian; Keese, Paul K. (19 de outubro de 1995), «In search of the origins of viral genes», ISBN 9780521455336, Cambridge University Press, Molecular Basis of Virus Evolution, pp. 76–90, doi:10.1017/cbo9780511661686.008, consultado em 3 de dezembro de 2021
- ↑ a b c d Pavesi, Angelo; Magiorkinis, Gkikas; Karlin, David G.; Wilke, Claus O. (15 de agosto de 2013). «Viral Proteins Originated De Novo by Overprinting Can Be Identified by Codon Usage: Application to the "Gene Nursery" of Deltaretroviruses». PLOS Computational Biology. 9 (8): e1003162. Bibcode:2013PLSCB...9E3162P. PMC 3744397
 . PMID 23966842. doi:10.1371/journal.pcbi.1003162
. PMID 23966842. doi:10.1371/journal.pcbi.1003162
- ↑ Anderson S, Bankier AT, Barrell BG, de Bruijn MH, Coulson AR, Drouin J, Eperon IC, Nierlich DP, Roe BA, Sanger F, Schreier PH, Smith AJ, Staden R, Young IG (abril de 1981). «Sequence and organization of the human mitochondrial genome». Nature. 290 (5806): 457–465. Bibcode:1981Natur.290..457A. PMID 7219534. doi:10.1038/290457a0
- ↑ a b c Fukuda, Yoko; Nakayama, Yoichi; Tomita, Masaru (dezembro de 2003). «On dynamics of overlapping genes in bacterial genomes». Gene. 323: 181–187. PMID 14659892. doi:10.1016/j.gene.2003.09.021
- ↑ a b c d e f Johnson Z, Chisholm S (2004). «Properties of overlapping genes are conserved across microbial genomes». Genome Res. 14 (11): 2268–72. PMC 525685
 . PMID 15520290. doi:10.1101/gr.2433104
. PMID 15520290. doi:10.1101/gr.2433104
- ↑ a b Normark S.; Bergstrom S.; Edlund T.; Grundstrom T.; Jaurin B.; Lindberg F.P.; Olsson O. (1983). «Overlapping genes». Annual Review of Genetics. 17: 499–525. PMID 6198955. doi:10.1146/annurev.ge.17.120183.002435
- ↑ a b c d Rogozin, Igor B.; Spiridonov, Alexey N.; Sorokin, Alexander V.; Wolf, Yuri I.; Jordan, I.King; Tatusov, Roman L.; Koonin, Eugene V. (maio de 2002). «Purifying and directional selection in overlapping prokaryotic genes». Trends in Genetics. 18 (5): 228–232. PMID 12047938. doi:10.1016/S0168-9525(02)02649-5
- ↑ a b Mizokami, Masashi; Orito, Etsuro; Ohba, Ken-ichi; Ikeo, Kazuho; Lau, Johnson Y. N.; Gojobori, Takashi (janeiro de 1997). «Constrained evolution with respect to gene overlap of hepatitis B virus». Journal of Molecular Evolution. 44 (S1): S83–S90. Bibcode:1997JMolE..44S..83M. ISSN 0022-2844. PMID 9071016. doi:10.1007/pl00000061
- ↑ Allison, Jane R.; Lechner, Marcus; Hoeppner, Marc P.; Poole, Anthony M. (12 de fevereiro de 2016). «Positive Selection or Free to Vary? Assessing the Functional Significance of Sequence Change Using Molecular Dynamics». PLOS ONE. 11 (2): e0147619. Bibcode:2016PLoSO..1147619A. ISSN 1932-6203. PMC 4752228
 . PMID 26871901. doi:10.1371/journal.pone.0147619
. PMID 26871901. doi:10.1371/journal.pone.0147619
- ↑ Fujii, Yutaka; Kiyotani, Katsuhiro; Yoshida, Tetsuya; Sakaguchi, Takemasa (2001). «Conserved and non-conserved regions in the Sendai virus genome: Evolution of a gene possessing overlapping reading frames». Virus Genes. 22 (1): 47–52. ISSN 0920-8569. PMID 11210938. doi:10.1023/a:1008130318633
- ↑ Guyader, Sébastien; Ducray, Danièle Giblot (1 de julho de 2002). «Sequence analysis of Potato leafroll virus isolates reveals genetic stability, major evolutionary events and differential selection pressure between overlapping reading frame products». Journal of General Virology. 83 (7): 1799–1807. ISSN 0022-1317. PMID 12075102. doi:10.1099/0022-1317-83-7-1799

- ↑ Stamenković, Gorana G.; Ćirković, Valentina S.; Šiljić, Marina M.; Blagojević, Jelena V.; Knežević, Aleksandra M.; Joksić, Ivana D.; Stanojević, Maja P. (24 de outubro de 2016). «Substitution rate and natural selection in parvovirus B19». Scientific Reports. 6 (1): 35759. Bibcode:2016NatSR...635759S. ISSN 2045-2322. PMC 5075947
 . PMID 27775080. doi:10.1038/srep35759
. PMID 27775080. doi:10.1038/srep35759
- ↑ Krakauer, David C. (junho de 2000). «Stability and Evolution of Overlapping Genes». Evolution. 54 (3): 731–739. PMID 10937248. doi:10.1111/j.0014-3820.2000.tb00075.x

- ↑ Delaye, Luis; DeLuna, Alexander; Lazcano, Antonio; Becerra, Arturo (2008). «The origin of a novel gene through overprinting in Escherichia coli». BMC Evolutionary Biology. 8 (1). 31 páginas. PMC 2268670
 . PMID 18226237. doi:10.1186/1471-2148-8-31
. PMID 18226237. doi:10.1186/1471-2148-8-31
- ↑ Saha, Deeya; Podder, Soumita; Panda, Arup; Ghosh, Tapash Chandra (maio de 2016). «Overlapping genes: A significant genomic correlate of prokaryotic growth rates». Gene. 582 (2): 143–147. PMID 26853049. doi:10.1016/j.gene.2016.02.002
- ↑ a b Luo, Yingqin; Battistuzzi, Fabia; Lin, Kui; Gibas, Cynthia (29 de novembro de 2013). «Evolutionary Dynamics of Overlapped Genes in Salmonella». PLOS ONE. 8 (11): e81016. Bibcode:2013PLoSO...881016L. PMC 3843671
 . PMID 24312259. doi:10.1371/journal.pone.0081016
. PMID 24312259. doi:10.1371/journal.pone.0081016
- ↑ Wei, X.; Zhang, J. (31 de dezembro de 2014). «A Simple Method for Estimating the Strength of Natural Selection on Overlapping Genes». Genome Biology and Evolution. 7 (1): 381–390. PMC 4316641
 . PMID 25552532. doi:10.1093/gbe/evu294
. PMID 25552532. doi:10.1093/gbe/evu294
- ↑ Grassé, Pierre-Paul (1977). Evolution of Living Organisms: Evidence for a New Theory of Transformation. [S.l.]: Academic Press. ISBN 9781483274096
- ↑ Ohno, S (abril de 1984). «Birth of a unique enzyme from an alternative reading frame of the preexisted, internally repetitious coding sequence.». Proceedings of the National Academy of Sciences of the United States of America. 81 (8): 2421–5. Bibcode:1984PNAS...81.2421O. PMC 345072
 . PMID 6585807. doi:10.1073/pnas.81.8.2421
. PMID 6585807. doi:10.1073/pnas.81.8.2421
- ↑ a b c Willis, Sara; Masel, Joanna (19 de julho de 2018). «Gene Birth Contributes to Structural Disorder Encoded by Overlapping Genes». Genetics. 210 (1): 303–313. PMC 6116962
 . PMID 30026186. doi:10.1534/genetics.118.301249
. PMID 30026186. doi:10.1534/genetics.118.301249
- ↑ a b c d Sabath, N.; Wagner, A.; Karlin, D. (19 de julho de 2012). «Evolution of Viral Proteins Originated De Novo by Overprinting». Molecular Biology and Evolution. 29 (12): 3767–3780. PMC 3494269
 . PMID 22821011. doi:10.1093/molbev/mss179
. PMID 22821011. doi:10.1093/molbev/mss179
- ↑ a b c Sanger, F.; Air, G. M.; Barrell, B. G.; Brown, N. L.; Coulson, A. R.; Fiddes, J. C.; Hutchison, C. A.; Slocombe, P. M.; Smith, M. (1977). «Nucleotide sequence of bacteriophage ΦX174 DNA». Nature. 265 (5596): 687–95. Bibcode:1977Natur.265..687S. PMID 870828. doi:10.1038/265687a0
- ↑ a b Ye, Keqiong; Malinina, Lucy; Patel, Dinshaw J. (3 de dezembro de 2003). «Recognition of small interfering RNA by a viral suppressor of RNA silencing». Nature. 426 (6968): 874–878. Bibcode:2003Natur.426..874Y. PMC 4694583
 . PMID 14661029. doi:10.1038/nature02213
. PMID 14661029. doi:10.1038/nature02213
- ↑ a b Barrell, B. G.; Air, G. M.; Hutchison, C. A. (novembro de 1976). «Overlapping genes in bacteriophage φX174». Nature (em inglês). 264 (5581): 34–41. Bibcode:1976Natur.264...34B. ISSN 1476-4687. PMID 1004533. doi:10.1038/264034a0
- ↑ LINNEY, ELWOOD; HAYASHI, MASAKI (maio de 1974). «Intragenic regulation of the synthesis of ΦX174 gene A proteins». Nature. 249 (5455): 345–348. Bibcode:1974Natur.249..345L. ISSN 0028-0836. PMID 4601823. doi:10.1038/249345a0
- ↑ Roznowski, Aaron P.; Doore, Sarah M.; Kemp, Sundance Z.; Fane, Bentley A. (6 de janeiro de 2020). «Finally, a Role Befitting A star : Strongly Conserved, Unessential Microvirus A* Proteins Ensure the Product Fidelity of Packaging Reactions». Journal of Virology. 94 (2). ISSN 0022-538X. PMC 6955274
 . PMID 31666371. doi:10.1128/jvi.01593-19
. PMID 31666371. doi:10.1128/jvi.01593-19
- ↑ Dockrill, Peter (11 de novembro de 2020). «Scientists Just Found a Mysteriously Hidden 'Gene Within a Gene' in SARS-CoV-2». ScienceAlert. Consultado em 11 de novembro de 2020
- ↑ Chirico, N.; Vianelli, A.; Belshaw, R. (7 de julho de 2010). «Why genes overlap in viruses». Proceedings of the Royal Society B: Biological Sciences. 277 (1701): 3809–3817. PMC 2992710
 . PMID 20610432. doi:10.1098/rspb.2010.1052
. PMID 20610432. doi:10.1098/rspb.2010.1052
- ↑ Brandes, Nadav; Linial, Michal (21 de maio de 2016). «Gene overlapping and size constraints in the viral world». Biology Direct. 11 (1): 26. PMC 4875738
 . PMID 27209091. doi:10.1186/s13062-016-0128-3
. PMID 27209091. doi:10.1186/s13062-016-0128-3
- ↑ a b c Schlub, Timothy E; Holmes, Edward C (1 de janeiro de 2020). «Properties and abundance of overlapping genes in viruses». Virus Evolution. 6 (1): veaa009. ISSN 2057-1577. PMC 7017920
 . PMID 32071766. doi:10.1093/ve/veaa009
. PMID 32071766. doi:10.1093/ve/veaa009
- ↑ Chirico, Nicola; Vianelli, Alberto; Belshaw, Robert (7 de julho de 2010). «Why genes overlap in viruses». Proceedings of the Royal Society B: Biological Sciences. 277 (1701): 3809–3817. ISSN 0962-8452. PMC 2992710
 . PMID 20610432. doi:10.1098/rspb.2010.1052
. PMID 20610432. doi:10.1098/rspb.2010.1052
- ↑ Brandes, Nadav; Linial, Michal (21 de maio de 2016). «Gene overlapping and size constraints in the viral world». Biology Direct. 11 (1): 26. ISSN 1745-6150. PMC 4875738
 . PMID 27209091. doi:10.1186/s13062-016-0128-3
. PMID 27209091. doi:10.1186/s13062-016-0128-3
- ↑ Pavesi, Angelo (julho de 2020). «New insights into the evolutionary features of viral overlapping genes by discriminant analysis». Virology. 546: 51–66. ISSN 0042-6822. PMC 7157939
 . PMID 32452417. doi:10.1016/j.virol.2020.03.007
. PMID 32452417. doi:10.1016/j.virol.2020.03.007
- ↑ Rancurel, C.; Khosravi, M.; Dunker, A. K.; Romero, P. R.; Karlin, D. (29 de julho de 2009). «Overlapping Genes Produce Proteins with Unusual Sequence Properties and Offer Insight into De Novo Protein Creation». Journal of Virology. 83 (20): 10719–10736. PMC 2753099
 . PMID 19640978. doi:10.1128/JVI.00595-09
. PMID 19640978. doi:10.1128/JVI.00595-09
- ↑ Abroi, Aare (1 de dezembro de 2015). «A protein domain-based view of the virosphere–host relationship». Biochimie. 119: 231–243. PMID 26296474. doi:10.1016/j.biochi.2015.08.008
- ↑ Vargason, Jeffrey M; Szittya, György; Burgyán, József; Hall, Traci M.Tanaka (dezembro de 2003). «Size Selective Recognition of siRNA by an RNA Silencing Suppressor». Cell. 115 (7): 799–811. PMID 14697199. doi:10.1016/S0092-8674(03)00984-X

- ↑ Huvet, Maxime; Stumpf, Michael PH (1 de janeiro de 2014). «Overlapping genes: a window on gene evolvability». BMC Genomics. 15 (1). 721 páginas. ISSN 1471-2164. PMC 4161906
 . PMID 25159814. doi:10.1186/1471-2164-15-721
. PMID 25159814. doi:10.1186/1471-2164-15-721
- ↑ a b Cock, Peter J. A.; Whitworth, David E. (19 de março de 2007). «Evolution of Gene Overlaps: Relative Reading Frame Bias in Prokaryotic Two-Component System Genes». Journal of Molecular Evolution. 64 (4): 457–462. Bibcode:2007JMolE..64..457C. PMID 17479344. doi:10.1007/s00239-006-0180-1
- ↑ Fonseca, M. M.; Harris, D. J.; Posada, D. (5 de novembro de 2013). «Origin and Length Distribution of Unidirectional Prokaryotic Overlapping Genes». G3: Genes, Genomes, Genetics. 4 (1): 19–27. PMC 3887535
 . PMID 24192837. doi:10.1534/g3.113.005652
. PMID 24192837. doi:10.1534/g3.113.005652
- ↑ Pallejà, Albert; Harrington, Eoghan D; Bork, Peer (2008). «Large gene overlaps in prokaryotic genomes: result of functional constraints or mispredictions?». BMC Genomics. 9 (1). 335 páginas. PMC 2478687
 . PMID 18627618. doi:10.1186/1471-2164-9-335
. PMID 18627618. doi:10.1186/1471-2164-9-335
- ↑ Fellner, Lea; Simon, Svenja; Scherling, Christian; Witting, Michael; Schober, Steffen; Polte, Christine; Schmitt-Kopplin, Philippe; Keim, Daniel A.; Scherer, Siegfried; Neuhaus, Klaus (18 de dezembro de 2015). «Evidence for the recent origin of a bacterial protein-coding, overlapping orphan gene by evolutionary overprinting». BMC Evolutionary Biology. 15 (1): 283. PMC 4683798
 . PMID 26677845. doi:10.1186/s12862-015-0558-z
. PMID 26677845. doi:10.1186/s12862-015-0558-z
- ↑ McLysaght, Aoife; Guerzoni, Daniele (31 de agosto de 2015). «New genes from non-coding sequence: the role of de novo protein-coding genes in eukaryotic evolutionary innovation». Philosophical Transactions of the Royal Society B: Biological Sciences. 370 (1678). 20140332 páginas. PMC 4571571
 . PMID 26323763. doi:10.1098/rstb.2014.0332
. PMID 26323763. doi:10.1098/rstb.2014.0332
- ↑ a b C. Sanna, W. Li et L. Zhang (2008). «Overlapping genes in the human and mouse genomes». BMC Genomics. 9 (169). 169 páginas. PMC 2335118
 . PMID 18410680. doi:10.1186/1471-2164-9-169
. PMID 18410680. doi:10.1186/1471-2164-9-169
- ↑ a b c Makałowska, Izabela; Lin, Chiao-Feng; Hernandez, Krisitina (2007). «Birth and death of gene overlaps in vertebrates». BMC Evolutionary Biology. 7 (1). 193 páginas. PMC 2151771
 . PMID 17939861. doi:10.1186/1471-2148-7-193
. PMID 17939861. doi:10.1186/1471-2148-7-193
- ↑ Veeramachaneni, V. (1 de fevereiro de 2004). «Mammalian Overlapping Genes: The Comparative Perspective». Genome Research. 14 (2): 280–286. PMC 327103
 . PMID 14762064. doi:10.1101/gr.1590904
. PMID 14762064. doi:10.1101/gr.1590904
- ↑ Behura, Susanta K; Severson, David W (2013). «Overlapping genes of Aedes aegypti: evolutionary implications from comparison with orthologs of Anopheles gambiae and other insects». BMC Evolutionary Biology. 13 (1). 124 páginas. PMC 3689595
 . PMID 23777277. doi:10.1186/1471-2148-13-124
. PMID 23777277. doi:10.1186/1471-2148-13-124
- ↑ Murphy, Daniel N.; McLysaght, Aoife; Carmel, Liran (21 de novembro de 2012). «De Novo Origin of Protein-Coding Genes in Murine Rodents». PLOS ONE. 7 (11): e48650. Bibcode:2012PLoSO...748650M. PMC 3504067
 . PMID 23185269. doi:10.1371/journal.pone.0048650
. PMID 23185269. doi:10.1371/journal.pone.0048650
- ↑ Knowles, D. G.; McLysaght, A. (2 de setembro de 2009). «Recent de novo origin of human protein-coding genes». Genome Research. 19 (10): 1752–1759. PMC 2765279
 . PMID 19726446. doi:10.1101/gr.095026.109
. PMID 19726446. doi:10.1101/gr.095026.109
- ↑ Wright, Bradley W.; Ruan, Juanfang; Molloy, Mark P.; Jaschke, Paul R. (20 de novembro de 2020). «Genome Modularization Reveals Overlapped Gene Topology Is Necessary for Efficient Viral Reproduction». ACS Synthetic Biology (em inglês). 9 (11): 3079–3090. ISSN 2161-5063. PMID 33044064. doi:10.1021/acssynbio.0c00323
- ↑ Pradhan, Prajakta; Li, Wen; Kaur, Parjit (janeiro de 2009). «Translational Coupling Controls Expression and Function of the DrrAB Drug Efflux Pump». Journal of Molecular Biology (em inglês). 385 (3): 831–842. PMID 19063901. doi:10.1016/j.jmb.2008.11.027
- ↑ Jaschke, Paul R.; Lieberman, Erica K.; Rodriguez, Jon; Sierra, Adrian; Endy, Drew (dezembro de 2012). «A fully decompressed synthetic bacteriophage øX174 genome assembled and archived in yeast». Virology. 434 (2): 278–284. ISSN 0042-6822. PMID 23079106. doi:10.1016/j.virol.2012.09.020

- ↑ Krakauer, D. C.; Plotkin, J. B. (29 de janeiro de 2002). «Redundancy, antiredundancy, and the robustness of genomes». Proceedings of the National Academy of Sciences. 99 (3): 1405–1409. Bibcode:2002PNAS...99.1405K. ISSN 0027-8424. PMC 122203
 . PMID 11818563. doi:10.1073/pnas.032668599
. PMID 11818563. doi:10.1073/pnas.032668599
- ↑ Feiss, Michael; Fisher, R.A.; Crayton, M.A.; Egner, Carol (março de 1977). «Packaging of the bacteriophage λ chromosome: Effect of chromosome length». Virology. 77 (1): 281–293. ISSN 0042-6822. PMID 841861. doi:10.1016/0042-6822(77)90425-1
- ↑ Aoyama, A; Hayashi, M (setembro de 1985). «Effects of genome size on bacteriophage phi X174 DNA packaging in vitro.». Journal of Biological Chemistry. 260 (20): 11033–11038. ISSN 0021-9258. PMID 3161888. doi:10.1016/s0021-9258(17)39144-5

- ↑ Wu, Zhijian; Yang, Hongyan; Colosi, Peter (janeiro de 2010). «Effect of Genome Size on AAV Vector Packaging». Molecular Therapy. 18 (1): 80–86. ISSN 1525-0016. PMC 2839202
 . PMID 19904234. doi:10.1038/mt.2009.255
. PMID 19904234. doi:10.1038/mt.2009.255
- ↑ Vaidyanathan, Sriram; Baik, Ron; Chen, Lu; Bravo, Dawn T.; Suarez, Carlos J.; Abazari, Shayda M.; Salahudeen, Ameen A.; Dudek, Amanda M.; Teran, Christopher A.; Davis, Timothy H.; Lee, Ciaran M. (março de 2021). «Targeted replacement of full-length CFTR in human airway stem cells by CRISPR-Cas9 for pan-mutation correction in the endogenous locus». Molecular Therapy (em inglês). 30 (1): 223–237. PMC 8753290
 . PMID 33794364. doi:10.1016/j.ymthe.2021.03.023
. PMID 33794364. doi:10.1016/j.ymthe.2021.03.023
- ↑ Willis, Sara; Masel, Joanna (19 de julho de 2018). «Gene Birth Contributes to Structural Disorder Encoded by Overlapping Genes». Genetics. 210 (1): 303–313. ISSN 1943-2631. PMC 6116962
 . PMID 30026186. doi:10.1534/genetics.118.301249
. PMID 30026186. doi:10.1534/genetics.118.301249
- ↑ Pavesi, Angelo; Vianelli, Alberto; Chirico, Nicola; Bao, Yiming; Blinkova, Olga; Belshaw, Robert; Firth, Andrew; Karlin, David (19 de outubro de 2018). «Overlapping genes and the proteins they encode differ significantly in their sequence composition from non-overlapping genes». PLOS ONE. 13 (10): e0202513. Bibcode:2018PLoSO..1302513P. ISSN 1932-6203. PMC 6195259
 . PMID 30339683. doi:10.1371/journal.pone.0202513
. PMID 30339683. doi:10.1371/journal.pone.0202513
- ↑ Pavesi, Angelo; Magiorkinis, Gkikas; Karlin, David G. (15 de agosto de 2013). «Viral Proteins Originated De Novo by Overprinting Can Be Identified by Codon Usage: Application to the "Gene Nursery" of Deltaretroviruses». PLOS Computational Biology. 9 (8): e1003162. Bibcode:2013PLSCB...9E3162P. ISSN 1553-7358. PMC 3744397
 . PMID 23966842. doi:10.1371/journal.pcbi.1003162
. PMID 23966842. doi:10.1371/journal.pcbi.1003162
- ↑ «Supplemental Information 2: NCBI genome database accession information (PDF file).». dx.doi.org. doi:10.7717/peerj.6447/supp-2. Consultado em 3 de dezembro de 2021
- ↑ Ahmed, Niyaz (27 de março de 2009). «Faculty Opinions recommendation of The RAST Server: rapid annotations using subsystems technology.». doi:10.3410/f.1157743.618965

- ↑ Ben-Tal, Nir, ed. (23 de junho de 2017). «Decision letter: Deep transcriptome annotation enables the discovery and functional characterization of cryptic small proteins». doi:10.7554/elife.27860.082

- ↑ Bazzini, Ariel; Wu, Qiushuang (6 de março de 2020). «Faculty Opinions recommendation of Pervasive functional translation of noncanonical human open reading frames.». doi:10.3410/f.737484924.793572056

- ↑ Prensner, John R.; Enache, Oana M.; Luria, Victor; Krug, Karsten; Clauser, Karl R.; Dempster, Joshua M.; Karger, Amir; Wang, Li; Stumbraite, Karolina; Wang, Vickie M.; Botta, Ginevra (28 de janeiro de 2021). «Noncanonical open reading frames encode functional proteins essential for cancer cell survival». Nature Biotechnology. 39 (6): 697–704. ISSN 1087-0156. PMC 8195866
 . PMID 33510483. doi:10.1038/s41587-020-00806-2
. PMID 33510483. doi:10.1038/s41587-020-00806-2
- ↑ Cao, Xiongwen; Khitun, Alexandra; Luo, Yang; Na, Zhenkun; Phoodokmai, Thitima; Sappakhaw, Khomkrit; Olatunji, Elizabeth; Uttamapinant, Chayasith; Slavoff, Sarah A. (5 de março de 2020). «Alt-RPL36 downregulates the PI3K-AKT-mTOR signaling pathway by interacting with TMEM24». Nature Communications. 12 (1): 508. PMC 7820019
 . PMID 33479206. doi:10.1038/s41467-020-20841-6
. PMID 33479206. doi:10.1038/s41467-020-20841-6
- ↑ Jaschke, Paul R.; Dotson, Gabrielle A.; Hung, Kay S.; Liu, Diane; Endy, Drew (12 de novembro de 2019). «Definitive demonstration by synthesis of genome annotation completeness». Proceedings of the National Academy of Sciences. 116 (48): 24206–24213. Bibcode:2019PNAS..11624206J. ISSN 0027-8424. PMC 6883844
 . PMID 31719208. doi:10.1073/pnas.1905990116
. PMID 31719208. doi:10.1073/pnas.1905990116
