Sítio CpG

Os sítios CpG ou sítios CG são regiões do DNA onde um nucleotídeo citosina é seguido por um nucleotídeo guanina na sequência linear de bases ao longo de sua direção 5' → 3'. Os sítios CpG ocorrem com alta frequência em regiões genômicas chamadas ilhas CpG (ou ilhas CG).
As citosinas em dinucleotídeos CpG podem ser metiladas para formar 5-metilcitosinas. As enzimas que adicionam um grupo metil são chamadas de DNA metiltransferases . Nos mamíferos, 70% a 80% das citosinas CpG são metiladas.[1] A metilação da citosina dentro de um gene pode alterar sua expressão, um mecanismo que faz parte de um campo maior da ciência que estuda a regulação do gene que é chamado de epigenética . As citosinas metiladas frequentemente sofrem mutação para timinas .
Em humanos, cerca de 70% dos promotores localizados perto do sítio de início da transcrição de um gene (promotores proximais) contêm uma ilha CpG.[2][3]
Características CpG[editar | editar código-fonte]
Definição[editar | editar código-fonte]
CpG é uma abreviação de 5'—C—fosfato—G—3', ou seja, citosina e guanina separadas por apenas um grupo fosfato; fosfato liga quaisquer dois nucleosídeos juntos no DNA. A notação CpG é usada para distinguir esta sequência linear de fita simples do pareamento de bases CG de citosina e guanina para sequências de fita dupla. A notação CpG deve, portanto, ser interpretada como a citosina sendo 5 prime da base guanina. CpG não deve ser confundido com GpC, este significando que uma guanina é seguida por uma citosina na direção 5' → 3' de uma sequência de fita simples.
Sub-representação causada pela alta taxa de mutação[editar | editar código-fonte]
Os dinucleotídeos CpG têm sido observados há muito tempo com uma frequência muito menor na sequência de genomas de vertebrados do que seria esperado devido ao acaso. Por exemplo, no genoma humano, que tem um conteúdo de 42% de GC,[4] seria esperado que ocorresse, do tempo um par de nucleotídeos consistindo de citosina seguida de guanina. A frequência de dinucleotídeos CpG em genomas humanos é inferior a um quinto da frequência esperada.[5]
Esta sub-representação é uma consequência da alta taxa de mutação de sítios CpG metilados: a desaminação que ocorre espontaneamente de uma citosina metilada resulta em uma timina, e as bases desemparelhadas G:T resultantes são muitas vezes inadequadamente resolvidas para A:T; enquanto a desaminação da citosina não metilada resulta em uma uracila, que como base estranha é rapidamente substituída por uma citosina pelo mecanismo de reparo por excisão de base. A taxa de transição de C para T em sítios CpG metilados é ~10 vezes maior do que em sítios não metilados.[6][7][8][9]
Distribuição genômica[editar | editar código-fonte]
| Sítios CpG | Sítios GpC |
|---|---|

|

|
| Distribuição de sítios CpG (esquerda: em vermelho) e sítios GpC (direita: em verde) no gene APRT humano. CpG são mais abundantes na região a montante do gene, onde formam uma ilha CpG, enquanto GpC são distribuídos de forma mais uniforme. Os 5 exons do gene APRT são indicados (azul), e os códons de início (ATG) e de parada (TGA) são enfatizados (negrito azul). | |
Os dinucleotídeos CpG ocorrem frequentemente em ilhas CpG (veja a definição de ilhas CpG abaixo). Existem 28.890 ilhas CpG no genoma humano (50.267 se incluirmos ilhas CpG em sequências repetidas).[10] Isso está de acordo com as 28.519 ilhas CpG encontradas por Venter et al.[11] uma vez que a sequência do genoma estudada por Venter et al. não incluiu os interiores de elementos repetitivos altamente semelhantes e as regiões de repetição extremamente densas próximas aos centrômeros.[12] Como as ilhas CpG contêm múltiplas sequências de dinucleotídeos CpG, parece haver mais de 20 milhões de dinucleotídeos CpG no genoma humano.
Ilhas CpG[editar | editar código-fonte]

As ilhas CpG (ou ilhas CG) são regiões com alta frequência de sítios CpG. Embora as definições objetivas para ilhas CpG sejam limitadas, a definição formal usual é uma região com pelo menos 200 pb, uma porcentagem de GC maior que 50% e uma razão CpG observada-esperada maior que 60%. A "razão CpG observada para o esperado" pode ser derivada onde o observado é calculado como: e o esperado como [13] ou .[14]
Muitos genes em genomas de mamíferos têm ilhas CpG associadas ao início do gene[15] (regiões promotoras). Por isso, a presença de uma ilha CpG é utilizada para auxiliar na predição e anotação de genes.
Em genomas de mamíferos, as ilhas CpG são tipicamente de 300 a 3.000 pares de bases de comprimento e foram encontradas em aproximadamente 40% dos promotores de genes de mamíferos.[16] Mais de 60% dos genes humanos e quase todos os genes de manutenção têm seus promotores incorporados em ilhas CpG.[17] Dada a frequência das sequências de dois nucleotídeos do GC, o número de dinucleotídeos CpG é muito menor do que seria esperado.[14]
Um estudo de 2002 revisou as regras de previsão de ilhas CpG para excluir outras sequências genômicas ricas em GC, como repetições Alu . Com base em uma extensa pesquisa nas sequências completas dos cromossomos humanos 21 e 22, as regiões de DNA maiores que 500 bp foram encontradas com maior probabilidade de serem as "verdadeiras" ilhas CpG associadas às regiões 5' dos genes se tivessem um conteúdo de GC maior que 55%, e uma razão CpG observada-esperada de 65%.[18]
As ilhas CpG são caracterizadas por um conteúdo de dinucleotídeo CpG de pelo menos 60% do que seria estatisticamente esperado (~4-6%), enquanto o resto do genoma tem uma frequência CpG muito menor (~1%), um fenômeno chamado supressão de CG. Ao contrário dos sítios CpG na região codificadora de um gene, na maioria dos casos os sítios CpG nas ilhas CpG de promotores não são metilados se os genes forem expressos. Esta observação levou à especulação de que a metilação de sítios CpG no promotor de um gene pode inibir a expressão do gene. A metilação, juntamente com a modificação de histonas, é fundamental para a impressão genômica.[19] A maioria das diferenças de metilação entre tecidos, ou entre amostras normais e de câncer, ocorre a uma curta distância das ilhas CpG (nas "costa da ilha CpG"), e não nas próprias ilhas.[20]
As ilhas CpG ocorrem tipicamente no local de início da transcrição de genes, particularmente genes de manutenção, em vertebrados.[14] A base C (citosina) seguida imediatamente por uma base G (guanina), um CpG, é raro no DNA de vertebrados porque as citosinas em tal arranjo tendem a ser metiladas. Essa metilação ajuda a distinguir a fita de DNA recém-sintetizada da fita-mãe, o que auxilia nos estágios finais da revisão do DNA após a duplicação. No entanto, com o tempo, as citosinas metiladas tendem a se transformar em timinas devido à desaminação espontânea. Existe uma enzima especial em humanos (Timina-DNA glicosilase, ou TDG) que substitui especificamente T's de incompatibilidades T/G. No entanto, devido à raridade dos CpGs, teoriza-se que seja insuficientemente eficaz na prevenção de uma mutação possivelmente rápida dos dinucleotídeos. A existência de ilhas CpG geralmente é explicada pela existência de forças seletivas para teores relativamente altos de CpG, ou baixos níveis de metilação nessa área genômica, talvez tendo a ver com a regulação da expressão gênica. Um estudo de 2011 mostrou que a maioria das ilhas CpG são resultado de forças não seletivas.[21]
Metilação, silenciamento, câncer e envelhecimento[editar | editar código-fonte]

Ilhas CpG em promotores[editar | editar código-fonte]
Em humanos, cerca de 70% dos promotores localizados perto do sítio de início da transcrição de um gene (promotores proximais) contêm uma ilha CpG.[2][3]
Os elementos promotores distais também contêm frequentemente ilhas CpG. Um exemplo é o gene de reparo de DNA ERCC1, onde o elemento contendo a ilha CpG está localizado cerca de 5.400 nucleotídeos a montante do sítio de início da transcrição do gene ERCC1.[22] As ilhas CpG também ocorrem com frequência em promotores de RNAs não codificantes funcionais, como microRNAs.[23]
Metilação de ilhas CpG silencia genes de forma estável[editar | editar código-fonte]
Em humanos, a metilação do DNA ocorre na posição 5 do anel pirimidina dos resíduos de citosina dentro dos sítios CpG para formar 5-metilcitosinas. A presença de múltiplos sítios CpG metilados em ilhas CpG de promotores causa silenciamento estável de genes.[24] O silenciamento de um gene pode ser iniciado por outros mecanismos, mas isso geralmente é seguido pela metilação de sítios CpG na ilha CpG do promotor para causar o silenciamento estável do gene.[24]
Hiper/hipometilação do promotor CpG no câncer[editar | editar código-fonte]
Em cânceres, a perda de expressão de genes ocorre cerca de 10 vezes mais frequentemente por hipermetilação de ilhas CpG promotoras do que por mutações. Por exemplo, em um câncer colorretal, geralmente há cerca de 3 a 6 mutações de motorista e 33 a 66 mutações de carona ou passageiro.[25] Em contraste, em um estudo de tumores de cólon em comparação com a mucosa colônica adjacente de aparência normal, 1.734 ilhas CpG foram fortemente metiladas em tumores, enquanto essas ilhas CpG não foram metiladas na mucosa adjacente.[26] Metade das ilhas CpG estavam em promotores de genes codificadores de proteínas anotados,[26] sugerindo que cerca de 867 genes em um tumor de cólon perderam expressão devido à metilação da ilha CpG. Um estudo separado encontrou uma média de 1.549 regiões diferencialmente metiladas (hipermetiladas ou hipometiladas) nos genomas de seis cânceres de cólon (em comparação com a mucosa adjacente), das quais 629 estavam em regiões promotoras conhecidas de genes.[27] Um terceiro estudo encontrou mais de 2.000 genes metilados diferencialmente entre cânceres de cólon e mucosa adjacente. Usando a análise de enriquecimento do conjunto de genes, 569 dos 938 conjuntos de genes foram hipermetilados e 369 foram hipometilados em cânceres.[28] A hipometilação de ilhas CpG em promotores resulta em superexpressão dos genes ou conjuntos de genes afetados.
Um estudo de 2012[29] listou 147 genes específicos com promotores hipermetilados associados ao câncer de cólon, juntamente com a frequência com que essas hipermetilações foram encontradas em cânceres de cólon. Pelo menos 10 desses genes tinham promotores hipermetilados em quase 100% dos cânceres de cólon. Eles também indicaram 11 microRNAs cujos promotores foram hipermetilados em cânceres de cólon em frequências entre 50% e 100% dos cânceres. MicroRNAs (miRNAs) são pequenos RNAs endógenos que emparelham com sequências em RNAs mensageiros para direcionar a repressão pós-transcricional. Em média, cada microRNA reprime várias centenas de genes-alvo.[30] Assim, os microRNAs com promotores hipermetilados podem permitir a superexpressão de centenas a milhares de genes em um câncer.
A informação acima mostra que, em cânceres, a hiper/hipometilação do promotor CpG de genes e de microRNAs causa perda de expressão (ou, às vezes, expressão aumentada) de muito mais genes do que a mutação.
Genes de reparo de DNA com promotores hiper/hipometilados em cânceres[editar | editar código-fonte]
Os genes de reparo do DNA são frequentemente reprimidos em cânceres devido à hipermetilação das ilhas CpG dentro de seus promotores. Em carcinomas de células escamosas de cabeça e pescoço, pelo menos 15 genes de reparo de DNA têm frequentemente promotores hipermetilados; estes genes são XRCC1, MLH3, PMS1, RAD51B, XRCC3, RAD54B, BRCA1, SHFM1, GEN1, FANCE, FAAP20, SPRTN, SETMAR, HUS1 e PER1.[31] Cerca de dezessete tipos de câncer são frequentemente deficientes em um ou mais genes de reparo do DNA devido à hipermetilação de seus promotores.[32] Como exemplo, a hipermetilação do promotor do gene de reparo do DNA MGMT ocorre em 93% dos cânceres de bexiga, 88% dos cânceres de estômago, 74% dos cânceres de tireóide, 40%-90% dos cânceres colorretais e 50% dos cânceres cerebrais. A hipermetilação do promotor de LIG4 ocorre em 82% dos cânceres colorretais. A hipermetilação do promotor de NEIL1 ocorre em 62% dos cânceres de cabeça e pescoço e em 42% dos cânceres de pulmão de células não pequenas . A hipermetilação do promotor de ATM ocorre em 47% dos cânceres de pulmão de células não pequenas . A hipermetilação do promotor de MLH1 ocorre em 48% dos carcinomas de células escamosas de câncer de pulmão de células não pequenas. A hipermetilação do promotor de FANCB ocorre em 46% dos cânceres de cabeça e pescoço .
Por outro lado, os promotores de dois genes, PARP1 e FEN1, foram hipometilados e esses genes foram superexpressos em vários cânceres. PARP1 e FEN1 são genes essenciais na junção de extremidades mediada por microhomologia da via de reparo de DNA mutagênica e propensa a erros. Se essa via for superexpressa, o excesso de mutações que ela causa pode levar ao câncer. PARP1 é superexpresso em leucemias ativadas por tirosina quinase,[33] em neuroblastoma,[34] em tumores testiculares e de outras células germinativas,[35] e no sarcoma de Ewing,[36] FEN1 é superexpresso na maioria dos cânceres da mama,[37] próstata,[38] estômago,[39][40] neuroblastomas,[41] pancreático,[42] e pulmão.[43]
Danos ao DNA parecem ser a principal causa subjacente do câncer.[44][45] Se o reparo preciso do DNA for deficiente, os danos no DNA tendem a se acumular. Tal dano excessivo ao DNA pode aumentar os erros mutacionais durante a replicação do DNA devido à síntese de translesão propensa a erros. O excesso de dano ao DNA também pode aumentar as alterações epigenéticas devido a erros durante o reparo do DNA.[46][47] Tais mutações e alterações epigenéticas podem dar origem ao câncer (ver neoplasias malignas ). Assim, a hiper/hipometilação da ilha CpG nos promotores dos genes de reparo do DNA provavelmente é central para a progressão para o câncer.
Metilação de sítios CpG com a idade[editar | editar código-fonte]
Como a idade tem um forte efeito nos níveis de metilação do DNA em dezenas de milhares de locais CpG, pode-se definir um relógio biológico altamente preciso (referido como relógio epigenético ou idade de metilação do DNA) em humanos e chimpanzés.[48]
Sítios não metilados[editar | editar código-fonte]
Sítios de dinucleotídeos CpG não metilados podem ser detectados pelo receptor Toll-like 9 (TLR 9)[49] em células dendríticas plasmocitóides, monócitos, células natural killer (NK) e células B em humanos. Isso é usado para detectar infecção viral intracelular.
Papel dos sítios CpG na memória[editar | editar código-fonte]
Em mamíferos, DNA metiltransferases (que adicionam grupos metil a bases de DNA) exibem uma preferência de sequência para citosinas dentro de sítios CpG.[50] No cérebro do camundongo, 4,2% de todas as citosinas são metiladas, principalmente no contexto dos sítios CpG, formando 5mCpG.[51] A maioria dos sítios de 5mCpG hipermetilados aumenta a repressão de genes associados.[51]
Conforme revisado por Duke et al., a metilação do DNA do neurônio (repressão da expressão de genes particulares) é alterada pela atividade neuronal. A metilação do DNA do neurônio é necessária para a plasticidade sináptica ; é modificado pelas experiências; e a metilação e desmetilação ativa do DNA é necessária para a formação e manutenção da memória.[52]
Em 2016 Halder et al.[53] usando camundongos, e em 2017 Duke et al.[52] usando ratos, submeteu os roedores ao condicionamento contextual do medo, causando a formação de uma memória de longo prazo especialmente forte. Às 24 horas após o condicionamento, na região do hipocampo do cérebro de ratos, a expressão de 1.048 genes foi regulada negativamente (geralmente associada a 5mCpG em promotores de genes ) e a expressão de 564 genes foi regulada positivamente (muitas vezes associada à hipometilação de CpG sítios em promotores de genes). Às 24 horas após o treinamento, 9,2% dos genes no genoma do rato dos neurônios do hipocampo foram metilados diferencialmente. No entanto, embora o hipocampo seja essencial para aprender novas informações, ele não armazena informações. Nos experimentos com ratos de Halder, 1.206 genes metilados diferencialmente foram vistos no hipocampo uma hora após o condicionamento contextual do medo, mas essas metilações alteradas foram revertidas e não foram vistas após quatro semanas. Em contraste com a ausência de alterações de metilação de CpG a longo prazo no hipocampo, a metilação diferencial substancial de CpG pode ser detectada em neurônios corticais durante a manutenção da memória. Havia 1.223 genes metilados diferencialmente no córtex cingulado anterior de camundongos quatro semanas após o condicionamento contextual do medo.
A desmetilação em locais CpG requer atividade de ROS[editar | editar código-fonte]

Em células somáticas adultas, a metilação do DNA ocorre tipicamente no contexto de dinucleotídeos CpG (sítios CpG), formando 5-metilcitosina- pG, ou 5mCpG. As espécies reativas de oxigênio (ROS) podem atacar a guanina no sítio dinucleotídeo, formando 8-hidroxi-2'-desoxiguanosina (8-OHdG), e resultando em um sítio dinucleotídeo 5mCp-8-OHdG. A enzima de reparo de excisão de base OGG1 tem como alvo 8-OHdG e se liga à lesão sem excisão imediata. OGG1, presente em um sítio 5mCp-8-OHdG recruta TET1 e TET1 oxida o 5mC adjacente ao 8-OHdG. Isso inicia a desmetilação de 5mC.[54]

Conforme revisado em 2018,[55] em neurônios cerebrais, 5mC é oxidado pela família de dioxigenases de dez onze translocações (TET) (TET1, TET2, TET3) para gerar 5-hidroximetilcitosina (5hmC). Em etapas sucessivas, as enzimas TET hidroxilam ainda mais 5hmC para gerar 5-formilcitosina (5fC) e 5-carboxilcitosina (5caC). Timina-DNA glicosilase (TDG) reconhece as bases intermediárias 5fC e 5caC e extirpa a ligação glicosídica resultando em um sítio apirimidínico (sítio AP). Em uma via de desaminação oxidativa alternativa, 5hmC pode ser oxidativamente desaminado por desaminases do complexo de edição de mRNA de citidina desaminase/apolipoproteína B induzida por atividade (AID/APOBEC) para formar 5-hidroximetiluracil (5hmU) ou 5mC pode ser convertido em timina (Thy). 5hmU pode ser clivada por TDG, uracil-DNA glicosilase 1 monofuncional seletiva de fita simples (SMUG1), Nei-Like DNA Glicosilase 1 (NEIL1), ou proteína 4 de ligação a metil-CpG ( MBD4). Os sítios AP e as incompatibilidades T:G são então reparados por enzimas de reparo por excisão de base (BER) para produzir citosina (Cyt).
Duas revisões[56][57] resumem o grande corpo de evidências para o papel crítico e essencial de ROS na formação da memória. A desmetilação do DNA de milhares de sítios CpG durante a formação da memória depende da iniciação por ROS. Em 2016, Zhou et al.,[54] mostraram que as ROS têm um papel central na desmetilação do DNA .
TET1 é uma enzima chave envolvida na desmetilação de 5mCpG. No entanto, TET1 só é capaz de agir em 5mCpG se um ROS tiver atuado primeiro na guanina para formar 8-hidroxi-2'-desoxiguanosina (8-OHdG), resultando em um dinucleotídeo 5mCp-8-OHdG (veja a primeira figura neste seção).[54] Após a formação de 5mCp-8-OHdG, a enzima de reparo por excisão de base OGG1 liga-se à lesão de 8-OHdG sem excisão imediata. A adesão de OGG1 ao sítio 5mCp-8-OHdG recruta TET1, permitindo que TET1 oxide os 5mC adjacentes a 8-OHdG, como mostrado na primeira figura desta seção. Isso inicia a via de desmetilação mostrada na segunda figura desta seção.
A expressão alterada de proteínas em neurônios, controlada pela desmetilação dependente de ROS de sítios CpG em promotores de genes dentro do DNA do neurônio, é central para a formação da memória.[58]
Perda de CPG[editar | editar código-fonte]
A depleção de CPG foi observada no processo de metilação do DNA de Elementos Transponíveis (ETs), onde os ETs não são apenas responsáveis pela expansão do genoma, mas também pela perda de CpG no DNA do hospedeiro. Os ETs podem ser conhecidos como "centros de metilação", pelo qual, no processo de metilação, os ETs se espalham no DNA flanqueador uma vez no DNA do hospedeiro. Essa disseminação pode resultar subsequentemente em perda de CPG ao longo do tempo evolutivo. Tempos evolutivos mais antigos mostram uma maior perda de CpG no DNA flanqueador, em comparação com os tempos evolutivos mais jovens. Portanto, a metilação do DNA pode eventualmente levar à perda perceptível de sítios CpG no DNA vizinho.[59]
O tamanho do genoma e a razão CPG estão negativamente correlacionados[editar | editar código-fonte]
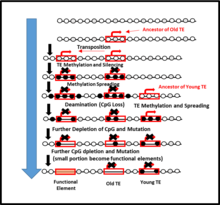
Estudos anteriores confirmaram a variedade de tamanhos de genomas em quantidade de espécies, onde invertebrados e vertebrados possuem genomas pequenos e grandes em comparação aos humanos. O tamanho do genoma está fortemente ligado ao número de elementos transponíveis. No entanto, existe uma correlação entre o número de metilação dos ETs versus a quantidade de CPG. Essa correlação negativa, consequentemente, causa depleção de CPG devido à metilação do DNA intergênico, que é principalmente atribuída à metilação dos ETs. No geral, isso contribui para uma quantidade notável de perda de CPG em diferentes espécies de genomas.[59]
Sequências de Alu como promotores de perda de CPG[editar | editar código-fonte]
Os elementos Alu são conhecidos como o tipo mais abundante de elementos transponíveis. Alguns estudos têm utilizado elementos Alu como forma de estudar a ideia de qual fator é responsável pela expansão do genoma. Os elementos Alu são ricos em CPG em uma quantidade maior de sequência, ao contrário de LINEs e ERVs. Alus pode funcionar como um centro de metilação, e a inserção em um DNA hospedeiro pode produzir metilação de DNA e provocar uma disseminação na área de DNA flanqueante. Essa disseminação é o motivo pelo qual há uma perda considerável de CPG e um aumento considerável na expansão do genoma.[59] No entanto, este é um resultado que é analisado ao longo do tempo, pois elementos Alus mais antigos apresentam maior perda de CPG em sítios de DNA vizinhos em comparação com os mais jovens.
Veja também[editar | editar código-fonte]
- TLR9, detector de sítios CpG não metilados
- Idade de metilação do DNA
Referências[editar | editar código-fonte]
- ↑ Jabbari K, Bernardi G (maio de 2004). «Cytosine methylation and CpG, TpG (CpA) and TpA frequencies». Gene. 333: 143–9. PMID 15177689. doi:10.1016/j.gene.2004.02.043
- ↑ a b Saxonov S, Berg P, Brutlag DL (2006). «A genome-wide analysis of CpG dinucleotides in the human genome distinguishes two distinct classes of promoters». Proc. Natl. Acad. Sci. U.S.A. 103 (5): 1412–7. Bibcode:2006PNAS..103.1412S. PMC 1345710
 . PMID 16432200. doi:10.1073/pnas.0510310103
. PMID 16432200. doi:10.1073/pnas.0510310103
- ↑ a b Deaton AM, Bird A (2011). «CpG islands and the regulation of transcription». Genes Dev. 25 (10): 1010–22. PMC 3093116
 . PMID 21576262. doi:10.1101/gad.2037511
. PMID 21576262. doi:10.1101/gad.2037511
- ↑ Lander, Eric S.; Linton, Lauren M.; Birren, Bruce; Nusbaum, Chad; Zody, Michael C.; Baldwin, Jennifer; Devon, Keri; Dewar, Ken; Doyle, Michael (15 de fevereiro de 2001). «Initial sequencing and analysis of the human genome». Nature (em inglês). 409 (6822): 860–921. Bibcode:2001Natur.409..860L. ISSN 1476-4687. PMID 11237011. doi:10.1038/35057062

- ↑ International Human Genome Sequencing Consortium (15 de fevereiro de 2001). «Initial sequencing and analysis of the human genome». Nature (em inglês). 409 (6822): 860–921. ISSN 0028-0836. PMID 11237011. doi:10.1038/35057062

- ↑ Hwang DG, Green P (2004). «Bayesian Markov chain Monte Carlo sequence analysis reveals varying neutral substitution patterns in mammalian evolution.». Proc Natl Acad Sci U S A. 101 (39): 13994–4001. Bibcode:2004PNAS..10113994H. PMC 521089
 . PMID 15292512. doi:10.1073/pnas.0404142101
. PMID 15292512. doi:10.1073/pnas.0404142101
- ↑ Walsh CP, Xu GL (2006). «Cytosine methylation and DNA repair.». Curr Top Microbiol Immunol. Current Topics in Microbiology and Immunology. 301: 283–315. ISBN 3-540-29114-8. PMID 16570853. doi:10.1007/3-540-31390-7_11
- ↑ Arnheim N, Calabrese P (2009). «Understanding what determines the frequency and pattern of human germline mutations.». Nat Rev Genet. 10 (7): 478–488. PMC 2744436
 . PMID 19488047. doi:10.1038/nrg2529
. PMID 19488047. doi:10.1038/nrg2529
- ↑ Ségurel L, Wyman MJ, Przeworski M (2014). «Understanding what determines the frequency and pattern of human germline mutations.». Annu Rev Genom Hum Genet. 15: 47–70. PMID 25000986. doi:10.1146/annurev-genom-031714-125740
- ↑ Lander ES, Linton LM, Birren B, Nusbaum C, Zody MC, Baldwin J, et al. (fevereiro de 2001). «Initial sequencing and analysis of the human genome». Nature. 409 (6822): 860–921. Bibcode:2001Natur.409..860L. PMID 11237011. doi:10.1038/35057062

- ↑ Venter JC, Adams MD, Myers EW, Li PW, Mural RJ, Sutton GG, et al. (fevereiro de 2001). «The sequence of the human genome». Science. 291 (5507): 1304–51. Bibcode:2001Sci...291.1304V. PMID 11181995. doi:10.1126/science.1058040

- ↑ Myers EW, Sutton GG, Smith HO, Adams MD, Venter JC (abril de 2002). «On the sequencing and assembly of the human genome». Proc. Natl. Acad. Sci. U.S.A. 99 (7): 4145–6. Bibcode:2002PNAS...99.4145M. PMC 123615
 . PMID 11904395. doi:10.1073/pnas.092136699
. PMID 11904395. doi:10.1073/pnas.092136699
- ↑ Gardiner-Garden M, Frommer M (1987). «CpG islands in vertebrate genomes». Journal of Molecular Biology. 196 (2): 261–282. PMID 3656447. doi:10.1016/0022-2836(87)90689-9
- ↑ a b c Saxonov S, Berg P, Brutlag DL (2006). «A genome-wide analysis of CpG dinucleotides in the human genome distinguishes two distinct classes of promoters». Proc Natl Acad Sci USA. 103 (5): 1412–1417. Bibcode:2006PNAS..103.1412S. PMC 1345710
 . PMID 16432200. doi:10.1073/pnas.0510310103
. PMID 16432200. doi:10.1073/pnas.0510310103
- ↑ Genetics: Analysis of Genes and Genomes
 6th ed. Missisauga: Jones & Bartlett, Canada. 2005. ISBN 978-0-7637-1511-3
6th ed. Missisauga: Jones & Bartlett, Canada. 2005. ISBN 978-0-7637-1511-3
- ↑ Fatemi M, Pao MM, Jeong S, Gal-Yam EN, Egger G, Weisenberger DJ, et al. (2005). «Footprinting of mammalian promoters: use of a CpG DNA methyltransferase revealing nucleosome positions at a single molecule level». Nucleic Acids Res. 33 (20): e176. PMC 1292996
 . PMID 16314307. doi:10.1093/nar/gni180
. PMID 16314307. doi:10.1093/nar/gni180
- ↑ Alberts, Bruce (18 de novembro de 2014). Molecular biology of the cell Sixth ed. New York, NY: [s.n.] 406 páginas. ISBN 978-0-8153-4432-2. OCLC 887605755
- ↑ Takai D, Jones PA (2002). «Comprehensive analysis of CpG islands in human chromosomes 21 and 22.». Proc Natl Acad Sci USA. 99 (6): 3740–5. Bibcode:2002PNAS...99.3740T. PMC 122594
 . PMID 11891299. doi:10.1073/pnas.052410099
. PMID 11891299. doi:10.1073/pnas.052410099
- ↑ Feil R, Berger F (2007). «Convergent evolution of genomic imprinting in plants and mammals». Trends Genet. 23 (4): 192–199. PMID 17316885. doi:10.1016/j.tig.2007.02.004
- ↑ Irizarry RA, Ladd-Acosta C, Wen B, Wu Z, Montano C, Onyango P, et al. (2009). «The human colon cancer methylome shows similar hypo- and hypermethylation at conserved tissue-specific CpG island shores». Nature Genetics. 41 (2): 178–186. PMC 2729128
 . PMID 19151715. doi:10.1038/ng.298
. PMID 19151715. doi:10.1038/ng.298
- ↑ Cohen N, Kenigsberg E, Tanay A (2011). «Primate CpG Islands Are Maintained by Heterogeneous Evolutionary Regimes Involving Minimal Selection». Cell. 145 (5): 773–786. PMID 21620139. doi:10.1016/j.cell.2011.04.024

- ↑ Chen HY, Shao CJ, Chen FR, Kwan AL, Chen ZP (2010). «Role of ERCC1 promoter hypermethylation in drug resistance to cisplatin in human gliomas». Int. J. Cancer. 126 (8): 1944–54. PMID 19626585. doi:10.1002/ijc.24772

- ↑ Kaur S, Lotsari-Salomaa JE, Seppänen-Kaijansinkko R, Peltomäki P (2016). «MicroRNA Methylation in Colorectal Cancer». Adv. Exp. Med. Biol. Advances in Experimental Medicine and Biology. 937: 109–22. ISBN 978-3-319-42057-8. PMID 27573897. doi:10.1007/978-3-319-42059-2_6
- ↑ a b Bird A (2002). «DNA methylation patterns and epigenetic memory». Genes Dev. 16 (1): 6–21. PMID 11782440. doi:10.1101/gad.947102

- ↑ Vogelstein B, Papadopoulos N, Velculescu VE, Zhou S, Diaz LA, Kinzler KW (2013). «Cancer genome landscapes». Science. 339 (6127): 1546–58. Bibcode:2013Sci...339.1546V. PMC 3749880
 . PMID 23539594. doi:10.1126/science.1235122
. PMID 23539594. doi:10.1126/science.1235122
- ↑ a b Illingworth RS, Gruenewald-Schneider U, Webb S, Kerr AR, James KD, Turner DJ, Smith C, Harrison DJ, Andrews R, Bird AP (2010). «Orphan CpG islands identify numerous conserved promoters in the mammalian genome». PLOS Genet. 6 (9): e1001134. PMC 2944787
 . PMID 20885785. doi:10.1371/journal.pgen.1001134
. PMID 20885785. doi:10.1371/journal.pgen.1001134
- ↑ Wei J, Li G, Dang S, Zhou Y, Zeng K, Liu M (2016). «Discovery and Validation of Hypermethylated Markers for Colorectal Cancer». Dis. Markers. 2016: 1–7. PMC 4963574
 . PMID 27493446. doi:10.1155/2016/2192853
. PMID 27493446. doi:10.1155/2016/2192853
- ↑ Beggs AD, Jones A, El-Bahrawy M, El-Bahwary M, Abulafi M, Hodgson SV, et al. (2013). «Whole-genome methylation analysis of benign and malignant colorectal tumours». J. Pathol. 229 (5): 697–704. PMC 3619233
 . PMID 23096130. doi:10.1002/path.4132
. PMID 23096130. doi:10.1002/path.4132
- ↑ Schnekenburger M, Diederich M (2012). «Epigenetics Offer New Horizons for Colorectal Cancer Prevention». Curr Colorectal Cancer Rep. 8 (1): 66–81. PMC 3277709
 . PMID 22389639. doi:10.1007/s11888-011-0116-z
. PMID 22389639. doi:10.1007/s11888-011-0116-z
- ↑ Friedman RC, Farh KK, Burge CB, Bartel DP (2009). «Most mammalian mRNAs are conserved targets of microRNAs». Genome Res. 19 (1): 92–105. PMC 2612969
 . PMID 18955434. doi:10.1101/gr.082701.108
. PMID 18955434. doi:10.1101/gr.082701.108
- ↑ Rieke DT, Ochsenreither S, Klinghammer K, Seiwert TY, Klauschen F, Tinhofer I, et al. (2016). «Methylation of RAD51B, XRCC3 and other homologous recombination genes is associated with expression of immune checkpoints and an inflammatory signature in squamous cell carcinoma of the head and neck, lung and cervix». Oncotarget. 7 (46): 75379–75393. PMC 5342748
 . PMID 27683114. doi:10.18632/oncotarget.12211
. PMID 27683114. doi:10.18632/oncotarget.12211
- ↑ Jin B, Robertson KD (2013). «DNA methyltransferases, DNA damage repair, and cancer». Adv. Exp. Med. Biol. Advances in Experimental Medicine and Biology. 754: 3–29. ISBN 978-1-4419-9966-5. PMC 3707278
 . PMID 22956494. doi:10.1007/978-1-4419-9967-2_1
. PMID 22956494. doi:10.1007/978-1-4419-9967-2_1
- ↑ Muvarak N, Kelley S, Robert C, Baer MR, Perrotti D, Gambacorti-Passerini C, et al. (2015). «c-MYC Generates Repair Errors via Increased Transcription of Alternative-NHEJ Factors, LIG3 and PARP1, in Tyrosine Kinase-Activated Leukemias». Mol. Cancer Res. 13 (4): 699–712. PMC 4398615
 . PMID 25828893. doi:10.1158/1541-7786.MCR-14-0422
. PMID 25828893. doi:10.1158/1541-7786.MCR-14-0422
- ↑ Newman EA, Lu F, Bashllari D, Wang L, Opipari AW, Castle VP (2015). «Alternative NHEJ Pathway Components Are Therapeutic Targets in High-Risk Neuroblastoma». Mol. Cancer Res. 13 (3): 470–82. PMID 25563294. doi:10.1158/1541-7786.MCR-14-0337

- ↑ Mego M, Cierna Z, Svetlovska D, Macak D, Machalekova K, Miskovska V, et al. (2013). «PARP expression in germ cell tumours». J. Clin. Pathol. 66 (7): 607–12. PMID 23486608. doi:10.1136/jclinpath-2012-201088
- ↑ Newman RE, Soldatenkov VA, Dritschilo A, Notario V (2002). «Poly(ADP-ribose) polymerase turnover alterations do not contribute to PARP overexpression in Ewing's sarcoma cells». Oncol. Rep. 9 (3): 529–32. PMID 11956622. doi:10.3892/or.9.3.529
- ↑ Singh P, Yang M, Dai H, Yu D, Huang Q, Tan W, Kernstine KH, Lin D, Shen B (2008). «Overexpression and hypomethylation of flap endonuclease 1 gene in breast and other cancers». Mol. Cancer Res. 6 (11): 1710–7. PMC 2948671
 . PMID 19010819. doi:10.1158/1541-7786.MCR-08-0269
. PMID 19010819. doi:10.1158/1541-7786.MCR-08-0269
- ↑ Lam JS, Seligson DB, Yu H, Li A, Eeva M, Pantuck AJ, Zeng G, Horvath S, Belldegrun AS (2006). «Flap endonuclease 1 is overexpressed in prostate cancer and is associated with a high Gleason score». BJU Int. 98 (2): 445–51. PMID 16879693. doi:10.1111/j.1464-410X.2006.06224.x
- ↑ Kim JM, Sohn HY, Yoon SY, Oh JH, Yang JO, Kim JH, et al. (2005). «Identification of gastric cancer-related genes using a cDNA microarray containing novel expressed sequence tags expressed in gastric cancer cells». Clin. Cancer Res. 11 (2 Pt 1): 473–82. PMID 15701830
- ↑ Wang K, Xie C, Chen D (2014). «Flap endonuclease 1 is a promising candidate biomarker in gastric cancer and is involved in cell proliferation and apoptosis». Int. J. Mol. Med. 33 (5): 1268–74. PMID 24590400. doi:10.3892/ijmm.2014.1682

- ↑ Krause A, Combaret V, Iacono I, Lacroix B, Compagnon C, Bergeron C, et al. (2005). «Genome-wide analysis of gene expression in neuroblastomas detected by mass screening» (PDF). Cancer Lett. 225 (1): 111–20. PMID 15922863. doi:10.1016/j.canlet.2004.10.035
- ↑ Iacobuzio-Donahue CA, Maitra A, Olsen M, Lowe AW, van Heek NT, Rosty C, et al. (2003). «Exploration of global gene expression patterns in pancreatic adenocarcinoma using cDNA microarrays». Am. J. Pathol. 162 (4): 1151–62. PMC 1851213
 . PMID 12651607. doi:10.1016/S0002-9440(10)63911-9
. PMID 12651607. doi:10.1016/S0002-9440(10)63911-9
- ↑ Nikolova T, Christmann M, Kaina B (2009). «FEN1 is overexpressed in testis, lung and brain tumors». Anticancer Res. 29 (7): 2453–9. PMID 19596913
- ↑ Kastan MB (2008). «DNA damage responses: mechanisms and roles in human disease: 2007 G.H.A. Clowes Memorial Award Lecture». Mol. Cancer Res. 6 (4): 517–24. PMID 18403632. doi:10.1158/1541-7786.MCR-08-0020

- ↑ Bernstein, C; Prasad, AR; Nfonsam, V; Bernstein, H. (2013). «Chapter 16: DNA Damage, DNA Repair and Cancer». In: Chen. New Research Directions in DNA Repair. [S.l.: s.n.] ISBN 978-953-51-1114-6
- ↑ O'Hagan HM, Mohammad HP, Baylin SB (2008). «Double strand breaks can initiate gene silencing and SIRT1-dependent onset of DNA methylation in an exogenous promoter CpG island». PLOS Genetics. 4 (8): e1000155. PMC 2491723
 . PMID 18704159. doi:10.1371/journal.pgen.1000155
. PMID 18704159. doi:10.1371/journal.pgen.1000155
- ↑ Cuozzo C, Porcellini A, Angrisano T, et al. (julho de 2007). «DNA damage, homology-directed repair, and DNA methylation». PLOS Genetics. 3 (7): e110. PMC 1913100
 . PMID 17616978. doi:10.1371/journal.pgen.0030110
. PMID 17616978. doi:10.1371/journal.pgen.0030110
- ↑ Horvath S (2013). «DNA methylation age of human tissues and cell types». Genome Biology. 14 (10): R115. PMC 4015143
 . PMID 24138928. doi:10.1186/gb-2013-14-10-r115
. PMID 24138928. doi:10.1186/gb-2013-14-10-r115
- ↑ Ramirez-Ortiz ZG, Specht CA, Wang JP, Lee CK, Bartholomeu DC, Gazzinelli RT, Levitz SM (2008). «Toll-like receptor 9-dependent immune activation by unmethylated CpG motifs in Aspergillus fumigatus DNA». Infect. Immun. 76 (5): 2123–2129. PMC 2346696
 . PMID 18332208. doi:10.1128/IAI.00047-08
. PMID 18332208. doi:10.1128/IAI.00047-08
- ↑ Ziller MJ, Müller F, Liao J, Zhang Y, Gu H, Bock C, et al. (dezembro de 2011). «Genomic distribution and inter-sample variation of non-CpG methylation across human cell types». PLOS Genet. 7 (12): e1002389. PMC 3234221
 . PMID 22174693. doi:10.1371/journal.pgen.1002389
. PMID 22174693. doi:10.1371/journal.pgen.1002389
- ↑ a b Fasolino M, Zhou Z (maio de 2017). «The Crucial Role of DNA Methylation and MeCP2 in Neuronal Function». Genes (Basel). 8 (5). 141 páginas. PMC 5448015
 . PMID 28505093. doi:10.3390/genes8050141
. PMID 28505093. doi:10.3390/genes8050141
- ↑ a b Duke CG, Kennedy AJ, Gavin CF, Day JJ, Sweatt JD (julho de 2017). «Experience-dependent epigenomic reorganization in the hippocampus». Learn. Mem. 24 (7): 278–288. PMC 5473107
 . PMID 28620075. doi:10.1101/lm.045112.117
. PMID 28620075. doi:10.1101/lm.045112.117
- ↑ Halder R, Hennion M, Vidal RO, Shomroni O, Rahman RU, Rajput A, et al. (janeiro de 2016). «DNA methylation changes in plasticity genes accompany the formation and maintenance of memory». Nat. Neurosci. 19 (1): 102–10. PMC 4700510
 . PMID 26656643. doi:10.1038/nn.4194
. PMID 26656643. doi:10.1038/nn.4194
- ↑ a b c Zhou X, Zhuang Z, Wang W, He L, Wu H, Cao Y, Pan F, Zhao J, Hu Z, Sekhar C, Guo Z (setembro de 2016). «OGG1 is essential in oxidative stress induced DNA demethylation». Cell. Signal. 28 (9): 1163–71. PMID 27251462. doi:10.1016/j.cellsig.2016.05.021
- ↑ Bayraktar G, Kreutz MR (2018). «The Role of Activity-Dependent DNA Demethylation in the Adult Brain and in Neurological Disorders». Front Mol Neurosci. 11. 169 páginas. PMC 5975432
 . PMID 29875631. doi:10.3389/fnmol.2018.00169
. PMID 29875631. doi:10.3389/fnmol.2018.00169
- ↑ Massaad CA, Klann E (maio de 2011). «Reactive oxygen species in the regulation of synaptic plasticity and memory». Antioxid. Redox Signal. 14 (10): 2013–54. PMC 3078504
 . PMID 20649473. doi:10.1089/ars.2010.3208
. PMID 20649473. doi:10.1089/ars.2010.3208
- ↑ Beckhauser TF, Francis-Oliveira J, De Pasquale R (2016). «Reactive Oxygen Species: Physiological and Physiopathological Effects on Synaptic Plasticity». J Exp Neurosci. 10 (Suppl 1): 23–48. PMC 5012454
 . PMID 27625575. doi:10.4137/JEN.S39887
. PMID 27625575. doi:10.4137/JEN.S39887
- ↑ Day JJ, Sweatt JD (novembro de 2010). «DNA methylation and memory formation». Nat. Neurosci. 13 (11): 1319–23. PMC 3130618
 . PMID 20975755. doi:10.1038/nn.2666
. PMID 20975755. doi:10.1038/nn.2666
- ↑ a b c Zhou, Wanding; Liang, Gangning; Molloy, Peter L.; Jones, Peter A. (2020). «DNA methylation enables transposable element-driven genome expansion». Proceedings of the National Academy of Sciences of the United States of America. 117 (32): 19359–19366. ISSN 1091-6490. PMC 7431005
 . PMID 32719115. doi:10.1073/pnas.1921719117
. PMID 32719115. doi:10.1073/pnas.1921719117
- ↑ Zhou, Wanding; Liang, Gangning; Molloy, Peter L.; Jones, Peter A. (11 de agosto de 2020). «DNA methylation enables transposable element-driven genome expansion». Proceedings of the National Academy of Sciences of the United States of America. 117 (32): 19359–19366. ISSN 1091-6490. PMC 7431005
 . PMID 32719115. doi:10.1073/pnas.1921719117
. PMID 32719115. doi:10.1073/pnas.1921719117




