Haplogrupo L2 (mtDNA)
O haplogrupo L2 é um haplogrupo de DNA mitocondrial humano (mtDNA) com ampla distribuição moderna, particularmente na África Subequatorial. Seu subclado L2a é um cluster de mtDNA bastante frequente e amplamente distribuído no continente, bem como entre afro-americanos.
Origem
[editar | editar código-fonte]L2 é uma linhagem comum na África. Acredita-se que tenha evoluído entre 87.000 e 107.000 anos atrás[1] ou aprox. 90.000 anos até o presente. Sua idade e ampla distribuição e diversidade em todo o continente tornam seu ponto de origem exato na África difícil de rastrear com confiança.[2] Vários haplótipos L2 observados em guineenses e outras populações da África Ocidental compartilharam correspondências genéticas com a África Oriental e a África do Norte .[3] Uma origem para L2b, L2c, L2d e L2e na África Ocidental ou Central parece provável.[2] A diversidade inicial de L2 pode ser observada em todo o continente africano, mas, como podemos ver abaixo na seção Subclados, a maior diversidade é encontrada na África Ocidental . A maioria dos subclados está amplamente confinada à África Ocidental e Centro-Ocidental.[4]
De acordo com um estudo de 2015, "os resultados mostram que as linhagens na África Austral se agrupam com linhagens da África Ocidental/Central em uma escala de tempo recente, enquanto as linhagens orientais parecem ser substancialmente mais antigas. Três momentos de expansão de origem da África Central estão associados a L2: uma migração a 70-50 mil anos para a África Oriental ou Austral, movimentos pós-glaciais 15-10 mil anos para a África Oriental; e a Expansão Bantu para o sul nos últimos 5 mil anos. A população complementar e as análises de filogeografia L0a indicam nenhuma forte evidência de fluxo gênico de mtDNA entre as populações do leste e do sul durante o movimento posterior, sugerindo baixa mistura entre as populações da África Oriental e os migrantes Bantu. Isso implica que, pelo menos nos estágios iniciais, a expansão bantu foi principalmente uma difusão dêmica (do próprio povo) com pouca incorporação de populações locais".[5]
Distribuição
[editar | editar código-fonte]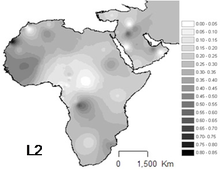
L2 é o haplogrupo mais comum na África e tem sido observado em todo o continente. É encontrado em aproximadamente um terço dos africanos e seus descendentes recentes.
A maior frequência ocorre entre os pigmeus Mbuti (64%).[6] Presença importante na África Ocidental, especialmente no Senegal (43-54%).[3] Também importante em populações não-bantu da África Oriental (44%),[7] no Sudão e Moçambique .
É particularmente abundante no Chade e no Kanembou (38% da amostra), mas também é relativamente frequente em árabes nômades (33%) [Cerny et al. 2007][2] e o povo Akan (~33%) [8]
Subclados
[editar | editar código-fonte]
|
L2 tem cinco subhaplogrupos principais: L2a, L2b, L2c, L2d e L2e. Dessas linhagens, o subclado mais comum é L2a, encontrado tanto na África quanto no Levante .
O haplogrupo L2 foi observado entre os espécimes no cemitério da ilha em Kulubnarti, Sudão, que datam do período cristão primitivo (550-800 d.C).[9]
Haplogrupo L2a
[editar | editar código-fonte]L2a é difundido na África e o Haplogrupo mais comum e amplamente distribuído da África Subsaariana e, também, é um pouco frequente em 19% nas Américas entre descendentes de africanos (Salas et al., 2002). O L2a surgiu a aproximadamente 48.000 anos atrás.
É particularmente abundante no Chade (38% da amostra; 33% de L2 indiferenciado entre os árabes do Chade,[10] ) e em populações não-bantu da África Oriental ( Quênia, Uganda e Tanzânia ) em 38%.[7] Cerca de 33% em Moçambique[11] e 32% no Gana .[8]
Este subclado é caracterizado por mutações em 2789, 7175, 7274, 7771, 11914, 13803, 14566 e 16294. Representa 52% do total de L2 e é o único subclado de L2 a ser difundido em toda a África.[12]
A ampla distribuição de L2a e a diversidade dificultam a identificação de uma origem geográfica. O principal quebra-cabeça é o quase onipresente Haplogrupo L2a, que pode ter se espalhado para leste e oeste ao longo do Corredor do Sahel no norte da África após o Último Máximo Glacial, ou as origens dessas expansões podem ser anteriores, no início da Idade da Pedra Posterior ∼ 40.000 anos atrás.[2][12]
Na África Oriental L2a foi encontrado 15% no Vale do Nilo – Núbia, 5% dos egípcios, 14% dos falantes de cuchita, 15% do povo semita Amara, 10% do Gurage, 6% de tigrínios, 13% dos etíopes e 5% dos iemenitas .[11]
Haplogrupo L2a também aparece no norte da África, com a maior frequência de 20% Tuareg, Fulani (14%). Encontrado também entre alguns árabes da Argélia, é encontrado em 10% entre os árabes marroquinos, alguns berberes marroquinos e berberes tunisianos. (watson 1997) et al., (vigilant 1991) et al. 1991.
Em pacientes que recebem a droga estavudina para tratar o HIV, o Haplogrupo L2a está associado a uma menor probabilidade de neuropatia periférica como efeito colateral.[13]
Haplogrupo L2a1
[editar | editar código-fonte]L2a pode ser dividido em L2a1, abrigando a transição em 16309 (Salas et al. 2002).
Este subclado é observado em frequências variadas na África Ocidental entre os Malinke, Wolof e outros; entre os norte-africanos ; no Sahel, entre os Hausa, Fulbe e outros; na África Central entre os Bamileke, Fali e outros; na África do Sul entre a família Khoisan, incluindo os falantes Khwe e Bantu; e na África Oriental entre os Kikuyus do Quênia .
Todos os clados L2 presentes na Etiópia são derivados principalmente dos dois subclados, L2a1 e L2b. L2a1 é definido por mutações em 12693, 15784 e 16309. A maioria das sequências L2a1 etíopes compartilham mutações em nps 16189 e 16309. No entanto, enquanto a maioria (26 de 33) afro-americanos compartilham sequências completas do Haplogrupo L2a podem ser particionadas em quatro subclados por substituições em nps L2a1e-3495, L2a1a-3918, L2a1f-5581 e L2a1i-15229. Nenhuma dessas sequências foi observada em amostras etíopes 16309 L2a1. (Salas 2002) et al.
Haplogrupo L2a1 também foi observado entre os Mahra (4,6%).[14]
O haplogrupo L2a1 foi encontrado em fósseis antigos associados à cultura pré-cerâmica neolítica em Tell Halula, na Síria .[15] Um espécime escavado no sítio neolítico Savanna Pastoral de Luxmanda, na Tanzânia, também carregava o clado L2a1. A análise de agrupamento de mistura indicou ainda que o indivíduo tinha ascendência significativa do antigo Levante, confirmando os laços ancestrais entre os criadores do Neolítico Pastoral de Savana e o Neolítico Pré-Cerâmico.[16]
Haplogrupo L2a1a
[editar | editar código-fonte]O subclado L2a1a é definido por substituições em 3918, 5285, 15244 e 15629. Existem dois clusters L2a que estão bem representados no sudeste da África, L2a1a e L2a1b, ambos definidos por transições em posições HVS-I bastante estáveis. Ambos parecem ter origem na África Ocidental ou no Noroeste da África (como indicado pela distribuição de tipos correspondentes ou vizinhos), e ter sofrido uma expansão dramática no Sudeste da África ou em uma população ancestral dos atuais africanos do Sudeste.
A dispersão muito recente nos subclados L2a1a e L2a2 sugerem uma assinatura para as expansões Bantu, como também proposto por Pereira et al. (2001).
L2a1a é definido por uma mutação em 16286. O candidato a fundador da L2a1a data de 2.700 (SE 1.200) anos atrás. (Pereira et al. 2001). No entanto, L2a1a, conforme definido por uma substituição em (np 16286) (Salas et al. 2002), é agora suportado por um marcador de região de codificação (np 3918) (fig. 2A) e foi encontrado em quatro das seis linhagens L2a1 iemenitas . L2a1a ocorre com maior frequência no sudeste da África (Pereira et al. 2001; Salas et al. 2002). Tanto o haplótipo fundador frequente quanto as linhagens derivadas (com a mutação 16092) encontrados entre os iemenitas têm correspondências exatas nas sequências de Moçambique (Pereira et al. 2001; Salas et al. 2002). L2a1a também ocorre em menor frequência no noroeste da África, entre os Maure e Bambara do Mali e da Mauritânia .[17] (Rando et al. 1998; Maca-Meyer et al. 2003)
Haplogrupo L2a1a1
[editar | editar código-fonte]L2a1a1 é definido pelos marcadores 6152C, 15391T, 16368C
Haplogrupo L2a1b
[editar | editar código-fonte]L2a1b é definido por substituições em 16189 e 10143. 16192 também é comum em L2a1b e L2a1c; aparece no norte da África no Egito, também aparece no sudeste da África e, portanto, também pode ser um marcador da expansão bantu .[2]
Haplogrupo L2a1c
[editar | editar código-fonte]L2a1c frequentemente compartilha a mutação 16189 com L2a1b, mas tem seus próprios marcadores em 3010 e 6663. 16192 também é comum em L2a1b e L2a1c; ele aparece no sudeste da África, bem como na África Oriental.[18] Isso sugere alguma diversificação deste clado in situ.
As posições T16209C C16301T C16354T no topo de L2a1 definem um pequeno subclado, apelidado de L2a1c por Kivisild et al. (2004, Figura 3) (ver também Figura 6 em Salas et al. 2002), que aparece principalmente na África Oriental (por exemplo, Sudão, Núbia, Etiópia ), entre os Turkana e a África Ocidental (por exemplo, Kanuri ).
Na Bacia do Chade, foram identificados quatro tipos diferentes de L2a1c, um ou dois passos mutacionais dos tipos da África Oriental e Ocidental. (Kivisild et al.) 2004.[18] (citação na página 9 ou 443) [19][20]
Haplogrupo L2a1c1
[editar | editar código-fonte]L2a1c1 tem origem norte-africana.[21] É definido pelos marcadores 198, 930, 3308, 8604, 16086. É observado na Tunísia sefardita, ashkenazi, judeus, hebreus, marroquinos, egípcios, núbios e iemenitas.
Haplogrupo L2a1f
Khosian, Zâmbia, Madagáscar
Haplogrupo L2a1k
[editar | editar código-fonte]L2a1k é definido pelos marcadores G6722A e T12903C. Foi descrito anteriormente como um subclado L2a1a específico da Europa e detectado em tchecos e eslovacos .[22]
Haplogrupo L2a1l2a
[editar | editar código-fonte]L2a1l2a é reconhecido como um haplogrupo "específico Ashkenazi ", visto entre os judeus Ashkenazi com ascendência na Europa Central e Oriental. Também foi detectado em pequenos números em populações polonesas ostensivamente não judaicas, onde se presume que tenha vindo da mistura Ashkenazi.[23] No entanto, este haplótipo constitui apenas uma proporção muito pequena de linhagens mitocondriais Ashkenazi; vários estudos (incluindo o de Behar) colocaram sua incidência entre 1,4-1,6%.
Haplogrupo L2a2
[editar | editar código-fonte]L2a2 é característica dos pigmeus Mbuti .[6]
Haplogrupo L2b'c
[editar | editar código-fonte]L2b'c provavelmente evoluiu cerca de 62.000 anos atrás.
Haplogrupo L2b
[editar | editar código-fonte]Este subclado é predominantemente encontrado na África Ocidental, mas está espalhado por toda a África.[24]
Haplogrupo L2c
[editar | editar código-fonte]L2c é mais frequente na África Ocidental e pode ter surgido lá.[12] Especialmente presente no Senegal com 39%, Cabo Verde 16% e Guiné-Bissau 16%.[3]
Haplogrupo L2d
[editar | editar código-fonte]L2d é mais frequente na África Ocidental, onde pode ter surgido.[12] Também é encontrado no Iêmen, Moçambique e Sudão.[11]
Haplogrupo L2e
[editar | editar código-fonte]L2e (antigo L2d2) é típico na África Ocidental .[2] Também é encontrado na Tunísia,[25] e entre os Mandingas da Guiné-Bissau e afro-americanos.[24]
Árvore
[editar | editar código-fonte]
Esta árvore filogenética de subclades de haplogrupo L2 é baseada no artigo de Mannis van Oven e Manfred Kayser Atualizado árvore filogenética abrangente da variação global do DNA mitocondrial humano[26] e pesquisas subsequentes publicadas.
- Ancestral comum mais recente (MRCA)
- L1'2'3'4'5'6
- L2'3'4'6
- L2
- L2a'b'c'd
- L2a
- L2a1
- L2a1a
- L2a1a1
- L2a1a2
- L2a1a2a
- L2a1a2a1
- L2a1a2b
- L2a1a2a
- L2a1a3
- 16189 (16192)
- L2a1b
- L2a1b1
- L2a1f
- L2a1f1
- L2a1b
- 143
- L2a1c
- L2a1c1
- L2a1c2
- L2a1c3
- L2a1c4
- L2a1d
- L2a1e
- L2a1e1
- L2a1h
- 16189
- L2a1i
- L2a1j
- L2a1k
- 16192
- L2a1l
- L2a1l1
- L2a1l1a
- L2a1l2
- L2a1l1
- L2a1l
- L2a1c
- L2a1a
- L2a2
- L2a2a
- L2a2a1
- L2a2b
- L2a2b1
- L2a2a
- L2a1
- L2b'c
- L2b
- L2b1
- L2b1a
- L2b1a2
- L2b1a3
- L2b1a
- L2b1
- L2c
- L2c2
- L2c2a
- L2c3
- L2c2
- L2b
- L2d
- L2d1
- L2d1a
- L2d1
- L2a
- L2e
- L2a'b'c'd
- L2
- L2'3'4'6
- L1'2'3'4'5'6
Veja também
[editar | editar código-fonte]- Teste de DNA genealógico
- Genealogia genética
- Genética mitocondrial humana
- Genética de populações
- Haplogrupos de DNA mitocondrial humano
Referências
[editar | editar código-fonte]- ↑ Tishkoff et al., Whole-mtDNA Genome Sequence Analysis of Ancient African Lineages, Molecular Biology and Evolution, vol. 24, no. 3 (2007), pp. 757–768.
- ↑ a b c d e f Salas, Antonio et al., The Making of the African mtDNA Landscape, American Journal of Human Genetics, vol. 71, no. 5 (2002), pp. 1082–1111.
- ↑ a b c Rosa, Alexandra; Brehm, A; Kivisild, T; Metspalu, E; Villems, R; et al. (2004). «MtDNA Profile of West Africa Guineans: Towards a Better Understanding of the Senegambia Region». Annals of Human Genetics. 68 (Pt 4): 340–352. PMID 15225159. doi:10.1046/j.1529-8817.2004.00100.x
|hdl-access=requer|hdl=(ajuda) - ↑ Atlas of the Human Journey: Haplogroup L2 Arquivado em 2011-10-06 no Wayback Machine The Genographic Project, National Geographic.
- ↑ Silva, Marina; Alshamali, Farida; Silva, Paula; Carrilho, Carla; Mandlate, Flávio; Jesus Trovoada, Maria; Černý, Viktor; Pereira, Luísa; Soares, Pedro (2015). «60,000 years of interactions between Central and Eastern Africa documented by major African mitochondrial haplogroup L2». Scientific Reports. 5. 12526 páginas. Bibcode:2015NatSR...512526S. PMC 4515592
 . PMID 26211407. doi:10.1038/srep12526
. PMID 26211407. doi:10.1038/srep12526
- ↑ a b Quintana-Murci et al. 2008.
- ↑ a b Sadie Anderson-Mann 2006, Phylogenetic and phylogeographic analysis of African mitochondrial DNA variation.
- ↑ a b Veeramah, Krishna R et al 2010, Little genetic differentiation as assessed by uniparental markers in the presence of substantial language variation in peoples of the Cross River region of Nigeria.
- ↑ Sirak, Kendra; Frenandes, Daniel; Novak, Mario; Van Gerven, Dennis; Pinhasi, Ron (2016). «Abstract Book of the IUAES Inter-Congress 2016 – A community divided? Revealing the community genome(s) of Medieval Kulubnarti using next-generation sequencing». IUAES. Abstract Book of the Iuaes Inter-Congress 2016. 115 páginas
- ↑ Cerezo, María; et al. (2011). «New insights into the Lake Chad Basin population structure revealed by high-throughput genotyping of mitochondrial DNA coding SNPs». PLOS ONE. 6 (4): e18682. Bibcode:2011PLoSO...618682C. CiteSeerX 10.1.1.291.8871
 . PMC 3080428
. PMC 3080428 . PMID 21533064. doi:10.1371/journal.pone.0018682
. PMID 21533064. doi:10.1371/journal.pone.0018682
- ↑ a b c Toomas Kivisild et al., Ethiopian Mitochondria DNA Heritage: Tracking Gene Flow Across and Around the Gate of Tears, American Journal of Human Genetics, vol. 75, no. 5 (November 2004), pp. 752–770.
- ↑ a b c d Antonio Torroni et al., Do the Four Clades of the mtDNA Haplogroup L2 Evolve at Different Rates?, American Journal of Human Genetics, vol. 69 (2001), pp. 348–1356.
- ↑ Kampira, E; Kumwenda, J; van Oosterhout, JJ; Dandara, C (agosto de 2013). «Mitochondrial DNA subhaplogroups L0a2 and L2a modify susceptibility to peripheral neuropathy in malawian adults on stavudine containing highly active antiretroviral therapy». J Acquir Immune Defic Syndr. 63 (5): 647–652. PMC 3815091
 . PMID 23614993. doi:10.1097/QAI.0b013e3182968ea5
. PMID 23614993. doi:10.1097/QAI.0b013e3182968ea5
- ↑ Non, Amy. «ANALYSES OF GENETIC DATA WITHIN AN INTERDISCIPLINARY FRAMEWORK TO INVESTIGATE RECENT HUMAN EVOLUTIONARY HISTORY AND COMPLEX DISEASE» (PDF). University of Florida. Consultado em 2 de novembro de 2016
- ↑ Manco, Jean (2013). Ancestral Journeys: The Peopling of Europe from the First Venturers to the Vikings. [S.l.]: Thames & Hudson. ISBN 978-0500771822. Consultado em 29 de setembro de 2017
- ↑ Skoglund; et al. (21 de setembro de 2017). «Reconstructing Prehistoric African Population Structure». Cell. 171 (1): 59–71. PMC 5679310
 . PMID 28938123. doi:10.1016/j.cell.2017.08.049
. PMID 28938123. doi:10.1016/j.cell.2017.08.049
- ↑ González, A. M. et al 2006, Mitochondrial DNA Variation in Mauritania and Mali and their Genetic Relationship to Other Western Africa Populations
- ↑ a b «Wiley Online Library | Scientific research articles, journals, books, and reference works». Consultado em 19 de maio de 2009. Arquivado do original em 5 de agosto de 2010
- ↑ Cˇerny, V et al 2006, A Bidirectional Corridor in the Sahel-Sudan Belt and the Distinctive Features of the Chad Basin Populations: A History Revealed by the Mitochondrial DNA Genome.
- ↑ Lascaro, Daniela; castelhana, Stefano; Gasparre, Giuseppe; Romeo, Giovanni; Saccone, Cecilia; Attimonelli, Marcella (2008). «The RHNumtS compilation: Features and bioinformatics approaches to locate and quantify Human NumtS». BMC Genomics. 9: 267. PMC 2447851
 . PMID 18522722. doi:10.1186/1471-2164-9-267
. PMID 18522722. doi:10.1186/1471-2164-9-267
- ↑ Lascaro, Daniela; Castellana, Stefano; Gasparre, Giuseppe; Romeo, Giovanni; Saccone, Cecilia; Attimonelli, Marcella (2008). «The RHNumtS compilation: Features and bioinformatics approaches to locate and quantify Human NumtS». BMC Genomics. 9: 267. PMC 2447851
 . PMID 18522722. doi:10.1186/1471-2164-9-267
. PMID 18522722. doi:10.1186/1471-2164-9-267
- ↑ Boris A Malyarchuk, Miroslava Derenko, Maria Perkova, Tomasz Grzybowski, Tomas Vanecek and Jan Lazur, Reconstructing the phylogeny of African mitochondrial DNA lineages in Slavs, European Journal of Human Genetics, vol. 16 (2008), pp. 1091–1096
- ↑ Marta Mielnik-Sikorska, Patrycja Daca, Boris Malyarchuk, Miroslava Derenko, Katarzyna Skonieczna, Maria Perkova, Tadeusz Dobosz, Tomasz Grzybowski, The History of Slavs Inferred from Complete Mitochondrial Genome Sequences PLOSOne January 14, 2013; 10.1371/journal.pone.0054360
- ↑ a b Behar et al 2008b, The Dawn of Human Matrilineal Diversity Am J Hum Genet. 2008 May 9; 82(5): 1130–1140
- ↑ Costa MD et al 2009, Data from complete mtDNA sequencing of Tunisian centenarians: testing haplogroup association and the "golden mean" to longevity.
- ↑ van Oven, Mannis; Manfred Kayser (13 de outubro de 2008). «Updated comprehensive phylogenetic tree of global human mitochondrial DNA variation». Human Mutation. 30 (2): E386–E394. PMID 18853457. doi:10.1002/humu.20921

Ligações externas
[editar | editar código-fonte]- Ian Logan's Haplogroup L2. Mitochondrial DNA Site
- Ian Logan's L2bcd. Mitochondrial DNA Site
- Mannis van Oven's PhyloTree.org – mtDNA subtree L
- Spread of Haplogroup L2, from National Geographic
