Herpesvírus humano 6
| Herpesvírus humano 6 | |||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Classificação científica | |||||||||||||||||
| |||||||||||||||||
Herpesvírus humano 6 (HHV-6) é o nome coletivo comum para o Betaherpesvirus humano 6A (HHV-6A) e o Betaherpesvírus humano 6B (HHV-6B). Esses vírus intimamente relacionados são dois dos nove herpesvírus conhecidos por possuir seres humanos como hospedeiro principal.[1]
HHV-6A e HHV-6B são vírus de DNA de fita dupla na subfamília Betaherpesvirinae e do gênero Roseolovirus. HHV-6A e HHV-6B infectam quase todas as populações humanas que foram testadas.[2]
O HHV-6A foi descrito como mais neurovirulento,[3] e, como tal, é mais frequentemente encontrado em pacientes com doenças neuroinflamatórias, como esclerose múltipla.[4] Os níveis de HHV-6 (e HHV-7) no cérebro também são elevados em pessoas com doença de Alzheimer.[5]
A infecção primária pelo HHV-6B é a causa do exantema súbito comum da infância (também conhecida como roséola infantil ou sexta doença). Além disso, a reativação do HHV-6B é comum em receptores de transplante, o que pode causar várias manifestações clínicas, como encefalite, supressão da medula óssea e pneumonite.[6]
Uma variedade de testes é usada na detecção do HHV-6, alguns dos quais não diferenciam as duas espécies.[7]
História[editar | editar código-fonte]
Durante 1986, Syed Zaki Salahuddin, Dharam Ablashi e Robert Gallo cultivaram células mononucleares do sangue periférico de pacientes com AIDS e doenças linfoproliferativas. Células refratárias de vida curta e grandes que frequentemente continham corpos de inclusão intranucleares e/ou intracitoplasmáticos foram documentadas. A microscopia eletrônica revelou um novo vírus que eles denominaram Vírus B-Linfotrópico Humano (HBLV).[8][9]
Logo após sua descoberta, Ablashi et al. descreveram cinco linhas celulares que podem ser infectadas pelo recém-descoberto HBLV. Eles publicaram que o HSB-2, uma linha específica de linfócitos T, é altamente suscetível à infecção. A pesquisa pioneira de Ablashi concluiu sugerindo que o nome do vírus fosse alterado de HBLV para HHV-6, de acordo com a classificação provisória publicada de vírus do herpes.[10][11]
Anos depois, o HHV-6 foi dividido em subtipos. Pesquisas anteriores (1992) descreveram duas variantes muito semelhantes, porém únicas: HHV-6A e HHV-6B. A distinção foi justificada devido a clivagens únicas de endonucleases de restrição, reações de anticorpos monoclonais[12] e padrões de crescimento.[13]
O HHV-6A inclui várias cepas derivadas de adultos e seu espectro da doença não está bem definido, embora alguns pensem ser mais neurovirulento.[14][15] O HHV-6B é comumente detectado em crianças com roséola infantil, pois é o agente etiológico para essa condição. Dentro desses dois vírus, há uma homologia de sequência de 95%.[16]
Em 2012, o HHV-6A e o HHV-6B foram oficialmente reconhecidos como espécies distintas.[1]
Taxonomia[editar | editar código-fonte]
O HHV-6A e o HHV-6B foram reconhecidos pelo Comitê Internacional de Taxonomia de Vírus (ICTV) como espécies distintas em 2012. Roseolovírus humanos incluem HHV-6A, HHV-6B e HHV-7.[1]
O herpesvírus foi estabelecido como um gênero em 1971 no primeiro relatório do ICTV. Este gênero consistiu de 23 vírus em 4 grupos.[17] Em 1976, foi lançado um segundo relatório de ICTV, no qual esse gênero foi elevado ao nível da família - os herpetovirídeos. Devido à possível confusão com vírus derivados de répteis, o nome da família foi alterado no terceiro relatório (1979) para herpesviridae . Neste relatório, a família Herpesviridae foi dividida em 3 subfamílias (alfaherpesvirinae, betaherpesvirinae e gammaherpesvirinae) e 5 gêneros sem nome; 21 vírus foram reconhecidos como membros da família.[18]
Em 2009, foi criada a ordem Herpesvirales. Isso foi necessário pela descoberta de que os vírus do herpes de peixes e moluscos estão apenas distantes relacionados aos de pássaros e mamíferos. A ordem Herpesvirales contém três famílias, a Herpesviridae, que contém os herpesvírus de mamíferos, aves e répteis há muito reconhecidos, além de duas novas famílias - a família Alloherpesviridae, que incorpora o vírus do herpes de peixes e sapos ósseos, e a família Malacoherpesviridae, que contém vírus de moluscos.[19]
A partir de 2012, essa ordem atualmente possui 3 famílias, 4 subfamílias (1 não atribuída), 18 gêneros (4 não atribuídos) e 97 espécies.[1]
Estrutura[editar | editar código-fonte]
O diâmetro de um virião do HHV-6 é de cerca de 2000 Angstroms.[9] A porção externa do virião consiste em uma membrana de bicamada lipídica que contém glicoproteínas virais e é derivada da do hospedeiro. Abaixo deste envelope de membrana, há um tegumento que envolve um capsídeo icosaédrico, composto por 162 capsômeros. O capsídeo protetor do HHV-6 contém DNA linear de fita dupla.
Durante a maturação dos viriões HHV-6, as membranas celulares humanas são utilizadas para formar envelopes lipídicos virais (como é característico de todos os vírus envelopados). Durante esse processo, o HHV-6 utiliza balsas lipídicas, que são microdomínios membranosos enriquecidos por colesterol, esfingolípides e proteínas ancoradas em glicosilfosfatidilinositol.[20] Os primeiros pesquisadores suspeitaram que os virions do HHV-6 amadurecem no núcleo; alguns até publicaram incorretamente isso, pois generalizaram e aplicaram ao HHV-6 o que se sabia sobre outros vírus. No entanto, pesquisas publicadas em 2009 sugerem que o vírus HHV-6 utiliza vesículas derivadas da rede trans-Golgi para a montagem.[20]
Genoma[editar | editar código-fonte]
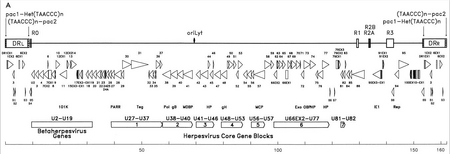
O material genético do HHV-6 é composto por DNA linear (circular durante uma infecção ativa), fita dupla que contém uma origem de replicação, dois terminais de replicação direta à esquerda e à direita de 8 a 10 kb e um segmento único de 143 a 145 kb.[22]
A origem da replicação (frequentemente rotulada como "oriLyt" na literatura) é onde a replicação do DNA começa.[21] Os terminais de replicação direta (RDE e RDD) possuem uma sequência TTAGGG repetida, idêntica à dos telômeros humanos. A variabilidade no número de replicações teloméricas é observada na faixa de 15 a 180.[23][24] Esses terminais também contêm sinais de clivagem e empacotamento pac-1 e pac-2 que são conservados entre os vírus do herpes.
O segmento único contém sete blocos genéticos principais (U27-U37, U38-U40, U41-U46, U48-U53, U56-U57, U66EX2-U77 e U81-U82),[21] que também é característica dos vírus do herpes. Esses genes conservados codificam proteínas envolvidas na replicação, clivagem e empacotamento do genoma viral em um virion maduro.[25] Além disso, eles codificam para um número de proteínas imunomoduladoras. O segmento único também possui um bloco de genes (U2-U19) que são conservados entre os vírus HHV-6, HHV-7 e citomegalovírus (vírus beta-herpes). Vários genes de segmentos únicos estão associados, por exemplo, à família HCMV US22. A tabela abaixo descreve algumas de suas propriedades conhecidas.[21]
Tratamento[editar | editar código-fonte]
Não existem produtos farmacêuticos aprovados especificamente para o tratamento da infecção pelo HHV-6, embora o uso de tratamentos com citomegalovírus (valganciclovir, ganciclovir,[26] cidofovir e foscarnet) tenha mostrado algum sucesso.[7] Esses fármacos são administrados com a intenção de inibir a polimerização apropriada do DNA, competindo com os nucleotídeos desoxitrifosfato[26] ou inativando especificamente as polimerases virais do DNA.[2]
Referências
- ↑ a b c d «Ratification vote on taxonomic proposals to the International Committee on Taxonomy of Viruses (2012)». Archives of Virology. 157: 1411–1422. 2012. PMID 22481600. doi:10.1007/s00705-012-1299-6
- ↑ a b «Divergent susceptibilities of human herpesvirus 6 variants to type I interferons». Proceedings of the National Academy of Sciences. 107: 8369–74. 2010. Bibcode:2010PNAS..107.8369J. PMC 2889514
 . PMID 20404187. doi:10.1073/pnas.0909951107
. PMID 20404187. doi:10.1073/pnas.0909951107
- ↑ «Quantitative analysis of human herpesvirus 6 cell tropism». Journal of Medical Virology. 75: 76–85. 2005. PMID 15543581. doi:10.1002/jmv.20240
- ↑ «Clinical parameters and HHV-6 active replication in relapsing—remitting multiple sclerosis patients». Journal of Clinical Virology. 37: S24–6. 2006. PMID 17276363. doi:10.1016/S1386-6532(06)70007-5
- ↑ «Multiscale Analysis of Independent Alzheimer's Cohorts Finds Disruption of Molecular, Genetic, and Clinical Networks by Human Herpesvirus». Neuron. 99: 64–82.e7. PMID 29937276. doi:10.1016/j.neuron.2018.05.023
- ↑ «Human herpesvirus 6 infection in hematopoietic stem cell transplant patients». British Journal of Haematology. 124: 421–32. 2004. PMID 14984492. doi:10.1046/j.1365-2141.2003.04788.x
- ↑ a b «Review, part 1: Human herpesvirus-6-basic biology, diagnostic testing, and antiviral efficacy». Journal of Medical Virology. 82: 1560–8. 2010. PMID 20648610. doi:10.1002/jmv.21839
- ↑ «Human Retrovirus Family: Cancer, Central Nervous System Disease, and AIDS». JNCI Journal of the National Cancer Institute. 80: 987–9. 1988. PMID 2842514. doi:10.1093/jnci/80.13.987
- ↑ a b «Isolation of a new virus, HBLV, in patients with lymphoproliferative disorders». Science. 234: 596–601. 1986. Bibcode:1986Sci...234..596Z. PMID 2876520. doi:10.1126/science.2876520
- ↑ «HBLV (or HHV-6) in human cell lines». Nature. 329. 207 páginas. Bibcode:1987Natur.329..207A. PMID 3627265. doi:10.1038/329207a0
- ↑ Ablashi, Dharam; Krueger, Gerhard (2006). Human Herpesvirus-6 General Virology, Epidemiology and Clinical Pathology. Elsevier 2nd ed. Burlington: [s.n.] ISBN 9780080461281
- ↑ «Analysis of a Neutralizing Antibody for Human Herpesvirus 6B Reveals a Role for Glycoprotein Q1 in Viral Entry». Journal of Virology. 85: 12962–71. 2011. PMC 3233151
 . PMID 21957287. doi:10.1128/JVI.05622-11
. PMID 21957287. doi:10.1128/JVI.05622-11
- ↑ «Human herpesvirus 6». Clinical Microbiology Reviews. 10: 521–67. 1997. PMC 172933
 . PMID 9227865. doi:10.1128/CMR.10.3.521
. PMID 9227865. doi:10.1128/CMR.10.3.521
- ↑ «The roles of viruses in brain tumor initiation and oncomodulation». Journal of Neuro-Oncology. 105: 451–66. 2011. PMC 3278219
 . PMID 21720806. doi:10.1007/s11060-011-0658-6
. PMID 21720806. doi:10.1007/s11060-011-0658-6
- ↑ «The latent human herpesvirus-6A genome specifically integrates in telomeres of human chromosomes in vivo and in vitro». Proceedings of the National Academy of Sciences. 107: 5563–5568. 2010. Bibcode:2010PNAS..107.5563A. PMC 2851814
 . PMID 20212114. doi:10.1073/pnas.0913586107
. PMID 20212114. doi:10.1073/pnas.0913586107
- ↑ «Human herpesvirus-6 entry into the central nervous system through the olfactory pathway». Proceedings of the National Academy of Sciences. 108: 13734–9. 2011. Bibcode:2011PNAS..10813734H. PMC 3158203
 . PMID 21825120. doi:10.1073/pnas.1105143108
. PMID 21825120. doi:10.1073/pnas.1105143108
- ↑ «The nomenclature and classification of viruses the International Committee on Nomenclature of Viruses». Virology. 46: 979–980. 1971. PMID 18619371. doi:10.1016/0042-6822(71)90102-4
- ↑ «Classification and nomenclature of viruses. Second report of the International Committee on Taxonomy of Viruses». Intervirology. 7: 1–115. 1976. PMID 826499. doi:10.1159/000149938
- ↑ Davison AJ (2010) Herpesvirus systematics. Vet. Microbiol. 143(1–2): 52–69
- ↑ a b «Y Human herpesvirus 6 envelope components enriched in lipid rafts: Evidence for virion-associated lipid rafts». Virology Journal. 6. 127 páginas. 2009. PMC 2743664
 . PMID 19689819. doi:10.1186/1743-422X-6-127
. PMID 19689819. doi:10.1186/1743-422X-6-127
- ↑ a b c d «Human herpesvirus 6B genome sequence: Coding content and comparison with human herpesvirus 6A». Journal of Virology. 73: 8040–8052. 1999. PMC 112820
 . PMID 10482553
. PMID 10482553
- ↑ «Human herpesvirus 6 encoded glycoprotein Q1 gene is essential for virus growth». Virology. 407: 360–7. 2010. PMID 20863544. doi:10.1016/j.virol.2010.08.018
- ↑ «The molecular biology of human herpesvirus-6 latency and telomere integration». Microbes and Infection. 13: 731–41. 2011. PMC 3130849
 . PMID 21458587. doi:10.1016/j.micinf.2011.03.006
. PMID 21458587. doi:10.1016/j.micinf.2011.03.006
- ↑ «Cloning human herpes virus 6A genome into bacterial artificial chromosomes and study of DNA replication intermediates». Proceedings of the National Academy of Sciences. 106: 19138–19143. 2009. Bibcode:2009PNAS..10619138B. PMC 2767366
 . PMID 19858479. doi:10.1073/pnas.0908504106
. PMID 19858479. doi:10.1073/pnas.0908504106
- ↑ «The molecular biology of human herpesvirus-6 latency and telomere integration». Microbes and Infection. 13: 731–41. 2011. PMC 3130849
 . PMID 21458587. doi:10.1016/j.micinf.2011.03.006
. PMID 21458587. doi:10.1016/j.micinf.2011.03.006
- ↑ a b «Detection and identification of U69 gene mutations encoded by ganciclovir-resistant human herpesvirus 6 using denaturing high-performance liquid chromatography». Journal of Virological Methods. 161: 223–30. 2009. PMID 19559728. doi:10.1016/j.jviromet.2009.06.016
Ligações externas[editar | editar código-fonte]
 Media relacionados com Herpesvírus humano 6 no Wikimedia Commons
Media relacionados com Herpesvírus humano 6 no Wikimedia Commons

