Retron
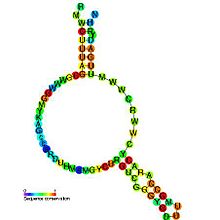
Um retron é uma sequência específica de ADN que se encontra no genoma de muitas espécies de bactérias, que codifica uma transcriptase reversa e um singular híbrido ARN/ADN monocatenário chamado ADN monocatenário multicópia (msDNA, do inglês multicopy single-stranded DNA). O chamado Retron msr RNA é o ARN não codificante produzido pelos elementos retron e é o imediato precursor da síntese do msDNA. O retron msr RNA prega-se numa estrutura secundária característica que contém um resíduo de guanosina conservado ao final duma estrutura de haste e ansa. A síntese do ADN pela transcriptase reversa (RT) codificada pelo retron tem como resultado uma quimera ADN/ARN que é composta por um ADN monocatenário ligado a um ARN monocatenário. A fibra de ARN está unida à extremidade 5' da cadeia de ADN por meio duma ligação fosfodiéster 2'-5' que ocorre na posição 2' do resíduo de guanosina conservada.

Os elementos retron possuem cerca de 2 kb de comprimento. Contêm um só operão que controla a síntese dum transcrito de ARN que leva três loci: msr, msd e ret, que estão envolvidos na síntese do msDNA. A porção de ADN do msDNA está codificada pelo gene msd, a porção de ARN está codificada pelo gene msr, enquanto que o produto do gene ret é uma transcritase reversa similar às transcritases reversas dos retrovírus e outros tipos de retroelementos.[1] Tal como outras transcritases reversas, o retron RT contém sete regiões de aminoácidos conservados (com números do 1 ao 7 na figura), incluindo uma sequência tyr-ala-asp-asp (YADD) muito conservada associada ao núcleo catalítico. O produto do gene ret é responsável por processar a porção msd/msr do transcrito de ARN em msDNA.
Durante muitos anos após a sua descoberta em viroses animais, pensava-se que a transcriptase reversa estava ausente nos procarióticas. Actualmente, não obstante, os elementos que codificam RT, ou seja, retroelementos, foram encontrados numa ampla variedade de bactérias. Os retrons foram a primeira família de retroelementos descobertos em bactérias; as outras duas famílias de retroelementos bacterianos conhecidas são os intrões do grupo II e os retroelementos geradores de diversidade (DGR, do inglês diversity-generating retroelements).[2] Os intrões do grupo II são os retroelementos bacterianos melhor caracterizados e o único tipo conhecido que apresenta mobilidade autónoma; consistem duma RT codificada numa estrutura de ARN com splicing catalítico. A mobilidade do intrão do grupo II é mediada por uma ribonucleoproteína que compreende um laço (lariat) intrão unido a duas proteínas codificadas no intrão. A segunda família de retroelementos bacterianos, os DGR, non son móbiles, pero funcionan diversificando as sequências de ADN.[3] Por exemplo, os DGR medeiam a mudança entre as fases patogénicas e de vida livre da bactéria Bordetella.[4]
Como os retron não são móveis, o seu aparecimento em diversas espécies bacterianas não é um fenômeno de "ADN egoísta". Em vez disso, os retronos devem dar alguma vantagem seletiva ao organismo hospedeiro, mas não se sabe qual pode ser essa vantagem. E com a exceção da produção de msDNA, nenhum fenótipo foi claramente associado a eles. Apesar do considerável número de investigações realizadas, pouco se sabe sobre a função dos msDNA, a mobilidade dos elementos retron ou os seus efeitos na célula hospedeira.[5][6]
Referências
- ↑ Lampson BC, Inouye M, Inouye S (2005). «Retrons, msDNA, and the bacterial genome» (PDF). Cytogenet Genome Res. 110 (1–4): 491–9. PMID 16093702. doi:10.1159/000084982
- ↑ Simon DM, Zimmerly S (2008). «A diversity of uncharacterized reverse transcriptases in bacteria». Nucleic Acids Res. 36 (22): 7219–29. doi:10.1093/nar/gkn867
- ↑ Medhekar B, Mille JF (2007). «Diversity-Generating Retroelements». Current Opinion in Microbiology. 10 (4): 388–395. PMC 2703298
 . PMID 17703991. doi:10.1016/j.mib.2007.06.004
. PMID 17703991. doi:10.1016/j.mib.2007.06.004
- ↑ Liu M, Gingery M, Doulatov SR, Liu Y, Hodes A, Baker S, Davis P, Simmonds M, Churcher C, Mungall K, Quail MA, Preston A, Harvill ET, Maskell DJ, Eiserling FA, Parkhill J, Miller JF (2004). «Genomic and Genetic Analysis of Bordetella Bacteriophages Encoding Reverse Transcriptase-Mediated Tropism-Switching Cassettes». J. Bacteriol. 186 (5): 1503–17. PMC 344406
 . PMID 14973019. doi:10.1128/JB.186.5.1503-1517.2004
. PMID 14973019. doi:10.1128/JB.186.5.1503-1517.2004
- ↑ Ahmed, AM; Shimamoto T (2003). «msDNA-St85, a multicopy single-stranded DNA isolated from Salmonella enterica serovar Typhimurium LT2 with the genomic analysis of its retron». FEMS Microbiol Lett. 224 (2): 291–297. PMID 12892895. doi:10.1016/S0378-1097(03)00450-6
- ↑ Lampson, BC; Xu C; Rice SA; Inouye S (2002). «A partial copy of msDNA from a new retron element is likely a retrotransposed DNA found in the myxobacterium Nannocystis exedens». Gene. 299 (1–2): 251–261. PMID 12459273. doi:10.1016/S0378-1119(02)00977-0
