Receptor de membrana
Principal[editar | editar código-fonte]
Receptores de superfície celular ( receptores de membrana , receptores transmembrana ) são receptores que estão embutidos na membrana plasmática das células [1] . Atuam na sinalização celular recebendo (ligando-se a) moléculas extracelulares . São proteínas integrais de membrana especializadas que permitem a comunicação entre a célula e o espaço extracelular . As moléculas extracelulares podem ser hormônios , neurotransmissores , citocinas , fatores de crescimento , moléculas de adesão celular ou nutrientes ; eles reagem com o receptor para induzir alterações no metabolismo e na atividade de uma célula. No processo de transdução de sinal , a ligação do ligante afeta uma mudança química em cascata através da membrana celular.
Estrutura e mecanismo[editar | editar código-fonte]
Muitos receptores de membrana são proteínas transmembrana . Existem vários tipos, incluindo glicoproteínas e lipoproteínas . Centenas de receptores diferentes são conhecidos e muitos mais ainda precisam ser estudados. Os receptores transmembrana são normalmente classificados com base em sua estrutura terciária (tridimensional). Se a estrutura tridimensional é desconhecida, eles podem ser classificados com base na topologia da membrana . Nos receptores mais simples, as cadeias polipeptídicas atravessam a bicamada lipídica uma vez, enquanto outros, como os receptores acoplados à proteína G, cruze até sete vezes. Cada membrana celular pode ter vários tipos de receptores de membrana, com diferentes distribuições de superfície. Um único receptor também pode ser distribuído de maneira diferente em diferentes posições da membrana, dependendo do tipo de membrana e da função celular. Os receptores geralmente estão agrupados na superfície da membrana, em vez de distribuídos uniformemente.
Mecanismo[editar | editar código-fonte]
Dois modelos foram propostos para explicar o mecanismo de ação dos receptores transmembrana.
- Dimerização : O modelo de dimerização sugere que antes da ligação do ligante, os receptores existem em uma forma monomérica . Quando ocorre a ligação do agonista, os monômeros se combinam para formar um dímero ativo .
- Rotação : A ligação do ligante à parte extracelular do receptor induz uma rotação (mudança conformacional) de parte das hélices transmembranares do receptor. A rotação altera quais partes do receptor são expostas no lado intracelular da membrana, alterando como o receptor pode interagir com outras proteínas dentro da célula.
Domínios[editar | editar código-fonte]
Os receptores transmembrana na membrana plasmática geralmente podem ser divididos em três partes.

Domínios extracelulares[editar | editar código-fonte]
O domínio extracelular está apenas externamente à célula ou organela . Se a cadeia polipeptídica atravessa a bicamada várias vezes, o domínio externo compreende alças entrelaçadas através da membrana. Por definição, a principal função de um receptor é reconhecer e responder a um tipo de ligante.
Por exemplo, um neurotransmissor , hormônio ou íons atômicos podem se ligar ao domínio extracelular como um ligante acoplado ao receptor. Klotho é uma enzima que efetua um receptor para reconhecer o ligando ( FGF23 ).
Domínios intracelulares[editar | editar código-fonte]
O domínio intracelular (ou citoplasmático ) do receptor interage com o interior da célula ou organela, retransmitindo o sinal. Existem dois caminhos fundamentais para essa interação:
- O domínio intracelular se comunica por meio de interações proteína-proteína contra proteínas efetoras , que por sua vez passam um sinal para o destino.
- Com receptores ligados a enzimas , o domínio intracelular tem atividade enzimática . Muitas vezes, esta é a atividade da tirosina quinase . A atividade enzimática também pode ser devida a uma enzima associada ao domínio intracelular.
Transdução de sinal[editar | editar código-fonte]

Os processos de transdução de sinal através de receptores de membrana envolvem as reações externas, nas quais o ligante se liga a um receptor de membrana, e as reações internas, nas quais a resposta intracelular é desencadeada.
A transdução de sinal através de receptores de membrana requer quatro partes:
- Molécula de sinalização extracelular: uma molécula de sinalização extracelular é produzida por uma célula e é pelo menos capaz de viajar para as células vizinhas.
- Proteína receptora: as células devem ter proteínas receptoras de superfície celular que se ligam à molécula de sinalização e se comunicam para dentro da célula.
- Proteínas sinalizadoras intracelulares: passam o sinal para as organelas da célula. A ligação da molécula de sinal à proteína receptora ativará as proteínas de sinalização intracelular que iniciam uma cascata de sinalização.
- Proteínas-alvo: as conformações ou outras propriedades das proteínas-alvo são alteradas quando uma via de sinalização está ativa e altera o comportamento da célula.

Os receptores de membrana são divididos principalmente por estrutura e função em 3 classes: O receptor ligado ao canal iônico ; O receptor ligado à enzima ; e o receptor acoplado à proteína G.
- Os receptores ligados a canais iônicos possuem canais iônicos para ânions e cátions e constituem uma grande família de proteínas transmembranares multipassagem. Eles participam de eventos de sinalização rápida geralmente encontrados em células eletricamente ativas, como neurônios . Eles também são chamados de canais iônicos controlados por ligantes . A abertura e o fechamento dos canais iônicos são controlados por neurotransmissores .
- Os receptores ligados a enzimas são as próprias enzimas ou ativam diretamente as enzimas associadas. Estes são tipicamente receptores transmembranares de passagem única, com o componente enzimático do receptor mantido intracelular. A maioria dos receptores ligados a enzimas são ou estão associados a proteínas quinases.
- Os receptores acoplados à proteína G são proteínas integrais de membrana que possuem sete hélices transmembranares. Esses receptores ativam uma proteína G após a ligação do agonista , e a proteína G medeia os efeitos do receptor nas vias de sinalização intracelular.
Receptor ligado ao canal iônico[editar | editar código-fonte]

Desde 2009, existem 6 tipos conhecidos de receptores ligados a enzimas : Receptor tirosina quinase ; receptores associados à tirosina quinase; Fosfatases de tirosina tipo receptor ; Receptores serina / treonina quinases ; Receptores guanilil ciclases e receptores associados a histidina quinase . As tirosina quinases receptoras têm a maior população e a aplicação mais ampla. A maioria dessas moléculas são receptores para fatores de crescimento , como fator de crescimento epidérmico (EGF), fator de crescimento derivado de plaquetas (PDGF), fator de crescimento de fibroblastos (FGF),fator de crescimento de hepatócitos (HGF), fator de crescimento nervoso (NGF) e hormônios como a insulina . A maioria desses receptores dimerizará após a ligação com seus ligantes, a fim de ativar outras transduções de sinal. Por exemplo, após o receptor do fator de crescimento epidérmico (EGF) se ligar ao seu ligante EGF, os dois receptores dimerizam e então sofrem fosforilação dos resíduos de tirosina na porção enzimática de cada molécula receptora. Isso ativará a tirosina quinase e catalisará outras reações intracelulares.
Receptores acoplados á proteína G[editar | editar código-fonte]
Os receptores acoplados à proteína G compreendem uma grande família de proteínas de receptores transmembrana. Eles são encontrados apenas em eucariotos . Os ligantes que ligam e ativam esses receptores incluem: compostos fotossensíveis, odores , feromônios , hormônios e neurotransmissores . Estes variam em tamanho de pequenas moléculas a peptídeos e grandes proteínas . Os receptores acoplados à proteína G estão envolvidos em muitas doenças e, portanto, são os alvos de muitas drogas medicinais modernas.
Existem duas vias principais de transdução de sinal envolvendo os receptores acoplados à proteína G: a via de sinalização do AMPc e a via de sinalização do fosfatidilinositol . Ambos são mediados pela ativação da proteína G. A proteína G é uma proteína trimérica, com três subunidades designadas como α, β e γ. Em resposta à ativação do receptor, a subunidade α libera o difosfato de guanosina (GDP) ligado, que é deslocado pelo trifosfato de guanosina (GTP), ativando assim a subunidade α, que então se dissocia das subunidades β e γ. A subunidade α ativada pode afetar ainda mais as proteínas de sinalização intracelular ou direcionar diretamente as proteínas funcionais.
Doença relacionada ao receptor de membrana[editar | editar código-fonte]
Se os receptores de membrana estiverem desnaturados ou deficientes, a transdução do sinal pode ser prejudicada e causar doenças. Algumas doenças são causadas por distúrbios da função do receptor de membrana. Isso ocorre devido à deficiência ou degradação do receptor por meio de alterações nos genes que codificam e regulam a proteína receptora. O receptor de membrana TM4SF5 influencia a migração de células hepáticas e hepatoma . Além disso, o receptor NMDA cortical influencia a fluidez da membrana e está alterado na doença de Alzheimer. Quando a célula é infectada por um vírus não envelopado, o vírus primeiro se liga a receptores de membrana específicos e então passa a si mesmo ou a um componente subviral para o lado citoplasmático da membrana celular. No caso de poliovírus , sabe-se in vitro que as interações com os receptores causam rearranjos conformacionais que liberam uma proteína virion chamada VP4. O terminal N do VP4 é miristilado e, portanto, hidrofóbico【ácido mirístico =CH 3 (CH 2 ) 12 COOH】. Propõe-se que as alterações conformacionais induzidas pela ligação ao receptor resultam na ligação do ácido mirístico ao VP4 e na formação de um canal para o RNA.
Projeto de drogas baseado em estrutura[editar | editar código-fonte]
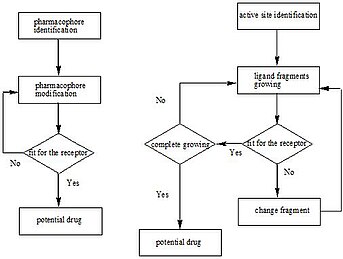
Por meio de métodos como a cristalografia de raios X e a espectroscopia de RMN , as informações sobre as estruturas 3D das moléculas alvo aumentaram drasticamente, assim como as informações estruturais sobre os ligantes. Isso impulsiona o rápido desenvolvimento do design de drogas baseado em estrutura.
Algumas dessas novas drogas têm como alvo os receptores de membrana. As abordagens atuais para o design de drogas baseadas em estrutura podem ser divididas em duas categorias.
A primeira categoria é sobre a determinação de ligantes para um determinado receptor. Isso geralmente é feito por meio de consultas ao banco de dados, simulações biofísicas e construção de bibliotecas químicas. Em cada caso, um grande número de potenciais moléculas de ligantes é rastreado para encontrar aquelas que se encaixam no bolso de ligação do receptor. Esta abordagem é geralmente referida como design de drogas baseado em ligantes. A principal vantagem de pesquisar um banco de dados é que ele economiza tempo e energia para obter novos compostos eficazes. Outra abordagem de design de drogas baseado em estrutura é sobre o mapeamento combinatório de ligantes, que é referido como design de drogas baseado em receptor. Nesse caso, as moléculas de ligantes são projetadas dentro das restrições de um bolso de ligação, montando pequenos pedaços de maneira gradual. Essas peças podem ser átomos ou moléculas. A principal vantagem desse método é que novas estruturas podem ser descobertas.
Outros exemplos[editar | editar código-fonte]
- Receptor adrenérgico
- Receptores olfativos
- Tirosina quinase receptora
- Receptor do fator de crescimento epidérmico
- Receptor de Insulina
- Receptores do fator de crescimento de fibroblastos ,
- Receptores de neurotrofina de alta afinidade
- Receptores de efrina
- Integrinas
- Receptor do Fator de Crescimento Nervoso de Baixa Afinidade
- receptor NMDA
- Vários receptores imunológicos
Referências[editar | editar código-fonte]
- Monnot GC, Wegrecki M, Cheng TY, Chen YL, Sallee BN, Chakravarthy R, Karantza IM, Tin SY, Khaleel AE, Monga I, Uwakwe LN, Tillman A, Cheng B, Youssef S, Ng SW, Shahine A, Garcia -Vilas JA, Uhlemann AC, Bordone LA, Han A, Rohde CH, Ogg G, Moody DB, Rossjohn J, de Jong A (janeiro de 2023). "Antígenos de fosfatidilglicerol estafilocócico ativam células T humanas via CD1a". Natureza Imunologia . 24 (1): 110–122 . doi : 10.1038/s41590-022-01375-z . PMID 35265979 .
- Cuatrecasas P (1974) "Membrane Receptors". Annual Review of biochemistry. 43 : 169-214 doi:10.1146/annurev.bi.43.070174.001125. PMID 4368906. S2CID 44727052.
- Dautzenberg FM, Hauger RL (February 2002). "The CRF peptide family and their receptors: yet more partners discovered". Trends Pharmacol. Sci. 23 (2): 71–7. doi:10.1016/S0165-6147(02)01946-6. PMID 11830263.
- Rivière S, Challet L, Fluegge D, Spehr M, Rodriguez I (May 2009). "Formyl peptide receptor-like proteins are a novel family of vomeronasal chemosensors". Nature. 459 (7246): 574–7. doi:10.1038/nature08029. PMID 19387439.
- Rothberg K.G.; Ying Y.S.; Kamen B.A.; Anderson R.G. (1990). "Cholesterol controls the clustering of the glycophospholipid-anchored membrane receptor for 5-methyltetrahydrofolate". The Journal of Cell Biology. 111 (6): 2931–2938. doi:10.1083/jcb.111.6.2931. PMC 2116385. PMID 2148564.
- Jacobson C.; Côté P.D.; Rossi S.G.; Rotundo R.L.; Carbonetto S. (2001). "The Dystroglycan Complex Is Necessary for Stabilization of Acetylcholine Receptor Clusters at Neuromuscular Junctions and Formation of the Synaptic Basement Membrane". The Journal of Cell Biology. 152 (3): 435–450. doi:10.1083/jcb.152.3.435. PMC 2195998. PMID 11157973.
- Maruyama, Ichiro N. (2015-09-01). "Activation of transmembrane cell-surface receptors via a common mechanism? The "rotation model"". BioEssays. 37 (9): 959–967. doi:10.1002/bies.201500041. ISSN 1521-1878. PMC 5054922. PMID 26241732
- Superfamilies of single-pass transmembrane receptors in Membranome database
- Superfamilies of single-pass transmembrane protein ligands and regulators of receptors in Membranome database
- Ullricha A., Schlessingerb J.; Schlessinger, J (1990). "Signal transduction by receptors with tyrosine kinase activity". Cell. 61 (2): 203–212. doi:10.1016/0092-8674(90)90801-K. PMID 2158859.
- Kenneth B. Storey (1990). Functional Metabolism. Wiley-IEEE. pp. 87–94. ISBN 978-0-471-41090-4.
- Hille B. (2001). Ion channels of excitable membranes. Sunderland, Mass. ISBN 978-0-87893-321-1
- Miyazawa A.; Fujiyoshi Y.; Unwin N. (2003). "Structure and gating mechanism of the acetylcholine receptor pore". Nature. 423 (6943): 949–955. doi:10.1038/nature01748. PMID 12827192.
- Akabas M.H.; Stauffer D.A.; Xu M.; Karlin A. (1992). "Acetylcholine receptor channel structure probed in cysteine-substitution mutants". Science. 258 (5080): 307–310. doi:10.1126/science.1384130. PMID 1384130.
- King N, Hittinger CT, Carroll SB (2003). "Evolution of key cell signaling and adhesion protein families predates animal origins". Science. 301 (5631): 361–3. doi:10.1126/science.1083853. PMID 12869759.
- Filmore, David (2004). "It's a GPCR world". Modern Drug Discovery. 2004 (November): 24–28.
- Gilman A.G. (1987). "G Proteins: Transducers of Receptor-Generated Signals". Annual Review of Biochemistry. 56: 615–649. doi:10.1146/annurev.bi.56.070187.003151. PMID 3113327. S2CID 33992382.
- Müller-Pillascha F.; Wallrappa C.; Lachera U.; Friessb H.; Büchlerb M.; Adlera G.; Gress T. M. (1998). "Identification of a new tumour-associated antigen TM4SF5 and its expression in human cancer". Gene. 208 (1): 25–30. doi:10.1016/S0378-1119(97)00633-1. PMID 9479038.
- Scheuer K.; Marasb A.; Gattazb W.F.; Cairnsc N.; Förstlb H.; Müller W.E. (1996). "Cortical NMDA Receptor Properties and Membrane Fluidity Are Altered in Alzheimer's Disease". Dementia. 7 (4): 210–214. doi:10.1159/000106881. PMID 8835885.
- Wang R.; Gao Y.; Lai L. (2000). "LigBuilder: A Multi-Purpose Program for Structure-Based Drug Design". Journal of Molecular Modeling. 6 (7–8): 498–516. doi:10.1007/s0089400060498.
- Schneider G.; Fechner U. (2005). "Computer-based de novo design of drug-like molecules". Nature Reviews Drug Discovery. 4 (8): 649–663. doi:10.1038/nrd1799. PMID 16056391.
- Jorgensen W.L. (2004). "The Many Roles of Computation in Drug Discovery". Science. 303 (5665): 1813–1818. doi:10.1126/science.1096361. PMID 15031495. S2CID 1307935
