Haplogrupo U (mtADN)
O haplogrupo U é um haplogrupo mitocondrial do ADN humano (mtADN).
Decorrente do haplogrupo R com o início do Paleolítico Superior, os seus diferentes subconjuntos (etiquetados U1–U9, divergendo no curso do Paleolítico Superior) encontram-se amplamente distribuídos pela Europa do norte, central e oriental, Ásia ocidental e do sul, assim como na África do Norte e o Chifre da África.
O subconjunto mais comum do haplogrupo U8b, que surgiu entre 30.000 e 22.000 anos atrás, tem sido referido diferenciadamente como haplogrupo K.
Origem[editar | editar código-fonte]
O haplogrupo U desce do ramal da árvore filogenética do haplogrupo R mtADN. A mutaçoes definitórias são (A11467G, A12308G, G12372A). Estima-se que surgiu aproximadamente entre 43.000 e 50.000 anos atrás, no início do Paleolítico Superior (95% CI: 46,530 ± 3,290 de acordo com Behar et al., 2012)
DNA antigo, classificado como pertencente ao haplogrupo mitocondrial U* foi recuperado a partir de restos humanos na Sibéria Ocidental, datados de cerca de 45.000 anos atrás.[1]
O haplogrupo U também foi encontrado entre as múmias do Egito antigo, encontradas na escavação arqueológica em Abusir el-Meleq no Egito Central, datadas do 1º milênio antes de Cristo.[2]
O haplogrupo U é encontrado em 15% das castas indianas, e no 8% das populações tribais da índia.[3] Ele também É encontrado em cerca do 11% dos europeus nativos e é considerado como o haplogrupo materno mais antigo encontrado na região.[4][5] Num estudo a partir de 2013, todas, exceto uma, das sequências humanas antigas da Europa analisadas pertenciam ao haplogrupo materno U, confirmando assim as descobertas anteriores, que mostravam o haplogrupo U como o tipo dominante de ADN mitocondrial (mtADN) na Europa antes da propagação da agricultura na Europa e a presença e a extensão dos indo-europeus pela Europa Ocidental.[6][7]
O haplogrupo U tem vários subconjuntos contados a partir de U1 até U9. O haplogrupo K é uma sub-linhagem de U8.[8] A sua antiguidade permitiu uma distribuição ampla dos subgrupos descendentes através da Eurásia ocidental, África do norte e Ásia do sul. Algumas sub-linhagens do haplogrupo U teem uma geográfica ainda mais específica.
Sub-linhagens[editar | editar código-fonte]
Os subconjuntos são etiquetados U1–U9; l'haplogrupo K é uma sub-linhagem de U8.
Van Oven e Kayser (2009) proposaram as sub-linhagens "U2'3'4'7'8' e 'U4'9".[9] Behar et al (2012) o modificaram agrupando "U4'9" como subordinada a "U2'3'4'7'8" para uma nova sub-linhagem intermediária "U2'3'4'7'8'9".
O haplogrupo U1 é estimado que surgiu entre 26,000 e 37.000 anos atrás. Está localizado com frequência muito baixa em toda a Europa. É mais frequente observá-lo na Europa oriental, a Anatólia e o Oriente Próximo. Também é encontrado em baixas frequencias na Índia. A sub-linhagem U1 está localizada na região de Suanécia , na Geórgia, num 4.2%. O subconjunto U1a é encontrado a partir da Índia até a Europa, mas é muito raro entre as franjas norte e atlântica da Europa, incluindo o reino Unido e Escandinávia. Têm-se observado diversos exemplos na Toscana. Na Índia, U1a foi encontrado na região de Kerala. U1b tem uma propagação semelhante, mas é mais raro do que U1a. Alguns exemplos de U1b foram encontrados entre os judeus da diáspora. Sub-linhagens U1a e U1b aparecem com igual frequência na Europa de leste.[10]
A sub-linhagens de U1 são: U1a (com sub-sub-linhagens U1a1, U1a1a, U1a1a1, U1a1b)[11] e U1b.
A análise de DNA de restos escavados, onde se encontram as ruínas da igreja de Santo Agostinho, em Goa, na Índia, revelou a sub-linhagem única U1b, que está ausente na Índia, mas presente na Geórgia e regiões circunvizinhas.[12] Dado que a análise genética confirma as evidências arqueológicas e literárias, acredita-se que os restos pertencem a Ketevan a Mártir, rainha da Geórgia.
O haplogrupo U1 tem sido observado entre amostras no cemitério de Kulubnarti, Sudão, que datam do período de cristã primitivo (d.C. 550-800).[13]
O conjunto raro U1 também é encontrado entre os Argelinos em Orã (0,83%-1.08%) e na tribo dos reguibates dos Saarauís (0,93%).[14]
Haplogrupo U5[editar | editar código-fonte]
A idade do U5 é estimada entre 25.000 e 35.000 anos.[15] Cerca do 11% do total de europeus e 10% dos europeu-americanos estão dentro do haplogrupo U5.
U5 foi encontrado em restos humanos que datam do Mesolítico na Inglaterra, Alemanha, Lituânia, Polônia, Portugal, Rússia, Suécia, França e Espanha.Esqueletos do neolítico (cerca de 7.000 anos de antiguidade) que foram escavados na caverna Avellaner na Catalunha, incluem um indivíduo que também leva o haplogrupo U5.
O haplogrupo U5 e as suas sub-linhagens U5a e U5b formam hoje a maior concentração de população no extremo norte, entre os Sami, Finlandeses e Estonianos. No entanto, ele é distribuído amplamente em níveis mais baixos por toda a Europa. Esta distribuição, e a idade do haplogrupo, indicam que os indivíduos pertencentes a este grupo foram parte da expansão inicial após a retirada da coberta de gelo da Europa por volta de 10.000 anos atrás.
Além disso, o haplogrupo U5 é encontrado em frequências pequenas e com muita menos diversidade no Oriente médio e em partes da África do Norte (áreas com concentrações de U6 notáveis), sugerindo uma migração para trás do povo da Europa para o sul.[16]
O haplogrupo mitocondrial U5a também tem sido associado com a progressão acelerada em indivíduos afetados pelo HIV para AIDS e morte.[17]
- U5 tem polimorfismos nas localizações de 3197 9477 13617 16192 16270
- U5a surgiu ao redor de entre 17,000 e 27,000 anos atrás e tem polimorfismos em 14793 16256 (+ Ua5 polimorfismos).
- U5a1 surgiu entre 14,000 e 20,000 anos atrás e tem polimorfismos em 15218 16399 (+ U5a polimorfismos).
- U5a1a surgiu entre 8,000 e 6,000 anos atrás e tem polimorfismos dentro 1700 16192 (+ U5a1 polimorfismos).
- U5a1a1 surgiu entre 3,000 e 11,000 anos atrás e tem polimorfismos em 5495 15924 (+ Ua5a1a polimorfismos).
- U5a1a1a surgiu há menos de 6,000 anos e tem polimorfismos em 3816 (A3816G) (e perdeu o seu polimorfismo em 152 (mutação posterior) + Ua51a1 polimorfismos).[18]
- U5a1a1b surgiu ao redor de entre 600 e 6,000 anos atrás e tem polimorfismos em 15110 (G15110A) (e perdeu o seu polimorfismo em 152 (retromutação) + U5a1a1 polimorfismos).
- U5a1a1c tem polimorfismos em 6905 (A6905G) 13015 (T13015C) (+ U5a1a1 polimorfismos).
- U5a1a1d surgiu há menos de 4,300 anos atrás e tem os polimorfismos: G185A T204C T16362C (e perdeu o seu polimorfismo em 152 (retromutação) + U5a1a1 polimorfismos).
- U5a1a2 surgiu entre 7,000 e 14,000 anos atrás e tem polimorfismos em 573.1C (eliminação) 12346 (+ U5a1a polimorfismos).
- U5a1a2a surgiu há menos de 5,400 anos atrás e tem polimorfismos em 5319 6629 6719 (+ U5a1a2 polimorfismos).
- U5a1a2a1 surgiu há menos de 3,400 anos atrás e tem polimorfismos T6293C (+ U5a1a2a polimorfismos).
- U5a1a2a surgiu há menos de 5,400 anos atrás e tem polimorfismos em 5319 6629 6719 (+ U5a1a2 polimorfismos).
- U5a1a1 surgiu entre 3,000 e 11,000 anos atrás e tem polimorfismos em 5495 15924 (+ Ua5a1a polimorfismos).
- U5a1b surgiu entre 6,000 e 11,000 anos atrás e tem polimorfismos dentro 9667 (A9667G) (+ U5a1 polimorfismos).
- U5a1b1 surgiu entre 5,000 e 9,000 anos atrás e tem polimorfismos em 16291 (C16291T) (+U5a1b polimorfismos).
- U5a1b1a surgiu entre 2,500 e 7,500 anos atrás e tem polimorfismos T4553C (+ U5a1b1 polimorfismos).
- U5a1b1a1 surgiu há menos de 4,000 anos atrás e tem polimorfismos C14574T (+ U5a1b1a polimorfismos).
- U5a1b1b surgiu ha menos 8,000 de anos atrás e tem polimorfismos em 8119 (T8119C) (+ U5a1b1 polimorfismos).
- U5a1b1c surgiu entre 3,000 e 7,000 anos atrás e tem polimorfismos em 9055 (G9055A) (+ U5a1b1 polimorfismos).
- U5a1b1c1 surgiu há menos de 5,000 anos atrás e tem polimorfismos em 1187 (T1187C) (+ U5a1b1c polimorfismos).
- U5a1b1c2 surgiu há menos de 5,000 anos atrás e tem polimorfismos em 3705 (G3705A) (+ U5a1b1c polimorfismos).
- U5a1b1d tem polimorfismos em 12358 16093 (+ U5a1b1 polimorfismos).
- U5a1b1e tem polimorfismos em 12582 16192 16294 (+ U5a1b1 polimorfismos).
- U5a1b1a surgiu entre 2,500 e 7,500 anos atrás e tem polimorfismos T4553C (+ U5a1b1 polimorfismos).
- U5a1b2 tem polimorfismos em 9632 (+ U5a1b polimorfismos).
- U5a1b3 tem polimorfismos em 16362 16428 (+ U5a1b polimorfismos).
- U5a1b1 surgiu entre 5,000 e 9,000 anos atrás e tem polimorfismos em 16291 (C16291T) (+U5a1b polimorfismos).
- U5a1c surgiu ao redor de há 13000 anos e tem polimorfismos em 16320 (+ U5a1 polimorfismos).
- U5a1c1 tem polimorfismos em 195 13802 (+ U5a1c polimorfismos).
- U5a1c2 tem polimorfismos em 961 965.1C (eliminação) (+ U5a1c polimorfismos).
- U5a1d surgiu ao redor de há 19000 anos e tem polimorfismos em 3027 (+ U5a1 polimorfismos).
- U5a1d1 tem polimorfismos em 5263 13002 (a adenosina) (+ U5a1d polimorfismos).
- U5a1d2 tem polimorfismos em 573.1C (eliminação) 3552 (+ U5a1d polimorfismos).
- U5a1d2a tem polimorfismos em 195 4823 5583 16145 16189 (+ U5a1d2 polimorfismos).
- U5a1e tem polimorfismos em 3564 8610 (+ U5as1 polimorfismos).
- U5a1f tem polimorfismos em 6023 (+ U5as1 polimorfismos).
- U5a1a surgiu entre 8,000 e 6,000 anos atrás e tem polimorfismos dentro 1700 16192 (+ U5a1 polimorfismos).
- U5a2 surgiu ao redor de há 14000 anos e tem polimorfismos em 16526 ( + U5a polimorfismos).
- U5a2a surgiu ao redor de há 6000 anos e tem polimorfismos em 13827 13928C 16114 16294 ( + U5a2 polimorfismos).
- U5a2b surgiu ao redor de há 8000 anos e tem polimorfismos em 9548 ( + U5a2 polimorfismos).
- U5a2c surgiu ao redor de há 13000 anos e tem polimorfismos em 10619 ( + U5a2 polimorfismos).
- U5a2d tem polimorfismos em 7843 7978 8104 11107 16192! (retromutação em 16192 da sequência Cambridge original) (+ U5a2 polimorfismos).
- U5a2e tem polimorfismos em 151 152 3768 15289 16189 16311 16362 (+ U5a2 polimorfismos).
- U5a1 surgiu entre 14,000 e 20,000 anos atrás e tem polimorfismos em 15218 16399 (+ U5a polimorfismos).
- U5b surgiu entre 19,000 e 26,000 anos atrás e tem polimorfismos em 150 7768 14182 (+ U5 polimorfismos).[19] Encontrado nos Berberes Siwa do oasi Siwa.[20]
- U5b1 surgiu entre 11,000 e 20,000 anos atrás e tem polimorfismos em 5656 (+ U5b polimorfismos).[21]
- U5b1a tem polimorfismos em 5656 15097, 16189 e perdeu o seu polimorfismo em 7028 (retromutação) (+ U5b1 polimorfismos).
- U5b1b: foi encontrado nos Samis de Escandinàvia, finlandeses e o Berberes de África do norte, os quais se encontrou que compartilham um ramal extremamente jovem, com uma idade de meramente ∼9,000 anos. U5bib foi também encontrado nos Fulbe e Papel na Guiné Bissau e nos Yakuts da Siberia norte-oriental.[22][23] Surgiu ao redor de há 11000 anos e tem polimorfismos em 12618 16189 ( + Ua5b1 polimorfismos).
- U5b1c tem polimorfismos em 5656 15191, 16189, 16311 + U5b1 polimorfismos) e surgiu aproximadamente há 13,000 anos
- U5b1d tem polimorfismos em 5437 5656 e perdeu o seu polimorfismo em 16192 (retromutção) (+ U5b1 polimorfismos).
- U5b1e tem polimorfismes em 152 2757 10283 12616 16189 e perdeu o seu polimorfismo em 16192 (retromutação) (+ U5b1 polimorfismos) e surgiu aproximadamente há 6600 anos. U5b1e é encontrado principalmente na Europa central entre tchecos, eslovacos, húngaros e russos do sul.[24]
- U5b1g tem polimorfismos em 151 228 573.1C 5656 10654 13759 14577 (+ U5b1 polimorfismos).
- U5b2 surgiu entre 17,000 e 23,000 anos atrás e tem polimorfismos em 1721 13637(+ U5b polimorfismos).[25] O grupo foi encontrado em restos que datam de tempos pré-históricos na Europa, como o subconjunto U5b2c1 do homem da Braña (encontrado no jazimento arqueológico La Braña na Espanha).
- U5b2a entre 12,000 e 19,000 anos atrás, prevalecendo em Europa Central.[26][27]
- U5b2a1 entre 9,000 e 18,000 anos atrás, descendentes U5b2a1a, U5b2a1b.
- U5b2a2 entre 7,000 e 14,000 anos atrás, descendentes U5b2a2a, U5b2a2b, U5b2a2c. Frequente na Europa central, U5b2a2a1 especialmente em Polônia.
- U5b2a3 entre há 3,000 e 14,000 anos atrás, descendente U5b2a3a.
- U5b2a4 entre 1,000 e 10,000 anos atrás, descendente U5b2a4a.
- U5b2a5 há menos de 2,600 anos atrás, descendente U5b2a5a.
- U5b2a6 há menos de 12,000 anos atrás.
- U5b2b entre 12,000 e 17,000 anos atrás.[28]
- U5b2c entre 7,000 e 18,000 anos atrás.[29]
- U5b2a entre 12,000 e 19,000 anos atrás, prevalecendo em Europa Central.[26][27]
- U5b3 a sub-linhagem provavelmente originou-se na península italiana, e encontra-se principalmente entre os Sardos.[32]
- U5b1 surgiu entre 11,000 e 20,000 anos atrás e tem polimorfismos em 5656 (+ U5b polimorfismos).[21]
- U5a surgiu ao redor de entre 17,000 e 27,000 anos atrás e tem polimorfismos em 14793 16256 (+ Ua5 polimorfismos).
Haplogrupo U6[editar | editar código-fonte]
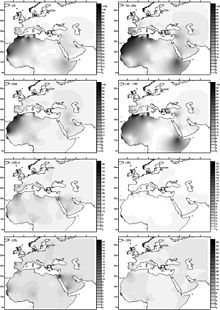
O haplogrupo U6 é datado entre 31,000 e 43,000 anos atrás por Behar et al. (2012). Isto é consistente com a descoberta de U6* basal nas mostras de ADN antigo dum indivíduo romeno (Peștera Muierilor) datado de há 35.000 anos.[33] Hervella et al. (2016) tomaram este achado como prova pela migração de volta do paleolítico do Homo Sapiens desde Eurásia para a África. A descoberta do U6* ADN basal antigo contribuiu a atrasar a configuração da idade estimada de U6 ao redor de há 46.000 anos.[34]
O Haplogrupo U6 é comum (com uma prevalência de cerca de 10%) na África do Norte-oeste (com um máximo de 29% nos algerianos Mozabites[35]) e nas Canárias (18%, em média, com uma frequência de pico de 50,1% na ilha de La Gomera). É encontrado também na península ibérica, onde tem a diversidade mais elevada (10 de 19 de sub-linhagens encontrada somente nesta região, e não na África), África do nordeste e ocasionalmente noutros locais.[36] U6 é também encontrado em frequências mais baixas, na bacia do lago Chade, incluindo o ramal canário raro. Isto sugere que os antigos portadores da linhagem podem haver habitado ou passado através da bacia do Chade no seu caminho para o oeste, em direção às Ilhas Canárias.[37]
O haplogrupo U6 acredita-se ter vido do norte da África, a partir do Oriente Próximo, em torno de 30.000 anos atrás. Foi encontrado entre as amostras Ibero-Mauritanas do epipaleolítico no local pré-histórico Taforalt.[38] Apesar da maior diversidade do U6 ibérico, Maca-Meyer defende uma origem no Oriente médio desta linhagem baseando-se na diversidade máxima do sub-linhagem U6a na região, onde teria chegado a partir do oeste da Ásia, com a incidência da ibérica representando principalmente os fluxos migratórios a partir do Magrebe e não a persistência duma população com raízes europeias.
De acordo com Hernández et al., 2015 "a entrada calculada para as linhagens U6 do norte da África, na península Ibérica ha 10 ky correlacionam-se bem com outras linhagens L africanas, indicando que U6 e algumas linhagens mexeram juntos da África para a Ibéria no Holoceno inicial".[39]
U6 tem quatro principais sub-linhagens:
O subgrupo U6a reflete a primeira expansão africana dos magrebinos voltando ao leste. A linhagem derivada U6a1 indica um movimento de volta da África oriental para o Magrebe e o Médio Oriente. Esta migração coincide com a expansão provável linguistica afro-asiática. As linhagens U6b e U6C, restritas à África ocidental, tiveram expansões mais localizadas. U6b provavelmente chegou na península ibérica durante a expansão capsiana na África do Norte. Dois derivados locais dessas linhagens (U6b1 e U6c1) indicam a chegada de colonos do norte da África para as Ilhas Canárias, em tempos pré-históricos, provavelmente devido à dessecação do Saara. A ausência destas linhagens presentes em canários atualmente à África sugere movimentos demográficos significativos na área ocidental deste continente.
— Maca-Meyer 2003
| Haplogrupo {{{nome}}} | |
| Tempo de origem | {{{data-origem}}} |
| Lugar de origem | {{{lugar-origem}}} |
| Ancestral | {{{ancestral}}} |
| Mutações definidas | {{{mutações}}} |
- U6a: esta sub-linhagem é a mais difundida, que se estende desde as ilhas Canárias e da península Ibérica para o Chifre da África e o Oriente médio. O sub-haplogrupo tem a sua máxima diversidade no nordeste da África. Idade estimada: 24-27,500 a.C.Tem uma sub-linhagem principal:
- U6b: mostra uma distribuição mais ocidental e fragmentada. Na Península Ibérica, U6b é mais frequente no norte, enquanto U6a no Sul). Ele também foi encontrado em baixas quantidades no Marrocos, a Argélia, o Senegal e a Nigéria. Idade estimada: de 8.500-24,500 a.C.Tem uma sub-linhagem:
- U6b1: encontrado apenas nas Ilhas Canárias e na península ibérica. Idade estimada: c. 6000 a.C.
- U6c: somente encontrado em Marrocos e ilhas Canárias. Idade estimada: 6.000-17,500 a.C.
- U6d: o mais estreitamente relacionado com U6b. Localizado na região do Magrebe, com presença na Europa. Surgiu entre 10.000 e 13.000.C.
U6a, U6b e U6d teem em comum uma mutação basal (16219) que não está presente em U6c, enquanto U6c tem 11 mutações únicas. U6b e U6d compartilham uma mutação (16311) não partilhadas por U6a, que tem três mutações únicas.
U2'3'4'7'8'9[editar | editar código-fonte]
As sub-linhagens U2, U3, U4, U7, U8 e U9 pensa-se agora que são monofiléticas, o seu ancestral comum "U2'3'4'7'8'9" definido pela mutação A1811G surgidos entre 42.000 e 48.000 anos atrás (Behar et al., 2012). Dentro do U2'3'4'7'8'9, U4 e U9 podem ser monofiléticos, tais como "U4 " 9" (mutação T195C!, G499A, T5999C) decorrentes entre 31,000 e 43.000 anos atrás (Behar et al., 2012).
Haplogrupo U2[editar | editar código-fonte]
O haplogrupo U2 é mais comum no sul da Ásia,[41] mas também é encontrado em baixa frequência no centro e no oeste da Ásia, bem como na Europa como U2e (a variedade europeia do U2 é chamada U2e).[42] A frequência geral do U2 no Sul da Ásia é reconhecida, em grande medida, no grupo U2i na Índia, enquanto o haplogrupo U2e, comum na Europa, é totalmente ausente; dado que estas linhagens se distanciaram cerca de 50.000 anos atrás, estes dados têm sido interpretados como uma indicação dum muito baixo fluxo gênico por linha materna entre o sul da Ásia e a Europa ao longo deste período. Cerca da metade do mtADN U na Índia pertence a ramais específicos índios do haplogrupo U2 (U2i: U2a, U2b e U2c). Enquanto o U2 é normalmente encontrado na Índia, também está presente nos Nogais, descendentes de várias tribos mongólicas e túrquicasque faziam parte da Horda de Nogai.[43] Tanto o U2 como o U4 são encontrados nos pobos Ket e Nganasan, nos habitantes indígenas da bacia do rio Yenisei e a Península Taymyr.[44]
As sub-linhagens do U2 são: U2a,[45] U2b,[46] U2c,[47] U2d,[48] e U2e.[49] Com as sub-linhagens específicas da Índia U2a, U2b, e U2c coletivamente referidas como U2i, o haplogrupo eurasiático U2d parece ser uma linhagem irmã com o haplogrupo indiano U2c,[50] enquanto U2e é considerada uma sub-linhagem europeia específica, mas também encontrada no Sul da Índia.[51]
O haplogrupo U2 foi encontrado nos restos de um caçador-recoletor de 30.000 anos atrás de Kostyonki, Oblast de Voronezh, no centro-sul da Rússia Europeia,[52] em restos humanos dum sítio arqueológico da cultura do vaso campaniforme do final do Neolítico em Kromsdorf, Alemanha, de 4800 a 4000 anos atrás,[53] e em restos humanos de Bøgebjerggård no sul da Dinamarca de 2.000 anos de idade. No entanto, o haplogrupo U2 é raro nos dias atuais em países Escandinavos.[54] Os restos de 2.000 anos de idade dum homem eurasiático ocidental do haplogrupo U2e1 foi encontrado no cemitério Xiongnu no norte-leste da Mongólia.[55]
Haplogrupo U3[editar | editar código-fonte]
O haplogrupo U3 é dividido entre dois sub-linhagens: U3a[56] e U3b.
A idade de coalescência para U3a é estimada como de 18.000 26.000 anos atrás, enquanto a idade de coalescência para U3b é estimada como de 18.000 24.000 anos atrás. U3a está localizado na Europa, o Oriente Médio, o Cáucaso e no norte da África. A subclasse quase completamente distribuída por a Europa U3a1, datada de 4000 a 7000 anos atrás, sugere uma expansão relativamente recente (final do Holoceno ou posterior) dessas linhagens na Europa. Há uma sub-linhagem menor U3c (derivada de U3a), representada por um único mtADN Azeri no Cáucaso. U3b é amplamente difundido em todo o Oriente Médio e a região do Cáucaso, e é encontrado principalmente no Irã, o Iraque e o Iêmen, com uma sub-linhagem menor europeia, U3b1b, datada de 2000 a 3000 anos atrás.[57] O haplogrupo U3 é definido pela transição HVR1 A16343G. Ele é encontrado em baixos níveis em toda a Europa (cerca de 1% da população), no Oriente Próximo (cerca de 2,5% da população), e na Ásia Central (cerca de 1% da população). U3 está presente na população suana da região Suanécia (cerca de 4,2% da população) e entre as populações ciganas lituanas, polonesas e espanholas (36-56%)[58][59][60] consistente com a rota de migração desde a Índia para os Balcãs.[61]
A linhagem U3 também é encontrada entre os Berberes Mozabites (e 10,59%), assim como no oásis de El-hayez (2.9%) e Gurna (2.9%) no Egito, e Argelinos em Oran (1.08%-1.25%).[62][63] A rara linhagem U3a está presente entre os tuaregues que habitam Níger (3.23%).[64]
O haplogrupo U3 foi encontrado nalguns dos restos de 6400 anos de idade (U3a) descobertos na caverna de Wadi El‐Makkukh perto de Jericó associados com o período de Calcolítico.[65] O haplogrupo U3 já estava presente no patrimônio genético eurasiático ocidental, cerca de 6000 anos atrás e, provavelmente, também na sua sub-linagem U3a.
Haplogrupo U4[editar | editar código-fonte]
O haplogrup U4 tem sua origem entre 21.000 e 14.000 anos atrás. A sua distribuição está associada com o efeito de gargalo, devido ao último máximo glacial.[66]
U4 foi encontrado em DNA antigo, e é relativamente raro nas populações modernas, embora seja encontrado em proporções substanciais dentro dalgumas populações indígenas do norte da Ásia e do norte da Europa, sendo associado com os restos dos antigos caçadores-recoletores europeus preservado em populações nativas da Sibéria.[67][68][69][70][71] U4 é encontrado na aldeia Nganasan da península de Taymyr, nos Mansi (16.3%), um povo em perigo de extinção, e nos Ket (28.9%) do rio Yenisei.[72] É encontrado na Europa, com as maiores concentrações na Escandinávia e no báltico e é encontrado nos Sami da península escandinava (embora U5b ter uma representação maior).[73][74][75] U4 também é preservado nos Kalash (tamanho da população atual de 3.700) uma tribo única entre os povos indo-arianos do Paquistão, onde U4 ( U4a1) atinge seu maior frequência de 34%.[76][77][78][79][80]
As sub-linhagens de U4 são: U4a,[81] U4b,[82] U4c,[83] e U4d.[84]
O haplogrupo U4 está associado com os antigos caçadores-recoletores europeus e tem sido encontrado em restos de 7.200 6.000 anos atrás, a cultura de cerâmica perfurada em Gotland, Suécia, e em restos de 4.400 a 3.800 anos atrás do sítio arqueológico Damsbo da cultura do vaso campaniforme dinamarquês.[85][86] Os restos identificados como sub-linhagens U4a2 estão associados com a cultura da cerâmica cordada, a qual floresceu de 5200 a 4300 anos atrás na Europa central e oriental e englobou a maior parte da Europa, do norte do continente desde o rio Volga a leste, ao Reno a oeste.[87] O DNA mitocondrial recuperado a partir dos restos de 3.500 a 3.300 anos atrás no sítio arqueológico Bredtoftegård na Dinamarca, associada com a Idade do Bronze Nórdica, que inclui o haplogrupo U4 com 16179T como indicador no seu HVR1 de sub-linhagem U4c1.[88][89][90]
Haplogrupo U7[editar | editar código-fonte]
O haplogrupo U7 é considerado um haplogrupo específicos do mtADN do oeste eurasiático, e é acreditado ter-se originado na área do Mar Negro, ha cerca de 30.000 anos.[91][92][93] Nas populações modernas, U7 é encontrado com frequências baixas no Cáucaso, em tribos siberianas ocidentais, no oeste da Ásia (cerca de 4% no Oriente Médio, com as densidades mais altas de 10% em Iranianos), Ásia do sul (cerca de 12%, no Gujarat, a parte mas a oeste da Índia, enquanto para o conjunto da Índia a sua frequência permanece em torno do 2%, e 5% no Paquistão), e a aldeia Vedda , de Sri Lanka, onde se atinge a maior frequência de 13.33% (sub-linhagem U7a).[94][95] Uma terceira parte do mtADN específico do oeste eurasiático encontrado na Índia aparece nos haplogrupos U7, R2 e W. Especula-se que a imigração em grande escala levou estes haplogrupos mitocondriais para a Índia.
As sub-linhagens de U7 são: U7a (com sub-linhagens U7a1, U7a2, U7a2a, U7a2b)[96] e U7b.
A análise genética dos indivíduos associados com a cultura tardia de Hallstatt , em Baden-Württemberg, na Alemanha consideradas exemplos de "enterros principescos" da Idade do Ferro, os inclui dentro do haplogrupo U7.[97] Foi relatado que o haplogrupo U7 foi encontrado em restos humanos de 1200 anos de idade (datados nas imediações de 834), numa mulher que se acreditava ser dum clã real enterrado com o navio víquingue Oseberg em Noruega.[98] O haplogrupo U7 foi encontrado em restos humanos de 1000 anos de idade (datados de cerca de 1000-1250 d.C.) num cemitério cristão em Kongemarken, Dinamarca. No entanto, U7 é raro entre os escandinavos étnica atuales. A sub-linhagem U7a é especialmente prevalente entre os árabes, constituindo cerca do 30% das linhagens) na província oriental.[99]
A sub-linhagem U7a4 aponta para uma origem no Oriente Próximo. Tem as máximas frequências entre os atuais habitantes do Azerbaijão (26%) e os azeris iranianos (16 a 22%), enquanto que é encontrado no resto do irã em frequências de 2 a 16%, com uma frequência moderada de 14 a 17%, na parte ocidental do Iraque e 1-19% na península arábica. Frequências mais baixas também podem ser encontradas no Turcomenistão (2-10%), na parte ocidental do Cazaquistão (2-10%), Síria (3-7%), Uzbequistão (2-6%), centro da península da Anatólia (2-4%), o sul do Punjab (1-2%) e Afeganistão (0-2%). U7a4 também tem sido observada num espécime na Toscana e outros dois na Rússia Europeia. Presume-se que esta sub-linhagem foi trazida para a Toscana pela civilização Etrusca. Acredita-se que os atuais habitantes da Toscana teem preservado algo desta herdança genética herdada.[100]
Haplogrup Um8[editar | editar código-fonte]
- U8a: Os Bascos são os que teem mais filogenia ancestral na Europa pel haplogrupo mitocondrial U8a. Este é um raro subgrupo de U8, colocando a origem desta linhagem basco no Paleolítico Superior. A falta de linhagem U8a na África sugere que seus antepassados podem ter sua origem no oeste da Ásia.
- U8b: Esta linhagem foi encontrada na Itália e Jordânia.
- O haplogrupo K faz parte duma fração considerável das linhagens da Europa e da Ásia ocidental. Este agora é conhecido como uma sub-linhagem do haplogrup U8b K, e acredita-se ter surgido no nordeste da Itália. O haplogrup UK mostrar alguns sinais de ser altamente protetor contra a progressão da AIDS.
Haplogrup U9[editar | editar código-fonte]
O Haplogrup U9 é uma rara linhagem na filogenia do mtADN, encontrada apenas recentemente nalgumas populações do Paquistão (Quintana-Murci et al., 2004). A sua presença na Etiópia e Iêmen, juntamente com algumas linhagens M específicas da Índia dentro da amostra iemenita, aponta para o fluxo gênico ao longo da costa do Mar da Arábia. Os haplogrupos U9 e U4 compartilham duas mutações comuns às raízes de sua filogenia. É interessante que, no Paquistão, U9 ocorre freqüentemente só entre os chamados Makrani. Nesta população, particularmente as linhagens específicas de partes do leste da África ocorrem tão frequentemente como 39%, o que sugere que as linhagens U9 no Paquistão pode ter uma origem nesta área (Quintana-Murci et al., 2004). Independentemente de qual parte da costa do Mar Arábico pode ter sido a origem da U9, a sua distribuição na Etiopía-Arábia do sul - bacia do Indo sugere que a diversificação da sub-linhagem U4 pode ter ocorrido em regiões muito longe da atual área de maior diversidade e frequência do haplogrupo U4—Europa do leste e Sibéria ocidental.[101]
Ver também[editar | editar código-fonte]
- Teste de ADN genealógico
- Genealogia genética
- Genética mitocondrial humana
- Genética da população
Referências
- ↑ «Carriers of mitochondrial DNA macrohaplogroup R colonized Eurasia and Australasia from a southeast Asia core area».
- ↑ Schuenemann, Verena J., et al.
- ↑ Karmin, Monika.
- ↑ Bryan Sykes.
- ↑ «Maternal Ancestry».
- ↑ [1], A Revised Timescale for Human Evolution Based on Ancient Mitochondrial Genomes, Fu et al. 2013
- ↑ [2]
- ↑ «The mitochondrial lineage U8a reveals a Paleolithic settlement in the Basque country».
- ↑ «Updated comprehensive phylogenetic tree of global human mitochondrial DNA variation».
- ↑ [3], FamilyTreeDNA - mtDNA haplogroup U1 project
- ↑ [4], Phylogeny of mt-hg U3, Ron Scott 2010
- ↑ «Relic excavated in western India is probably of Georgian Queen Ketevan».
- ↑ Abstract Book of the IUAES Inter-Congress 2016 - A community divided?
- ↑ «Genetic Heterogeneity in Algerian Human Populations».
- ↑ 30,248.3 ± 5,330.5; CI=95% (Behar et al., 2012) haplogroup.org
- ↑ The Genographic Project at National Geographic
- ↑ «Mitochondrial DNA haplogroups influence AIDS progression».
- ↑ Age: 1,398.1 ± 1,872.3; CI=95% (Behar et al., 2012) haplogroup.org
- ↑ 22,794.0 ± 3,590.3; CI=95% (Behar et al., 2012) haplogroup.org
- ↑ «The Complex and Diversified Mitochondrial Gene Pool of Berber Populations».
- ↑ Age: 15,529.7 ± 4,889.9; CI=95% (Behar et al., 2012) haplogroup.org
- ↑ «Y-chromosomal diversity in the population of Guinea-Bissau: a multiethnic perspective».
- ↑ «Saami and Berbers--an unexpected mitochondrial DNA link».
- ↑ Malyarchuk B, Derenko M, Grzybowski T, Perkowa M, Rogalla U, Vanecek T, Tsybovsky I «The Peopling of Europe from the Mitochondrial Haplogroup U5 Perspective».
- ↑ 20,040.2 ± 3,208.6; CI=95% (Behar et al., 2012) haplogroup.org
- ↑ 14,938.0 ± 3,656.4; CI=95% (Behar et al., 2012) haplogroup.org
- ↑ Boris Malyarchuk et al., "The Peopling of Europe from the Mitochondrial Haplogroup U5 Perspective", PLoS One. 2010; 5(4): e10285. 10.1371/journal.pone.0010285. "subcluster U5b2a is characterized by a predominantly central European distribution, since a large number of U5b samples from Poland, Slovakia and the Czech Republic fall into this subcluster.
- ↑ Age: 14,676.9 ± 2,716.6; CI=95% (Behar et al., 2012) haplogroup.org
- ↑ Age: 12,651.1 ± 5,668.2; CI=95% (Behar et al., 2012) haplogroup.org
- ↑ 4,015.9 ± 3,898.7; CI=95% (Behar et al., 2012) haplogroup.org
- ↑ The Young Man of Byrsa specimen dates from the late 6th century BCE, and his lineage is believed to represent early gene flow from Iberia to the Maghreb.
- ↑ «Mitochondrial haplogroup U5b3: a distant echo of the epipaleolithic in Italy and the legacy of the early Sardinians».
- ↑ Soficaru, A. D., Dobos, A. & Trinkaus, E. Early modern humans from the Peştera Muierii, Baia de Fier, Romania.
- ↑ Hervella, M.; Svensson, E. M.; Alberdi, A.; Günther, T. «The mitogenome of a 35,000-year-old Homo sapiens from Europe supports a Paleolithic back-migration to Africa».
- ↑ «Joining the Pillars of Hercules: mtDNA Sequences Show Multidirectional Gene Flow in the Western Mediterranean» (pdf).
- ↑ «Mitochondrial DNA transit between West Asia and North Africa inferred from U6 phylogeography».
- ↑ Cerezo, María, et al.
- ↑ «The history of the North African mitochondrial DNA haplogroup U6 gene flow into the African, Eurasian and American continents».
- ↑ «Early Holocenic and Historic mtDNA African Signatures in the Iberian Peninsula: The Andalusian Region as a Paradigm».
- ↑ «Genetic Patterns of Y-chromosome and Mitochondrial DNA Variation, with Implications to the Peopling of the Sudan».
- ↑ [5], Most of the extant mtDNA boundaries in South and Southwest Asia were likely shaped during the initial settlement of Eurasia by anatomically modern humans, Mait Metspalu, et al. 2004
- ↑ [6], Distribution of Mitochondrial DNA Macrohaplogroup N inIndia with Special Reference to Haplogroup R and its Sub-Haplogroup U, Vasulu, et al. 2008
- ↑ «[Phylogeografic analysis of mitochondrial DNA Nogays: the high level of mixture of maternal lineages from Eastern and Western Eurasia]».
- ↑ «[Mitochondrial DNA variation in Kets and Nganasans and the early peoples of Northern Eurasia]».
- ↑ [7], Phylogeny of mt-hg U2a, Ron Scott 2009
- ↑ [8], Phylogeny of mt-hg U2b, Ron Scott 2010
- ↑ [9], Phylogeny of mt-hg U2c, Ron Scott 2009
- ↑ [10], Phylogeny of mt-hg U2d, Ron Scott 2009
- ↑ [11], Phylogeny of mt-hg U2e, Ron Scott 2010
- ↑ «Mitochondrial haplogroup U2d phylogeny and distribution».
- ↑ [12]
- ↑ [13], BBC News: DNA analysed from early European, 2010
- ↑ «Emerging genetic patterns of the European Neolithic: perspectives from a late Neolithic Bell Beaker burial site in Germany».
- ↑ «Rare mtDNA haplogroups and genetic differences in rich and poor Danish Iron-Age villages».
- ↑ [14], A Western Eurasian Male Is Found in 2000-Year-Old Elite Xiongnu Cemetery in Northeast Mongolia, Kijeong Kim, et al. 2010
- ↑ [15], Phylogeny of mt-hg U3, Ron Scott 2010
- ↑ [16], Complete Mitochondrial DNA Diversity in Iranians, Derenko et al. 2013
- ↑ «Tracing European founder lineages in the Near Eastern mtDNA pool».
- ↑ «Where west meets east: the complex mtDNA landscape of the southwest and Central Asian corridor».
- ↑ «Mitochondrial DNA diversity in the Polish Roma».
- ↑ [17], Reconstructing the Indian Origin and Dispersal of the European Roma: A Maternal Genetic Perspective, Mendizabal et al. 2011
- ↑ «Near Eastern Neolithic Genetic Input in a Small Oasis of the Egyptian Western Desert».
- ↑ Stevanovitch, A.; Gilles, A.; Bouzaid, R. Kefi; F. Paris, R. P. Gayraud, J. L. Spadoni, F. El-Chenawi, E. Béraud-Colomb «Mitochondrial DNA Sequence Diversity in a Sedentary Population from Egypt».
- ↑ «Linking the sub-Saharan and West Eurasian gene pools: maternal and paternal heritage of the Tuareg nomads from the African Sahel».
- ↑ [18] Arquivado em 3 de março de 2016, no Wayback Machine., Ancient mtDNA sequences and radiocarbon dating of human bones from the Chalcolithic caves of Wadi El‐Makkukh,Mediterranean Archaeology and Archaeometry, Salamon et al. 2010
- ↑ [19], Tracing European Founder Lineages in the Near Eastern mtDNA Pool, Richards et al. 2000
- ↑ [20], TNeolithic pots and potters in Europe: the end of ‘demic diffusion’ migratory mode, Mihael Budja 2013
- ↑ [21], The Peopling of Europe from the Mitochondrial Haplogroup U5 Perspective, Malyarchuk et al. 2010
- ↑ [22], Ancient DNA Reveals Prehistoric Gene-Flow from Siberia in the Complex Human Population History of North East Europe, Sarkissian et al. 2013
- ↑ [23], Ancient DNA from European Early Neolithic Farmers Reveals Their Near Eastern Affinities, Haak et al. 2010
- ↑ [24], Traces of Early Eurasians in the Mansi of Northwest Siberia Revealed by Mitochondrial DNA, Derbeneva et al. 2002
- ↑ [25], Mitochondrial Genome Diversity in Arctic Siberians, with Particular Reference to the Evolutionary History of Beringia and Pleistocenic Peopling of the Americas, Volodko et al. 2008
- ↑ [26], Maternal Ancestry: The clan of Ulrike, Oxford Ancestors, 2013
- ↑ Population genetics of the Sami, Population genetics of the Sami
- ↑ [27], Oxford Ancestors Ltd.
- ↑ [28], Pakistan Today: Safeguarding Kalash heritage, Bashir, 2012
- ↑ [29], U4a1 Genbank Sequences, Ian Logan 2014
- ↑ Quintana-Murci, Lluís «Where West Meets East: The Complex mtDNA Landscape of the Southwest and Central Asian Corridor. Arquivado em 23 de novembro de 2009, no Wayback Machine.
- ↑ «Where west meets east: the complex mtDNA landscape of the southwest and Central Asian corridor».
- ↑ [30], U4 Haplogroup: A blog dedicated to the U4 Haplogroup - The Kalash,
- ↑ [31], Phylogeny of mt-hg U4a, Ron Scott 2011
- ↑ [32], Phylogeny of mt-hg U4b, Ron Scott 2011
- ↑ [33], Phylogeny of mt-hg U4c, Ron Scott 2011
- ↑ [34], Phylogeny of mt-hg U4d, Ron Scott 2010
- ↑ [35], Eupedia: Prehistoric European DNA - mtDNA & Y-DNA Haplogroup Frequencies by Period
- ↑ [36], Genetic Diversity among Ancient Nordic Populations, Melchior et al 2010
- ↑ [37], Mitochondrial DNA Phylogeny in Eastern and Western Slavs, Malyarchuk et al. 2008
- ↑ [38] Arquivado em 22 de janeiro de 2017, no Wayback Machine., Ancestral Journeys: Ancient Western Eurasian DNA of the Copper and Bronze Ages, 2009
- ↑ [39], Genetic Diversity among Ancient Nordic Populations: Table 1.
- ↑ [40], A Comprehensive hg U Mutation List, Ron Scott 2010
- ↑ [41], Most of the extant mtDNA boundaries in South and Southwest Asia were likely shaped during the initial settlement of Eurasia by anatomically modern humans.
- ↑ [42], The Oseberg women and the Gokstad man retain their genetic secrets, Kulturhistorisk Museum - University of Oslo
- ↑ [43], U7 Haplogroup Mitochondrial DNA Project
- ↑ «mtDNA analysis of human remains from an early Danish Christian cemetery».
- ↑ [44], Mitochondrial DNA history of Sri Lankan ethnic people: their relations within the island and with the Indian subcontinental populations, Ranaweera et al. 2013
- ↑ [45], Phylogeny of mt-hg U7, Ron Scott 2010
- ↑ [46], An ancient DNA perspective on the Iron Age "princely burials" from Baden-Württemberg Germany, Lee et al. 2012
- ↑ Berglund, Nina.
- ↑ «The Saudi Arabian Genome Reveals a Two Step Out-of-Africa Migration».
- ↑ «Mitogenomes from The 1000 Genome Project Reveal New Near Eastern Features in Present-Day Tuscans».
- ↑ Kivisild, Toomas; Reidla, Maere; Metspalu, Ene; Rosa, Alexandra «Ethiopian mitochondrial DNA heritage: tracking gene flow across and around the gate of tears».
Ligações externas[editar | editar código-fonte]
- «A "Copernican" reassessment of the human mitochondrial DNA tree from its root». Am J Hum Ginete, quer. 90, 4, 2012, pàg. 675–684. DOI: 10.1016/j.ajhg.2012.03.002. PMC: 3322232. PMID: 22482806.
- Geral
- Ian Logan's Mitochondrial DNA Site
- Mannis Van Oven's Phylotree
- «Most of the extant mtDNA boundaries in south and southwest Asia were likely shaped during the initial settlement of Eurasia by anatomically moderno humanos». BMC Genetics, quer. 5, 1, August 2004, pàg. 26. DOI: 10.1186/1471-2156-5-26. PMC: 516768. PMID: 15339343.
- Haplogrupo U
- Haplogrupo U1
- The mtDNA Haplogroup U1 Project Family Tree DNA
- Haplogroupo U2
- The mtDNA Haplogroup U2 Project Family Tree DNA
- Haplogrupo U4
- Haplogrupo U5
- The mtDNA Haplogroup U4 ProjectFamily Tree DNA
- Brian Hamman's Clan Ursula Website U5
- Haplogrupo U6
- The mtDNA Haplogroup U6 Project Family Tree DNA
- The U6b Website
- Haplogrupo U7
- The U7 Haplogroup Mitochondrial DNA ProjectFamily Tree DNA
- Haplogrupo U8
- The U8 Haplogroup Mitochondrial DNA Project Family Tree DNA
- Haplogrupo U9
- The U9 Haplogroup Mitochondrial DNA Project Family Tree DNA
