Archaea
| Archaea | |||
|---|---|---|---|
| Ocorrência: 3.5–0 Ga. | |||
| Classificação científica | |||
| |||
| Reinos | |||
| Sinónimos | |||
| |||
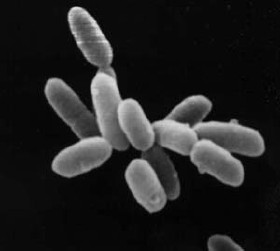
Archaea (singular: archaeon; do grego: ἀρχαῖος; archaĩos, antigo),[4][5] em português: arquea,[6][7][8] arqueiaAO 1990 ou arquaia,[8] é um domínio que agrupa microrganismos unicelulares procariontes (i.e. sem núcleo celular), morfologicamente semelhantes a bactérias, mas genética e bioquimicamente tão distintas destas como dos eucariontes. Estes organismos foram inicialmente classificadas como um tipo ancestral de bactérias, recebendo o nome Archaebacteria (no reino Archaebacteria), classificação agora considerada obsoleta, tendo o grupo sido renomeado para esclarecer que os seus membros estão filogeneticamente mais próximos dos eucariontes (organismos com núcleo celular) do que das bactérias.[9] Archaea, por outro lado, tem (como as bactérias) moléculas de DNA independentes (cromossomas circulares)[10] presentes no citoplasma, sem membrana envolvente, como equivalente nuclear. As células dos Archaea apresentam propriedades únicas que as separam dos outros dois domínios (Bacteria e Eukaryota), estando o grupo presentemente divididos em três reinos e com um número crescente de filos reconhecidos. A taxonomia do grupo é difícil porque a maioria das espécies conhecidas ainda não foi isolada em laboratório, tendo sido detetada apenas pelas sequências genéticas presentes em amostras ambientais. Não se sabe se estes organismos são capazes de produzir endósporos. Não é conhecido neste agrupamento qualquer patógeno que afete humanos.
Descrição[editar | editar código-fonte]
As células dos membros do agrupamento taxonómico Archaea apresentam propriedades únicas que as distinguem dos domínios Bacteria e Eukaryota. Apesar de presentemente os arqueias estarem divididas em vários filos reconhecidos, a sua classificação é difícil, pois a maioria não foi isolada em laboratório e somente foi detetada pela análise de sequências genómicas em amostras de DNA ambiental.
As arqueias partilham características que podem ser encontradas tanto entre os eucariotas como entre as bactérias. A título de exemplo, as arqueias possuem geralmente um único cromossoma circular, à semelhança das bactérias, mas os seus cromossomas podem ter mais do que uma origem de replicação, fenómeno que se pensava estar presente apenas nos eucariotas.[11]
Morfologia e estrutura[editar | editar código-fonte]

Também no que respeita à morfologia, embora as Archaea e as bactérias sejam geralmente semelhantes em tamanho e forma, algumas espécies de Archaea apresentam morfologia muito diferente, como sejam as células planas e quadradas de Haloquadratum walsbyi.[12] Ainda assim, apesar das semelhanças morfológicas com as bactérias, os Archaea possuem genes e várias vias metabólicass que estão mais intimamente relacionadas com as dos eucariotas, nomeadamente no que concerne as enzimas envolvidas na transcrição e tradução genómica. Outros aspectos da bioquímica dos Archaea são únicos, como a dependência de éteres lipídicos na estruturação das membranas celulares,[13] incluindo a presença de di-éteres do grupo arqueol (ou archaeol).
Morfologia[editar | editar código-fonte]
As células dos organismos do grupo Archaea têm um tamanho que varia de 0,1 micrómetros (μm) até 15 μm de diâmetro, e ocorrem numa variedade de formas, normalmente como esferas, bastonetes, espirais ou placas.[14] Outras morfologias nos Thermoproteota incluem células lobadas de forma irregular em Sulfolobus, filamentos em forma de agulha que têm menos que metade de um micrómetro de diâmetro em Thermofilum, e também bastonetes quase perfeitamente regulares em Thermoproteus e Pyrobaculum.[15] Existe mesmo uma espécie de Archaea com células de forma achatada e quase quadrangular chamada Haloquadra walsbyi que habita charcos hipersalinos.[16]
Estas formas pouco usuais são provavelmente mantidas quer pelas suas paredes celulares quer pelo citoesqueleto procariota. Proteínas relacionadas com os componentes do citoesqueleto de outros organismos ocorrem em Archaea,[17] e filamentos são formados dentro das suas células,[18] mas em contraste com outros organismos, estas estruturas celulares estão pouco estudadas nas arqueias.[19] Nos géneros Thermoplasma e Ferroplasma a falta de uma parede celular significa que as células têm formas irregulares e podem-se assemelhar a amebas.[20]
Algumas espécies de Archaea formam agregados ou filamentos de células com 200 μm de comprimento,[14] e estes organismos podem ser membros proeminentes da comunidade de micróbios que compõem os biofilmes.[21] Um exemplo extremo é a espécie Thermococcus coalescens, em que agregados de células se juntam formando células únicas gigantes.[22]
Uma particularmente elaborada forma de colónia multicelular é produzida por Archaea do género Pyrodictium, em que as células produzem conjuntos de longos e finos tubos ocos denominados cannulae que emergem da superfície das células e as interligam formando uma densa colónia aglomerada numa forma arbuscular.[23] A função destas cannulae é ainda desconhecida, mas poderão permitir que as células comuniquem ou troquem nutrientes com os seus vizinhos.[24]
As colónias podem também ser produzidas por associação ente diferentes espécies, incluindo organismos que não integram Archaea. Por exemplo, na comunidade de "cordão de pérolas" que foi descoberta em 2001 num pântano na Alemanha, colónias esbranquiçadas e arredondadas de uma nova espécie de Archaea do filo Euryarchaeota estão espaçadas ao longo de finos filamentos que podem ter até 15 cm de comprimento. Estes filamentos são formados de uma espécies particular de bactéria.[25]
Os membros de Archaea são semelhantes às bactérias na sua estrutura celular geral, mas a composição e organização de algumas dessas estruturas diferenciam as Archaea. Assim como as bactérias, as Archaea não possuem membranas internas, de modo que suas células não contêm organelos.[26] Estes organismos também se assemelham a bactérias em outros importantes aspectos: (1) a sua membrana celular é geralmente limitada por uma parede celular; e (2) nadam pelo uso de um ou mais flagelos.[27] Na estrutura geral, as Archaea são mais semelhante a bactérias gram-positivas, já que a maioria tem uma única membrana plasmática e parede celular e não apresentam espaço periplasmático. A exceção a esta regra geral é o género arqueano Ignicoccus, que possui um periplasma particularmente grande que contém vesículas ligadas à membrana e é envolvido por uma membrana externa.[28]
Parede celular e arcaelo[editar | editar código-fonte]
A maioria dos Archaea (mas não os géneros Thermoplasma e Ferroplasma) possui uma parede celular.[20] Na maioria das archaea, a parede é montada a partir de proteínas da camada superficial, que formam uma camada S (S-layer).[29]
A camada S é uma matriz rígida de moléculas de proteína que cobrem a parte externa da célula (como uma cota de malha numa armadura).[30] A presença dessa camada fornece proteção química e física e pode impedir que macromoléculas entrem em contato com a membrana celular.[31]
Ao contrário das bactérias, os Archaea não possuem peptidoglicano nas suas paredes celulares.[32] O grupo das Methanobacteriales apresenta paredes celulares contendo pseudopeptidoglicano, que se assemelha ao peptidoglicano eubacteriano em morfologia, função e estrutura física, mas é distinto na estrutura química. Neste composto faltam os D-aminoácidos e o ácido N-acetilmurâmico, estando o último substituído por ácido N-acetiltalosaminurónico.[31]
Os flagelos arqueanos são conhecidos como arcaelos (neolatim: archaellum, pl. archaella), que operam como os flagelos bacteriano, com longas caudas acionadas por motores rotatórios na base. Esses motores são alimentados por um gradiente de protões através da membrana. Contudo, embora morfologicamente similares, os arcaelos são notavelmente diferentes em composição e desenvolvimento em relação aos flagelos bacterianos.[27]
Os dois tipos de flagelos evoluíram de ancestrais diferentes. O flagelo bacteriano compartilha um ancestral comum com o sistema de secreção tipo III,[33][34] enquanto os flagelos de Archaea parecem ter evoluído de bactérias portadoras de pili do tipo IV.[35] Em contraste com o flagelo bacteriano, que é oco e montado por subunidades que se movem a partir de um poro central até a ponta do flagelo, os flagelos arqueanos são sintetizados pela adição de subunidades na base.[36]
Membrana celular[editar | editar código-fonte]

As membranas dos Archaea são constituídas por moléculas que são distintamente diferentes daquelas que estão presentes em todas as outras formas de vida, mostrando que as arqueias estão apenas distantemente relacionadas com as bactérias e os eucariotas.[37] Em todos os organismos, a membrana celular é constituída por moléculas conhecidas como fosfolípidos. Essas moléculas possuem uma parte polar que se dissolve em água (a "cabeça" de fosfato) e uma parte não polar, "gordurosa", que é hidrófuga (a cauda lipídica). Essas partes diferentes são conectadas por uma porção de glicerol. Na água, os fosfolipídios agrupam-se com as "cabeças" fosfatadas voltadas para a água e as caudas lipídicas voltadas para longe dela. A principal estrutura nas membranas celulares é uma dupla camada desses fosfolipídios, que é designada por bicamada lipídica.[38]
Os fosfolipídios de Archaea são incomuns de quatro maneiras:
- Apresentam membranas compostas de glicerol-éter, enquanto as bactérias e os eucariotas têm membranas compostas principalmente de lípidos glicerol-éster.[39] A diferença é o tipo de ligação que une os lipídios à porção glicerol; os dois tipos são mostrados em amarelo na figura à direita. Em lípidos com éster, esta é uma ligação éster, enquanto em lípidos com éter é uma ligação éter.[40]
- A estereoquímica da porção de glicerol arqueana é a imagem espelhada daquela encontrada em outros organismos. A porção de glicerol pode ocorrer em duas formas que são imagens espelhadas uma da outra, chamadas enantiómeros. Assim como uma mão direita não cabe facilmente numa luva para canhotos, os enantiómeros de um tipo geralmente não podem ser usados ou produzidos por enzimas adaptadas para o outro. Os fosfolipídios arqueais são construídos sobre uma espinha dorsal de sn-glicerol-1-fosfato, que é um enantiómero de sn-glicerol-3-fosfato, a espinha dorsal fosfolipídica encontrada em bactérias e eucariotas. Isso sugere que Archaea usa enzimas totalmente diferentes para sintetizar fosfolipídios em comparação com bactérias e eucariotas. Essas enzimas foram desenvolvidas muito cedo na história da vida, indicando uma divisão precoce em relação aos outros dois domínios.[37]
- As caudas lipídicas arqueais diferem das de outros organismos por serem baseadas em longas cadeias isoprenoides com múltiplas ramificações laterais, às vezes com anéis de ciclopropano ou ciclohexano.[41] Em contraste, os ácidos gordos presentes nas membranas de outros organismos têm cadeias rectas sem ramificações laterais ou anéis. Embora os isoprenoides desempenhem um papel importante na bioquímica de muitos organismos, apenas os Archaea os utilizam para produzir fosfolípidos. Essas cadeias ramificadas podem ajudar a evitar que as membranas arqueais se esvaziem em altas temperaturas.[42]
- Em alguns Archaea, a bicamada lipídica é substituída por uma monocamada. Com efeito, os Archaea fundem as caudas de duas moléculas de fosfolipídeos numa única molécula com duas cabeças polares (um bolaanfífilo ou bolaamphiphile); esta fusão pode tornar as membranas mais rígidas e mais capazes de resistir a ambientes agressivos.[43] Por exemplo, os lípidos presentes em Ferroplasma são deste tipo, o que ajudará a sobrevivência deste organismo no seu habitat altamente ácido.[44]
Metabolismo[editar | editar código-fonte]

Archaea exibe uma grande variedade de reações químicas em seu metabolismo e usa muitas fontes de energia. Essas reações são classificadas em grupos nutricionais, dependendo das fontes de energia e carbono. Alguns Archaea obtêm energia de compostos inorgânicos, como enxofre ou amónia (são organismos quimiotróficos). Entre estes incluem-se organismos nitrificadores, metanogénicos e oxidadores anaeróbicos do metano.[46]
Nessas reações, um composto cede eletrões a outro (numa reação redox), libertando energia para abastecer as atividades da célula. Um composto atua como um dador de eletrões e outro como um recetor de eletrões. A energia libertada é utilizada para gerar trifosfato de adenosina (ATP) por meio da quimiosmose, o mesmo processo básico que ocorre na mitocôndria das células eucarióticas.[47]
Outros grupos de Archaea usam a luz solar como fonte de energia (são fototróficos), mas a fotossíntese geradora de oxigénio não ocorre em nenhum desses organismos.[47] Muitas vias metabólicas básicas são compartilhadas entre todas as formas de vida; por exemplo, Archaea usa uma forma modificada de glicólise (a via Entner–Doudoroff) e um ciclo do ácido cítrico completo ou parcial.[48] Essas semelhanças com outros organismos provavelmente refletem tanto as primeiras origens na história da vida quanto o seu alto nível de eficiência.[49]
| Tipo nutricional | Fonte de energia | Fonte de carbono | Exemplos |
|---|---|---|---|
| Fototrofia | Radiação solar | Compostos orgânicos | Halobacterium |
| Litotrofia | Compostos inorgânicos | Compostos orgânicos ou fixação de carbono | Ferroglobus, Methanobacteria ou Pyrolobus |
| Organotrofia | Compostos orgânicos | Compostos orgânicos ou fixação de carbono | Pyrococcus, Sulfolobus ou Methanosarcinales |
Alguns Euryarchaeota são metanogénicos (Archaea que produzem metano como resultado do seu metabolismo) vivendo em ambientes anaeróbicos, como pântanos. Essa forma de metabolismo evoluiu cedo e é até possível que o primeiro organismo de vida livre tenha sido um organismo metanogénico.[50] Na metanogénese, uma reação comum envolve o uso de dióxido de carbono como um recetor de eletrões para oxidar hidrogénio. A metanogénese envolve uma variedade de coenzimas que são exclusivas dessas Archaea, como a coenzima M e o metanofurano.[51]
Outros compostos orgânicos, como álcoois, ácido acético ou ácido fórmico, são usados como aceitadores de eletrões alternativos pelos metanogénicos. Essas reações são comuns em Archaea que têm como habitat o tracto gastrointestinal de mamíferos. O ácido acético também é decomposto em metano e dióxido de carbono diretamente, por arqueias acetotróficas. Esses acetotrofos são Archaea da ordem Methanosarcinales e constituem a maior parte das comunidades de microrganismos produtores de biogás.[52]
Outras Archaea usam CO2 da atmosfera como fonte de carbono, num processo chamado fixação de carbono (são organismos autotróficoss). Este processo envolve uma forma altamente modificada do ciclo de Calvin[53] ou, em alternativa, a via metabólica designada por ciclo 3-hidroxipropionato/ 4-hidroxibutirato.[54]
Os Thermoproteota também usam o ciclo reverso de Krebs, enquanto os "Euryarchaeota" também usam a via redutora do acetil-CoA (via Wood–Ljungdahl).[55] A fixação de carbono é alimentada por fontes de energia inorgânicas. Nenhuma Archaea conhecida realiza fotossíntese[56] (Halobacterium é o único arconte fototrófico conhecido, mas usa um processo alternativo à fotossíntese). As fontes de energia usada pelos Archaea são extremamente diversas, e vão desde a oxidação de amónia pelos Nitrosopumilales[57][58] à oxidação de sulfeto de hidrogénio ou enxofre elementar por espécies de Sulfolobus, usando oxigénio ou iões metálicos como recetores de eletrões.[47]
As arqueias fototróficas usam a luz para produzir energia química na forma de ATP. Nas Halobacteria, bombas iónicas ativadas por luz, como a bacteriorrodopsina e a halorrodopsina, geram gradientes iónicos bombeando iões para fora e para dentro da célula através da membrana plasmática. A energia armazenada nesse gradiente eletroquímico é então convertida em ATP pela ATP sintase.[14] Este processo é uma forma de fotofosforilação. A capacidade dessas bombas de luz para mover iões através das membranas depende de mudanças de luz na estrutura de um cofator retinol incrustado no centro da proteína.[59]
Genética[editar | editar código-fonte]

Estrutura do genoma e transcrição[editar | editar código-fonte]
Os Archaea geralmente apresentam um único cromossomo circular,[61] que atinge 5 751 492 pares de bases em Methanosarcina acetivorans,[62] o maior genoma arqueano conhecido. O minúsculo genoma de apenas 490 885 pares de bases de Nanoarchaeum equitans tem um décimo desse tamanho e é o menor genoma arqueano conhecido, no quel se estima contenha apenas 537 genes que codificam proteínas.[63]
Pedaços independentes menores de DNA, designados por plasmídeos, também são encontrados em Archaea. Os plasmídeos podem ser transferidos entre células por contato físico, em num processo que pode ser semelhante à conjugação bacteriana.[64][65]
Os membros de Archaea são geneticamente distintos de bactérias e eucariontes, com até 15% das proteínas codificadas por genoma exclusivo do domínio, embora a maioria desses genes únicos não tenha função conhecida.[66]
A maioria da fração das proteínas únicas que têm uma função identificada pertence aos Euryarchaeota e está envolvida na metanogénese. As proteínas que as arqueias, as bactérias e os eucariotas compartilham formam um núcleo comum de função celular, relacionado principalmente coma transcrição, tradução e metabolismo dos nucleotídeos.[67] Outras características arqueanas diferenciadoras são a organização dos genes de função relacionada, tais como as enzimas que catalisam etapas na mesma via metabólica em novos operões e grandes diferenças em genes tRNA e suas aminoacil-tRNA sintetases.[67]
A transcrição em Archaea é mais semelhante à transcrição eucariótica do que à transcrição bacteriana, com a RNA polimerase arqueana a ser do ponto de vista molecular muito próxima da sua equivalente nos eucariotas,[61] enquanto a tradução em Archaea mostra sinais de equivalentes bacterianos e eucarióticos.[68]
Embora Archaea tenha apenas um tipo de RNA polimerase, a sua estrutura e função na transcrição parecem ser próximas às da RNA polimerase II eucariótica, com montagens de proteínas semelhantes (os factores de transcrição gerais) direcionando o ligação da RNA polimerase a um gene promotor,[69] mas outros fatores de transcrição arqueanos estão mais próximos daqueles que são encontrados em bactérias.[70]
A modificação pós-transcricional é mais simples do que nos eucariotas, uma vez que a maioria dos genes arqueanos não possui íntrons, embora existam muitos íntrons nos seus genes do RNA de transferência e RNA ribossomal,[71] e íntrons podem ocorrer em alguns genes que codificam proteínas.[72][73]
Transferência de genes e troca genética[editar | editar código-fonte]
A espécie Haloferax volcanii, uma arqueia halofílica extrema, forma pontes citoplasmáticas entre as células que parecem ser usadas para a transferência de DNA de uma célula para outra em ambas direções.[74]
Quando as arqueias hipertermofílicas Sulfolobus solfataricus[75] e Sulfolobus acidocaldarius[76] são expostas a irradiação UV prejudicial ao DNA ou aos agentes teratogénicos conhecidos por bleomicina ou mitomicina C, é induzida a agregação celular espécie-específica. A agregação em Sulfolobus solfataricus não pode ser induzida por outros factores físicos, como mudança de pH ou temperatura,[75] sugerindo que a agregação é induzida especificamente por danos no DNA.
Também foi demonstrado que que a agregação celular induzida por UV medeia a troca de marcadores cromossómicos com alta frequência em S. acidocaldarius.[76] As taxas de recombinação excederam as de culturas não induzidas em até três ordens de grandeza, o que indica que a agregação celular aumenta a transferência de DNA espécie-específica entre as células de Sulfolobus a fim de permitir o aumento da capacidade de reparação do DNA danificado por meio de recombinação homóloga.[75][77][76]
Essa resposta pode ser uma forma primitiva de interação sexual semelhante aos sistemas de transformação bacteriana mais bem estudados, que também estão associados à transferência de DNA espécie-específica entre células, levando ao reparo recombinatório homólogo de danos sofridos pelo DNA.[78]
Vírus que infectam Archaea[editar | editar código-fonte]
Archaea é o alvo de vários vírus que integram uma virosfera diversa distinta dos vírus bacterianos e eucarióticos. Os tipos conhecidos até agora foram organizados em 15-18 famílias baseadas em DNA, mas várias estirpes permanecem não isoladas e aguardam classificação.[79][80][81] Essas famílias podem ser informalmente divididas em dois grupos: vírus arqueia-específicos e cosmopolitas. Vírus específicos de Archaea têm como alvo apenas espécies arqueanas e atualmente incluem 12 famílias.
Numerosas estruturas virais únicas e não identificadas anteriormente foram observadas neste grupo, incluindo: vírus em forma de garrafa, em forma de fuso, em forma de espiral e em forma de gota.[80] Embora os ciclos reprodutivos e os mecanismos genómicos de espécies específicas de Archaea possam ser semelhantes a outros vírus, estes possuem características únicas que foram desenvolvidas especificamente devido à morfologia das células hospedeiras que infectam.[82] Os mecanismos de libertação destes vírus diferem dos de outros fagos. Os bacteriófagos geralmente passam por vias líticas, vias lisogénicas ou (raramente) uma mistura dos dois tipos de libertação do material genético na célula hospedeira.[83]
A maioria das estirpes virais específicas de Archaea mantém uma relação estável, um tanto lisogénica, com os seus hospedeiros, aparecendo como uma infecção crônica. Isso envolve a produção e libertação gradual e contínua de viriões sem matar a célula hospedeira.[84]
Foi levantada a hipótese de que os fagos dos Archaea com cauda tiveram origem a partir de bacteriófagos capazes de infetar espécies de Haloarchaea.[85] Se a hipótese estiver correta, pode-se concluir que outros vírus de DNA de cadeia dupla que compõem o restante do grupo específico de Archaea são um grupo exclusivo na comunidade viral global. Por outro lado, os altos níveis de transferência horizontal de genes, as rápidas taxas de mutação nos genomas virais e a falta de sequências gênicas universais levaram os investigadores a perceber o caminho evolutivo dos vírus arqueais como uma rede.[80] A falta de semelhanças entre marcadores filogenéticos nesta rede e na virosfera global, bem como ligações externas a elementos não virais, pode sugerir que algumas estirpes de vírus específicos de Archaea evoluíram de elementos genéticos móveis (MGE) não virais.[80]
Esses vírus foram estudados com mais detalhes entre os grupos termofílicos, particularmente as ordens Sulfolobales e Thermoproteales.[85] Dois grupos de vírus de DNA de cadeia simples que infectam Archaea foram recentemente isolados. Um grupo é exemplificado pelo Halorubrum pleomórfico vírus 1 (Pleolipoviridae) infectando arqueias halofílicas,[86] e o outro por Aeropyrum coil-shaped virus (Spiraviridae) que infecta um hospedeiro hipertermofílico (crescimento ideal a 90–95°C).[87] Notavelmente, o último destes vírus tem o maior genoma ssDNA atualmente conhecido. As defesas contra esses vírus podem envolver interferência de RNA a partir de sequências de DNA repetitivo relacionadas com os genes dos vírus.[88][89]
Reprodução[editar | editar código-fonte]
Os Archaea seguem um processo de reprodução assexuada por fissão binária, fragmentação ou brotamento. Ao contrário das bactérias, nenhuma espécie conhecida de Archaea forma endósporos.
Os membros conhecidos de Archaea reproduzem-se assexuadamente por fissão binária ou múltipla), fragmentação ou brotamento. A mitose e a meiose não ocorrem, e por isso, se uma espécie de Archaea existe em mais de uma forma, todas têm o mesmo material genético.[90]
A divisão celular é controlada num ciclo celular, no qual após o cromossoma da célula ter sido replicado, os dois cromossomas-filhos separam-se e a célula é dividida.[91] No género Sulfolobus, o ciclo apresenta características semelhantes aos sistemas bacterianos e eucarióticos. Os cromossomas são replicados a partir de múltiplos pontos de partida (origens de replicação) usando DNA polimerases que se assemelham às enzimas eucarióticas equivalentes.[92]
Em Euryarchaeota, a proteína de divisão celular FtsZ, que forma um anel de contração ao redor da célula, e os componentes do septo que é construído no centro da célula, são semelhantes aos seus equivalentes bacterianos.[91] Nos agrupamentos Crenarchaea[93][94] e Thaumarchaea,[95] a maquinaria de divisão celular Cdv desempenha um papel semelhante. Esta maquinaria está relacionada com a maquinaria ESCRT-III eucariótica que, embora mais conhecida pelo seu papel na classificação celular, também foi encontrada desempenhando um papel na separação entre células divididas, sugerindo um papel ancestral na divisão celular.[96]
Tanto as bactérias quanto os eucariontes, mas não Archaea, produzem esporos.[97] Algumas espécies de Haloarchaea sofrem mudança fenotípica e crescem como vários tipos diferentes de células, incluindo estruturas de paredes espessas que são resistentes ao choque osmótico e permitem que aquelas Archaea sobrevivam em água com baixas concentrações de sal, mas não são estruturas reprodutivas, antes adaptações destinadas a alcançar novos habitats.[98]
Ecologia[editar | editar código-fonte]

As primeiras arqueias observadas foram extremófilos vivendo em ambientes extremos como fontes termais e lagos salgados, em ambientes sem outros organismos presentes. A melhoria das ferramentas de deteção molecular levou à descoberta de Archaea em quase todos os habitats, incluindo o solo,[99] os oceanos e as áreas pantanosas. Os membros de Archaea são particularmente numerosos nos oceanos, e as arqueias presentes no plâncton podem ser um dos grupos de organismos mais abundantes no planeta.
Os Archaea são uma parte importante da vida na Terra, fazendo parte da microbiota de todos os organismos. No microbioma humano, estes organismos são importantes no intestino, na boca e na pele.[100]
A diversidade morfológica, metabólica e geográfica permite que as arqueias desempenhem múltiplos papéis ecológicos, entre os quais a fixação de carbono, o ciclo do azoto, a rotatividade de compostos orgânicos e a manutenção de comunidades microbianas simbióticas e sintróficas.[99][101]
Não se conhece qualquer exemplo claro de Archaea patogénicas ou parasitas, sendo, contudo, frequentemente mutualistas ou comensais, como é o caso das espécies matanogénicas (estirpes produtoras de metano) que habitam o trato gastrointestinal em humanos e ruminantes, onde o seu grande número facilita a digestão. As arqueias metanogénicas também são usados na produção de biogás e tratamento de esgotos. A biotecnologia explora enzimas de arqueias extremófilas que podem suportar altas temperaturas e a presença de solventes orgânicos tóxicos para a maioria dos microrganismos.
Comunicação[editar | editar código-fonte]
O fenómeno de detecção de quorum foi originalmente considerado como ausente em Archaea, mas estudos recentes mostraram evidências de que algumas espécies são capazes de realizar diafonia através da detecção de quorum. Outros estudos mostraram interações sintróficas entre Archaea e bactérias durante o crescimento de biofilmes.
Embora a pesquisa seja limitada na detecção de quorum entre os Archaea, alguns estudos descobriram proteínas LuxR em espécies arqueanas, exibindo semelhanças com as bactérias LuxR e, finalmente, permitindo a detecção de pequenas moléculas que são usadas em comunicação dda detecção de alta densidade populacional. Da mesma forma que as bactérias, os solos de Archaea LuxR demonstraram se ligar a AHLs (lactonas) e não-AHLs ligans, o que é uma grande parte na realização de comunicação intraespécies, interespécies e interreinos por meio da detecção de quorum.[102]
Habitats[editar | editar código-fonte]
Os Archaea ocorrem numa ampla gama de habitats, sendo reconhecidos como uma parte importante dos ecossistemas globais,[103] e pode representar cerca de 20% das células microbianas nos oceanos.[104] No entanto, os primeiros arqueanos descobertos eram extremófilos.[46] De facto, alguns Archaea sobrevivem a altas temperaturas, muitas vezes acima de 100 ºC, como encontrado em geysers, fumarolas negras (black smokers) e poços de petróleo. Outros habitats comuns incluem habitats muito frios e água altamente salinas, ácidas ou alcalinas, mas o grupo Archaea inclui mesófilos que crescem em condições amenas, em pântanos e alagadiços, esgotos, o oceano, o tracto intestinal dos animais e o solo.[99][103] Semelhante a rizobactérias promotoras do crescimento de plantas (PGPR), as Archaea são agora consideradas também como uma fonte de promoção do crescimento das plantas.[99]
Arqueias extremófilas são membros de quatro grupos fisiológicos principais. Estes são os halófilos, termófilos, alcalinófilos e acidófilos.[105] Esses grupos não são abrangentes ou específicos do filo, nem são mutuamente exclusivos, uma vez que alguns Archaea pertencem a vários grupos. No entanto, são um ponto de partida útil para a classificação.[106]
Os halófilos, incluindo o género Halobacterium, vivem em ambientes extremamente salinos, como os lagos hipersalinos, e superam em número os contrapartes bacterianos em salinidades superiores a 20–25%.[46]
Os termófilos crescem melhor em temperaturas acima de 45 ºC, em locais como as fontes termais. Os Archaea hipertermofílicos crescem melhor em temperaturas superiores a 80 ºC.[107] A espécie Methanopyrus kandleri estirpe 116 reproduz-se em águas a 122 ºC, a temperatura mais alta registada para qualquer organismo ativo.[108]
Outros Archaea existem em condições muito ácidas ou alcalinas.[105] Por exemplo, um dos acidófilos arqueanos mais extremos é Picrophilus torridus, que cresce a pH 0, o que equivale a prosperar em ácido sulfúrico a 1,2 molar.[109]
Essa resistência a ambientes extremos fez de Archaea o foco de especulação sobre as possíveis propriedades da vida extraterrestre.[110] Alguns habitats extremófilos não são muito diferentes daqueles que existem em Marte,[111] levando à hipótese de que micróbios viáveis poderiam ser transferidos entre planetas em meteoritos.[112]
Recentemente, vários estudos mostraram que Archaea existem não apenas em ambientes mesófilos e termofílicos, mas também estão presentes, às vezes em grande número, em baixas temperaturas. Por exemplo, Archaea são comuns em ambientes oceânicos frios, como os mares polares.[113] Ainda mais significativo é o grande número de arqueias encontradas nos oceanos do mundo em habitats não extremos, especialmente entre a comunidade plânctonica (como parte do picoplâncton).[114]
Embora os Archaea possam estar presentes em números extremamente altos nos oceanos (até 40% da biomassa microbiana), quase nenhuma das espécies conhecidas foi isolada e estudada em cultura pura.[115] Consequentemente, a compreensão do papel dos Archaea na ecologia oceânica é rudimentar, de modo que a sua influência total nos ciclos biogeoquímicos globais permanece amplamente inexplorada.[116]
Alguns Thermoproteota marinhos são capazes de nitrificação, sugerindo que esses organismos podem afetar o ciclo do azoto oceânico,[57] embora esses termoproteotas oceânicos também possam utilizar outras fontes de energia.[117]
Um grande número de Archaea também é encontrado nos sedimentos que cobrem o fundo do mar, com esses organismos constituindo a maioria das células vivas em profundidades superiores a 1 metro abaixo do fundo do oceano.[118][119]
Foi demonstrado que em todos os sedimentos da superfície oceânica (de 1 000 a 10 000 m de profundidade), o impacto da infecção viral é maior nos Archaea do que nas bactérias e a lise induzida por vírus das Archaea é responsável por até um terço da biomassa microbiana total morta, resultando na libertação de aproximadamente 0,3 a 0,5 gigatoneladas de carbono por ano globalmente.[120]
Papel nos ciclos biogeoquímicos[editar | editar código-fonte]
Os Archaea reciclam elementos como o carbono, o azoto e o enxofre através da participação no respetivo ciclo biogeoquímico nos vários habitats em que estes organismos estão presentes.[121] Os Archaea realizam muitas etapas no ciclo do azoto. Isso inclui tanto as reações que removem o azoto dos ecossistemas (como a respiração baseada em nitrato e a desnitrificação) quanto os processos que ali introduzem azoto (como a assimilação de nitrato e a fixação de azoto).[122][123]
Foi recentemente demonstrado o envolvimento dos Archaea em reações de oxidação de amónia. Essas reações são particularmente importantes nos oceanos.[58][124] As arqueias também parecem cruciais para a oxidação de amónia nos solos, produzindo o nitrito que outros micróbios então oxidam para nitrato. Plantas e outros organismos consomem o nitrato assim produzido.[125]
No ciclo do enxofre, os Archaea que crescem oxidando compostos de enxofre libertam esse elemento das rochas, tornando-o disponível para outros organismos, mas os Archaea que fazem isso, como Sulfolobus, produzem ácido sulfúrico como produto residual, e o crescimento desses organismos em minas abandonadas pode contribuir para a drenagem ácida de minas e outros danos ambientais.[126]
No ciclo do carbono, os Archaea metanogénicos removem o hidrogénio e desempenham um papel importante na decomposição da matéria orgânica pelas populações de microorganismos que atuam como decompositores em ecossistemas anaeróbios, como sedimentos, pântanos e em sistemas de tratamento de esgoto.[127]
Interação com outros organismos[editar | editar código-fonte]

As interações bem caracterizadas entre Archaea e outros organismos são o mutulalismo ou o comensalismo. Não há exemplos claros de Archaea patógenos ou parasitas conhecidos,[128][129] mas algumas espécies de organismos metanogénicos foram sugeridas como envolvidas em infecções na boca de humanos.[130][131]
A espécie Nanoarchaeum equitans pode ser um parasita de outra espécie de Archaea, já que só sobrevive e se reproduz dentro das células de Ignicoccus hospitalis (Crenarchaeota),[63] e parece não oferecer qualquer benefício ao seu hospedeiro.[132]
Mutualismo[editar | editar código-fonte]
Mutualismo é uma interação entre indivíduos de diferentes espécies que resulta em efeitos positivos (benéficos) na reprodução ou na sobrevivência das populações que interagem. Um exemplo bem compreendido de mutualismo é a interação entre protozoários e arqueias metanogénicas no tracto digestivo de animais que digerem celulose, como ruminante e térmitas.[133]
Nesses ambientes anaeróbicos, os protozoários quebram a celulose vegetal para obter energia. Este processo liberta hidrogénio como produto residual, mas altos níveis de hidrogénio reduzem a produção de energia. Quando arqueias metanogénicas convertem hidrogénio em metano, os protozoários beneficiam de mais energia.[134]
Em protozoários anaeróbicos, como Plagiopyla frontata, as arqueias residem no interior dos protozoários e consomem o hidrogénio produzido nos seus hidrogenossomas.[135][136]
Os Archaea também se associam a organismos maiores. Por exemplo, a arqueia marinha Cenarchaeum symbiosum vive dentro (é um endossimbionte) da esponja Axinella mexicana.[137]
Comensalismo[editar | editar código-fonte]
Os Archaea também podem ser comensais, beneficiando-se de uma associação sem ajudar ou prejudicar o outro organismo. Por exemplo, a arqueia metanogénica Methanobrevibacter smithii é de longe o arqueano mais comum na flora intestinal humana, constituindo cerca de um em cada dez de todos os procariotos no intestino humano.[138]
Em térmitas e em humanos, esses organismos metanogénicos podem de facto ser mutualistas, interagindo com outros micróbios no intestino para ajudar na digestão.[139] Comunidades arqueanas também se associam a uma variedade de outros organismos, como na superfície de corais,[140] e na região do solo que envolve as raízes das plantas (a rizosfera).[141][142]
Parasitismo[editar | editar código-fonte]
Embora os Archaea não tenham uma reputação histórica de serem patógenos, os Archaea são frequentemente encontrados com genomas semelhantes a patógenos mais comuns como Escherichia coli,[143] mostrando ligações metabólicas e história evolutiva com patógenos atuais. Os Archaea também têm detecção inconsistente em estudos clínicos devido à falta de categorização de Archaea em espécies mais específicas.[144]
Descoberta e classificação[editar | editar código-fonte]


O primeiro grupo de arqueias estudado foi o das espécies metanógenas. A metanogénese foi descoberta no lago Maggiore, na região alpina da Itália, em 1776, quando foi observado o borbulhar de ar combustível a partir de lamas situadas nas suas margens. Em 1882 observou-se que a produção de metano no intestino dos animais era devida à presença de microrganismos.[145]
Em 1936, ano que marcou o princípio da era moderna no estudo da metanogénese, o microbiologista norte-americano Horace Barker lançou as bases científicas para o estudo da fisiologia dos organismos produtores de metano e conseguiu desenvolver um meio de cultura apropriado para o crescimento dos organismos metanógenos. Nesse ano foram identificados os géneros Methanococcus e Methanosarcina.[146]
As primeiras arqueias extremófilas foram encontradas em ambientes quentes. Em 1970, Thomas D. Brock, da Universidade de Wisconsin, descobriu o género Thermoplasma, um grupo de arqueias termoacidófilas, e em 1972 o género Sulfolobus, um grupo de arqueias hipertermófilas.[147] Brock ter-se-á iniciado em 1969 no campo da biologia dos hipertermófilos com a descoberta da espécie Thermus aquaticus, que não é uma arqueia mas antes uma bactéria.
Em 1977 identificam-se as arqueias como o grupo procarionte filogeneticamente mais distante das bactérias ao ser demonstrado que os organismos metanógenos daquele agrupamento apresentam uma profunda divergência com todas as bactérias estudadas. Nesse mesmo ano propôs a categoria de super-reino para este grupo com o nome de Archaebacteria. Em 1978, o Bergey's Manual of Systematic Bacteriology (o manual de Bergey) dá-lhe a categoria de filo, com o nome de Mendosicutes, e em 1984 divide o reino Procaryotae, ou Monera, em 4 divisões, agrupando as Archaebacteria na divisão Mendosicutes.[148]
As arqueias hipertermófilas foram incluídas em 1984 num agrupamento que recebeu o nome de Eocyta, identificando-as como um grupo independente das então chamadas arqueobactérias (em relação aos organismos metanógenos) e às eubactérias, descobrindo-se para além disso que Eocyta era o grupo filogeneticamente mais próximo dos eucariontes.[149] A relação filogenética entre metanógenos e hipertermófilos faz com que em 1990 se renomeie o agrupamento Eocyta como Crenarchaeota e as metanógenas como Euryarchaeota, formando o novo grupo Archaea como um dos domínios do sistema dos três domínios.[150]
O novo domínio Archaea[editar | editar código-fonte]
Durante a maior parte do século XX, os procariotas foram vistos como um grupo singular de organismos e eram classificados com base na sua bioquímica, morfologia e metabolismo. Nesse período, os microbiólogos tentaram classificar os microrganismos com base nas estruturas das suas paredes celulares, nas suas formas e nas substâncias que consumiam.[151] No entanto, uma nova abordagem foi proposta em 1965 por Emile Zuckerkandl e Linus Pauling,[152] usando as sequências genéticas destes organismos para estabelecer quais procariotas eram genuinamente relacionados uns com os outros. Esta abordagem, conhecida como filogenética, é o principal método presentemente usado na classificação destes organismos.[153]
As Archaea foram primeiro classificados como um grupo separado de procariotas em 1977, por Carl Woese e George E. Fox, apoiados em árvores filogenéticas baseadas em sequências de genes do ARN ribossomal (rRNA),[154] sendo que, ao tempo, de entre os atuais Achaea, apenas eram conhecidos alguns microrganismos metanogénicos.[155]
Os dois grupos assim encontrados foram designados por Archaebacteria e Eubacteria e tratados como reinos ou sub-reinos, que Woese e Fox denominaram Urkingdoms (ou Arquirreinos), argumentando que estes grupos de procariotas eram formas de vida fundamentalmente diferentes. Para enfatizar esta diferença, estes dois domínios foram mais tarde renomeados de Archaea e Bacteria.[156] Para tal, Woese e Fox deram a como primeira evidência para suportar a existência do agrupamento Archaebacteria como uma "linha de descendência" separada os seguintes argumentos: (1) a falta de peptidoglicano nas suas paredes celulares; (2) a presença de duas coenzimas incomuns; e (3) resultados do sequenciamento do gene ARN ribossomal 16S apontando para uma origem genética distinta. Para enfatizar essa diferença, Woese, Otto Kandler e Mark Wheelis propuseram posteriormente a reclassificação dos organismos em três domínios naturais, numa estrutura que ficou conhecida como o «sistema dos três domínios»: os domínios (1) Eukarya; (2) Bacteria; e (3) Archaea.[157] Ao propor a existência de três domínios assentes em linhagens há muito separadas, Carl Woese deu origem ao processo que ficou para a história da biologia como a revolução Woeseiana.[158]
O termo archaea tem origem no grego clássico ἀρχαῖα, palavra que significava 'coisas antigas' ou 'antiguidades',[159] pois como os primeiros representantes do domínio Archaea fossem organismos metanogénicos assumiu-se que o seu metabolismo refletia a atmosfera primitiva da Terra (mais concretamente a atmosfera pré-biótica) e seria reflexo da antiguidade destes organismos. Contudo, à medida que novos habitats foram estudados, mais organismos foram descobertos, questionando esse entendimento. No grupo foram também sucessivamente incluídos microrganismos halofílicos extremos[160] e micróbios hipertermofílicos.[161]
Em conclusão, inicialmente, e durante um período alargado, apenas os microrganismos metanogénicos foram colocados neste novo domínio, e as arqueias eram vistos como extremófilos que existiam apenas em habitats como fontes termais com temperaturas elevadas, lagos salgados e lagos hipersalinos. Nos finais do século XX, os microbiólogos perceberam que as arqueias, grupo então já consolidado como o domínio Archaea, eram um grande e diverso grupo de organismos que tinham uma vasta distribuição na natureza e eram comuns em habitats não tão extremos, estando presentes nos solos e oceanos.[103] Já se conhece também que as arqueias estão presentes no microbioma humano, apesar de não se conhecer nenhuma espécie de arqueia patogénica.[162]
Esta nova apreciação da importância e ubiquidade dos membros de Archaea resultou da expansão do uso das técnicas de reação em cadeia da polimerase para detetar procariotas em amostras de água ou solo, a partir apenas dos seus ácidos nucleicos. Isto permite a deteção e identificação de organismos que não podem ser cultivados no laboratório, processo muitas vezes difícil dados os requisitos específicos e interdependências que não são conhecidos.[163][164]
Comparação com os outros domínios[editar | editar código-fonte]
Para além das claras diferenças metabólicas e morfológicas em relação às bactérias, os membros do agrupamento Archaea possuem características ultraestruturais próprias que também suportam o seu distanciamento filogenético em relação aos eucariotas. A mais marcante dessas características distintivas é a ausência de um núcleo delimitado por uma membrana, motivo pelo qual tanto as arqueias como as bactérias são denominadas de procariotas. Uma vez que esta definição de procariotas é baseada em uma ausência, ou seja, é feita com base numa característica que está presente nos eucariotas mas não está presente nos procariotas, alguns autores sugerem que a transcrição acoplada à tradução seja utilizada como característica apomórfica dos procariotas.[165]
Esta dupla distinção em relação às bactérias e aos eucariotas justificou a criação de um novo domínio para o agrupamento Archaea, levando ao aparecimento do sistema dos três domínios. A separação entre os domínios Bacteria e Archaea foi proposta na década de 1970, quando o microbiólogo Carl Woese verificou que ao comparar as sequências de RNA ribossómico de várias espécies era possível separá-las em três grupos distintos. Apesar do nome (Archaea em grego significa “antigo”), tal não significa que os membros do grupo Archaea sejam mais semelhantes aos organismos primitivos do que as bactérias ou os eucariotas. Carl Woese decidiu atribuir o nome Archaea a este domínio para fazer sobressair a sua natureza mais primitiva relativamente aos eucariotas,[82] e não como a atribuição de características basais ao agrupamento.
Outra característica distintiva resulta da capacidade dos membros de Archaea usarem fontes de energia mais diversificadas do que eucariotas, variando de compostos orgânicos como os açúcares, até à amónia, a iões metálicos ou mesmo hidrogénio gasoso. O agrupamento Haloarchaea (ex-Halobacteria), que reune organismos tolerantes ao sal, usam a luz solar como fonte de energia através de uma estrutura celular chamada bacteriorrodopsina,[166] o que constitui um fenómeno de fototrofia (mas não de fotossíntese). Também existem espécies de Archaea que são autotróficas, ou seja, que são capazes de fixar carbono atmosférico. Contudo, apesar de autotróficos, ao contrário das plantas e cianobactérias, nenhuma espécie conhecida de Archaea é capaz de usar energia solar para fixar carbono. Outro grupo de arqueias, pertencentes ao filo Euryarchaeota, conseguem produzir metano, sendo por isso chamadas de metanogénicas, na sua maior parte organismos que vivem no intestino de ruminantes.
Além disso, as arqueias possuem uma membrana celular com lípidos compostos de uma associação de glicerol-éter, enquanto que os das bactérias e eucariotas são compostos de glicerol-éster. Além disso, o grupo glicerol ao qual a cadeia hidrofóbica se encontra ligada tem estereoquímica diferente nas arqueias, comparativamente às bactérias e aos eucariotas. Também ao contrário das bactérias, as arqueias não possuem uma parede celular de peptidoglicanos. Apenas um grupo relativamente pequeno de arqueias possui uma parede celular composta por um polissacarídeo (pseudomureína), mas a maior parte das arqueias possui antes uma estrutura proteica para-cristalina chamada de S-layer (superfície S ou camada S).[167] Finalmente, o flagelo das arqueias é diferente em composição e desenvolvimento do das bactérias, tendo sido inclusivamente chamado de arcaelo (do neolatim archaellum) para evidenciar as diferenças relativamente ao flagelo bacteriano.[168]
A tabela a seguir compara algumas das principais características dos três domínios, para ilustrar as semelhanças e diferenças que as arqueias compartilham com os outros domínios, incluindo as que lhes são exclusivas:[169]
| Propriedade | Archaea | Bacteria | Eukaryota |
|---|---|---|---|
| Membrana celular | Lípidos ligados a éteres | Lípidos ligados a ésteres | Lípidos ligados a ésteres |
| Parede celular | Pseudopeptidoglicano, glicoproteína, ou S-layer | Peptidoglicano, S-layer, ou ausência de parede celular | Várias estruturas |
| Estrutura do genoma | Cromossomas circulares, tradução e transcrição semelhantes aos Eukaryota | Cromossomas circulares, tradução e transcrição únicas | Cromossomos múltiplos e lineares, mas tradução e transcrição semelhantes a Archaea |
| Estrutura interna da célula | Sem organelos rodeados por membrana (?[170]) e sem núcleo celular | Sem organelos rodeados por membrana e sem núcleo celular | Organelos rodeados por membrana e núcleo celular presente |
| Metabolismo[171] | Vários, incluindo diazotrofia, com metanogénese exclusiva de Archaea | Vários, incluindo fotossíntese, respiração aeróbica e respiração anaeróbica, fermentação, diazotrofia e autotrofia | Fotossíntese, respiração celular e fermentação; sem diazotrofia |
| Reprodução | Reprodução assexuada, transferência horizontal de genes | Reprodução assexuada, transferência horizontal de genes | Reprodução sexual e reprodução assexuada |
| Iniciação da síntese proteica | Metionina | Formilmetionina | Metionina |
| Polimerase do RNA | Uma | Uma | Múltiplas |
| EF-2/EF-G | Sensível à toxina da difteria | Resistente à toxina da difteria | Sensível à toxina da difteria |
As principais diferenças e semelhanças podem-se resumir na seguinte tabela:[169]
| Compartilhadas com Bacteria | Compartilhadas com Eukarya | Exclusivas de Archaea |
|---|---|---|
| Sem núcleo nem organelos membranosos | Sem peptidoglicano | Estrutura da parede celular (por exemplo, algumas arqueias têm paredes com pseudomureína) |
| Genoma circular | ADN associado com histonas[172][173] | Membrana celular que contêm lípidos com ligações éter |
| Genes agrupados em operões | Tradução de proteínas que se inicia com a metionina | Sistema de locomoção próprio (archaellum) |
| Sem intrões nem processamento do ARN | ARN polimerase, promotores, e outra maquinaria transcricional similar[174][175] | Estrutura dos ribossomas (característica compartilhada tanto com Bacteria como com Eukarya) |
| ARNm policistrónico | Replicação do ADN e reparação semelhantes[176] | Sequência do ARNt e metabolismo[177] |
| Tamanho celular (>100 vezes menor do que os eucariotas) | ATPase similar (ATPase V ou Tipo V) | Sem a enzima ácido gordo sintase |
Archaea foi separado como um terceiro domínio por causa das grandes diferenças na estrutura de RNA ribossómico. A molécula 16S rRNA destaca-se como a chave para a produção de proteínas em todos os organismos. Como essa função é tão central para a vida, é improvável que organismos com mutações no seu 16S rRNA sobrevivam, levando a uma grande (mas não absoluta) estabilidade na estrutura desse polinucleotídeo ao longo de gerações. A molécula 16S rRNA é suficientemente grande para mostrar variações específicas do organismo, mas ainda pequena o suficiente para ser comparada rapidamente. Em 1977, Carl Woese, um microbiologista que estudava as sequências genéticas de microrganismos, desenvolveu um novo método de comparação que envolvia dividir o RNA em fragmentos que poderiam ser classificados e comparados com outros fragmentos de outros organismos.[155] Quanto mais semelhantes os padrões deste gene entre as espécies, mais intimamente elas estão filogeneticamente relacionadas.[178]
Carl Woese usou seu novo método de comparação de rRNA para categorizar e contrastar diferentes organismos. Para isso, comparou uma variedade de espécies e identificou um grupo de organismos metanogénicos com rRNA muito diferente de qualquer procariota ou eucariota conhecido.[155] Esses organismos metanogénicos eram muito mais semelhantes entre si do que com outros organismos, levando Woese a propor o estabelecimento do novo domínio de Archaea.[155] Os resultados que obteve mostraram que os Archaea eram geneticamente mais semelhantes aos eucariontes do que aos procariontes, embora fossem mais semelhantes aos procariontes em estrutura.[179] Isso levou à conclusão de que Archaea e Eukarya compartilharam um ancestral comum mais recente que Eukarya e Bacteria[179] e que o desenvolvimento do núcleo celular ocorreu após a divisão entre Bacteria e esse ancestral comum.[179][157]
Uma propriedade exclusiva de Archaea é o uso abundante de lípidos ligados a éteres nas suas membranas celulares. As ligações éter são quimicamente mais estáveis do que as ligações éster encontradas em bactérias e eucariotas, o que pode ser um fator que contribui para a capacidade de muitas Archaea sobreviverem em ambientes extremos que impõem forte ataque às membranas celulares, como o calor extremo e a hipersalinidade. A análise comparativa de genomas de Archaea também identificou vários indels de assinatura conservados moleculares e a ocorrência de proteínas de assinatura exclusivamente presentes em todas as Archaeas ou em diferentes grupos principais dentro de Archaea.[180][181][182]
Outra característica única de Archaea, não encontrada em nenhum outro grupo de organismos, é metanogénese (a produção metabólica de metano). As Archaea metanogénicas desempenham um papel fundamental em ecossistemas com organismos que obtêm energia da oxidação do metano, muitos dos quais são bactérias, pois costumam ser a principal fonte de metano nesses ambientes e podem desempenhar um importante papel como produtores primários. Os organismos metanogénicos também desempenham um papel crítico no ciclo do carbono, quebrando o carbono orgânico em metano, que também é um dos principais gases de efeito estufa.[183]
Essa diferença na estrutura bioquímica de Bacteria e Archaea foi explicada por meio de processos evolutivos. É teorizado que ambos os domínios tiveram origem em fontes hidrotermais alcalinas em mares profundos. Pelo menos duas vezes, os micróbios desenvolveram a biossíntese lipídica e a bioquímica da parede celular. Essas origens paralelas fundaram as linhagens separadas Archaea e Bacteria. Tem sido sugerido que o último ancestral comum universal não era um organismo de vida livre.[184] No entanto, esta visão foi contestada por outros investigadores e está atualmente em disputa.[185]
Questionamento do sistema de três domínios[editar | editar código-fonte]
Em hipóteses anteriores como a de Carl Woese, foi argumentado que as bactérias, arqueias e eucariotas representavam três linhagens evolutivas distintas que divergiram há muitos milhões de anos dum grupo ancestral de organismos.[186][187] Segundo Woese, como as arqueias e as bactérias não estariam mais estritamente relacionadas umas com as outras do que com os eucariontes, propôs-se que o termo "procariota" não teria um verdadeiro sentido evolutivo e que por isso teria que ser desconsiderado por completo.[26]
Contudo, outros investigadores argumentam que as arqueias e eucariotas surgiram dum grupo de bactérias.[188] Tendo essa possibilidade em vista, muitos biólogos evolutivos consideram que no sistema de três domínios se exagerou na diferença entre arqueias e bactérias, e sustentam que a transição mais dramática produziu-se entre Prokaryota e Eukaryota (sistema dos dois impérios), este último de origem mais recente por eucariogénese e como resultado da fusão endosimbiótica de pelo menos dois procariontes: uma arqueia e uma bactéria.
Assim, em alternativa à tese de Carl Woese, Cavalier-Smith propôs o clado Neomura para representar esta teoria; Neomura significa 'paredes novas' e faz referência à teoria de que as arqueias e os eucariotas teriam derivado de bactérias que (entre outras adaptações) substituíram as paredes de peptidoglicano por outras de glicoproteínas.
O conceito de espécie em Archaea[editar | editar código-fonte]
A classificação das arqueias em espécies é também controversa, já que em biologia, e seguindo a definição de Ernst Mayr, uma espécie é definido como um grupo de organismos geneticamente relacionados de tal forma que se podem reproduzir entre si e não com outros, ou seja que estão em isolamento reprodutivo. Ora a condição atrás referida pode ser aplicada com relativa facilidade a animais ou plantas, mas dificilmente se aplica às arqueias, que se reproduzem assexuadamente.[189]
Além disso, os membros de Archaea apresentam um alto grau de transferência horizontal de genes entre distintas linhagens. Alguns investigadores sugerem que os indivíduos podem agrupar-se em populações similares a espécies quando se observa uma grande similaridade dos seus genomas e uma pouco frequente transferência de genes entre essas populações e populações de células que tenham genomas menos relacionados, como no caso do género Ferroplasma.[190]
Por outro lado, em estudos realizados sobre o género Halorubrum encontrou-se uma significativa transferência de genes com populações menos relacionadas, o que limita a aplicabilidade deste critério.[191] Em consequência, questiona-se se para os membros de Archaea a designação de espécies apresenta significado prático.[192]
Os conhecimentos presentemente disponíveis sobre a diversidade genética deste grupo são fragmentários e o número total de espécies de arqueias não pode ser estimado com precisão.[193] A estimativa dos possíveis filos que possam existir, quase todos sem terem sido completamente descritos, é de entre 18 e 23, dos quais só 8 integram representantes que foram cultivados e estudados diretamente (na LPSN, a List of Prokaryotic names with Standing in Nomenclature, em 2023 figuram apenas 2 filos, e ainda assim sem aprovação formal).[194] Muitos destes grupos hipotéticos só são conhecidos por uma só sequência de ARNr, o que indica que a diversidade entre estes organismos continua pouco clara.[195] Entre as bactérias também existem muitos micróbios não cultivados com implicações parecidas para a sua caracterização.[196]
Classificação[editar | editar código-fonte]

A classificação das Archaea e dos procariotas em geral é um campo contencioso e em rápida evolução. Os atuais sistemas de classificação, seguindo uma lógica assente em critérios filogenéticos, pretendem organizar as arqueias em grupos de organismos que partilham caracteres estruturais associados a ancestrais comuns.[197]
Estas classificações apoiam-se grandemente nas sequências de genes de ARN ribossomal para revelar as relações entre organismos (filogenética molelular).[198] A maioria das espécies de Archaea cultiváveis e bem investigadas são membros de dois filos principais, os Euryarchaeota e os [Thermoproteota]] (anteriormente designados por Crenarchaeota). Outros grupos foram tentativamente criados, como, por exemplo, para a espécie peculiar Nanoarchaeum equitans, que foi descoberta em 2003, e para a qual foi proposto o seu próprio filo, o filo Nanoarchaeota.[199]
Um novo filo, o Korarchaeota, foi também proposto, contendo um pequeno grupo de espécies termofílicas pouco usuais, que partilham caracteres de ambos os filos principais, mas que aparenta ser filogeneticamente mais próxima dos Crenarchaeota.[200][201] Outras espécies detetadas recentemente aparentam ser apenas filogeneticamente relacionadas de maneira distante com algum destes grupos, tais como os Archaeal Richmond Mine Acidophilic Nanoorganisms (ARMAN, incluindo Micrarchaeota e Parvarchaeota), descobertos em 2006,[202] grupo que inclui alguns dos menores organismos conhecidos.[203]
Um superfilo, provisoriamente designado por TACK, que inclui Thaumarchaeota (agora Nitrososphaerota), "Aigarchaeota", Crenarchaeota (agora Thermoproteota) e "Korarchaeota" foi proposto em 2011 para permitir ligar as Archaea com a origem dos eucariontes.[204] Em 2017, o recém-descoberto e recém-nomeado superfilo Asgard foi proposto para agrupar os organismos filogeneticamente mais diretamente relacionados com o ancestral comum dos eucarióticos, formando um potencial grupo irmão do clado TACK.[205]
Em 2013, foi proposto o superfilo DPANN para agrupar "Nanoarchaeota", "Nanohaloarchaeota", Archaeal Richmond Mine acidophilic nanoorganisms (ARMAN, compreendendo "Micrarchaeota" e "Parvarchaeota"), e outras arqueias semelhantes. Este superfilo arqueano abrange pelo menos 10 linhagens diferentes e inclui organismos com células e tamanhos de genoma extremamente pequenos e capacidades metabólicas limitadas. Em consequência, muitos membros do DPANN podem ser obrigatoriamente dependentes de interações simbióticas com outros organismos e podem até incluir novas formas de parasitismo. Porém, em outras análises filogenéticas foi constatado que DPANN não forma um grupo monofilético e que é causado pela atração de ramo longo (LBA), sugerindo que todas essas linhagens pertencem a "Euryarchaeota".[206][2]
Em 2015 foi sugerida a existência de um novo filo, o Lokiarchaeota.[207]
A classificação dos membros de Archaea em espécies é controversa. Em biologia, uma espécie é um grupo de organismos relacionados. Uma definição popular de espécie em animais é que são um grupo de organismos que se podem cruzar uns com os outros e que estão reprodutivamente isolados de outros grupos de organismos (isto é, não podem se cruzar com outras espécies).[208] No entanto, esforços para classificar procariotas como as Archaea em espécies são complicados por serem assexuais e mostrarem níveis altos de transferência horizontal de genes entre linhagens. Esta área é ainda contenciosa; com, por exemplo, alguns dados sugerindo que nos Archaea como o género Ferroplasma, células individuais podem ser agrupadas em populações que possuem genomas altamente similares e que raramente transferem genes com grupos de células mais divergentes.[209] Pensa-se que estes grupos de células sejam análogos a espécies. Por outro lado, estudos em Halorubrum encontraram trocas genéticas significantes entre tais populações.[210] Tais resultados levaram ao argumento de que classificar estes grupos de organismos em espécies terá pouco significado prático.[211]
O conhecimento atual sobre a diversidade dos Archaea é fragmentário e o número total de espécies de Archaea não pode ser estimado com precisão.[198] Mesmo estimativas do número total de filos em Archaea variam entre 18 a 23, dos quais apenas 8 filos possuem representantes que foram cultivados e estudados diretamente. Muitos destes grupos hipotéticos são conhecidos somente através de uma simples sequência de ARNr, indicando que a vasta maioria da diversidade entre estes organismos permanece completamente desconhecida.[212] O problema de como estudar e classificar micróbios não cultivados, ocorre também em Bacteria.[213]
Os seguintes filos foram validamente publicados de acordo com o Código Bacteriológico:[214]
Os seguintes filos foram propostos, mas não foram validamente publicados de acordo com o Código Bacteriológico (incluindo aqueles que têm estatuto de candidatus):
- "Candidatus Aenigmarchaeota"
- "Candidatus Aigarchaeota"
- "Candidatus Altiarchaeota"
- "Candidatus Asgardaeota"
- "Candidatus Bathyarchaeota"
- "Candidatus Brockarchaeota"
- "Candidatus Diapherotrites"
- "Euryarchaeota"
- "Candidatus Geoarchaeota"
- "Candidatus Hadarchaeota"
- "Candidatus Hadesarchaeota"
- "Candidatus Halobacterota"
- "Candidatus Heimdallarchaeota"
- "Candidatus Helarchaeota"
- "Candidatus Huberarchaeota"
- "Candidatus Hydrothermarchaeota"
- "Candidatus Korarchaeota"
- "Candidatus Lokiarchaeia"
- "Candidatus Lokiarchaeota"
- "Candidatus Mamarchaeota"
- "Candidatus Marsarchaeota"
- "Candidatus Methanobacteriota"
- "Candidatus Micrarchaeota"
- "Candidatus Nanoarchaeota"
- "Candidatus Nanohaloarchaeota"
- "Candidatus Nezhaarchaeota"
- "Candidatus Odinarchaeota"
- "Candidatus Pacearchaeota"
- "Candidatus Parvarchaeota"
- "Candidatus Thermoplasmatota"
- "Candidatus Thorarchaeota"
- "Candidatus Undinarchaeota"
- "Candidatus Verstraetearchaeota"
- "Candidatus Woesearchaeota"
Cladograma[editar | editar código-fonte]
Embora a árvore filogenética de Archaea continue sem obter amplo consenso, os seguintes cladogramas apresentam as versões mais recentes:[215][216][217][218][219]
| Tom A. Williams et al. 2017[215] e Castelle & Banfield 2018[216] | GTDB versão 08-RS214 (28 de abril de 2023)[217][218][219] | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
|
Origem e evolução[editar | editar código-fonte]
A idade da Terra é aproximadamente 4,54 mil milhões de anos.[221][222][223] Evidência científica sugere que a abiogénese ocorreu na Terra pelo menos há 3,5 mil milhões de anos.[224][225] A evidência mais antiga que comprova a existência de vida na Terra é um depósito de grafite, encontrada como substância biogénica em rochas metassedimentares formadas há 3,7 mil milhões de anos, descoberto na Gronelândia Ocidental,[226] e uma esteira microbiana fóssil encontrada em arenitos com 3,48 mil milhões de anos descoberta na Austrália Ocidental.[227][228] Em 2015, possíveis restos de matéria biótica foram encontrados em rochas de 4,1 mil milhões de anos na Austrália Ocidental.[229][230]
Origem de Archaea[editar | editar código-fonte]
Os Archaea são formas de vida antigas. Prováveis fósseis destas células foram datadas de perto de 3,42 mil milhões de anos,[231] e vestígios de lípidos que poderiam ser de Archaea ou de eucarióticos foram detectados em xistos que datam de há 2,7 bilhões de anos.[232]
Embora se conheçam prováveis células procarióticas fósseis que datam de quase 3,5 mil milhões de anos atrás, a maioria dos procariotas não tem morfologias distintas, e por isso as formas fósseis não podem ser usadas para identificá-los como Archaea.[233] Em vez disso, fósseis químicos de lípidos específicos são mais informativos porque tais compostos não ocorrem em outros organismos.[234]
Carl Woese argumentou que as bactérias, as arqueias e os eucariotas, cada qual representa uma linha de descendência que divergiu de uma colónia ancestral de organimos.[235][236] Uma possibilidade é que essa divergência tenha ocorrido antes da evolução das células,[236][237] quando a falta de uma membrana celular permitia a transferência lateral de genes irrestrita, e que os ancestrais comuns dos três domínios surgiram pela fixação de subconjuntos específicos de genes.[236][237] Alguns biólogos, no entanto, argumentaram que as arqueias e os eucariotas divergiram a partir de um grupo de bactérias.[238]
É possível que o último ancestral comum das bactérias e dos Archaea fosse um microrganismo termófilo, o que levanta a possibilidade de que temperaturas menores são "ambientes extremos" para as arqueias, e que organismos de vivem em ambientes mais frios apareceram mais tarde na história da vida na Terra.[239]
Visto que os agrupamentos taxonómicos Archaea e Bacteria não são filogeneticamente mais próximos entre si do que são em relação aos eucariotas, isto levou a que o termo procariota não tivesse significado evolutivo e devesse ser descartada inteiramente.[26] No entanto, semelhanças estruturais e funcionais entre linhagens geralmente ocorrem por causa de traços ancestrais compartilhados ou convergência evolutiva. Essas semelhanças são conhecidas como grados, e os procariontes são melhor pensados como um grado evolutivo da vida, distinto por características como a ausência de organelos rodeados por membrana.
A relação entre Archaea e os eucariotas permanece um problema importante. Para além das semelhanças na estrutura celular e função, que são discutidas abaixo, muitas árvores genéticas juntam os dois grupos. Algumas análises anteriores sugeriam que a relação entre eucariotas e o filo Euryarchaeota são mais próximas que as relações entre os Euryarchaeota e o filo Crenarchaeota.[240] No entanto, é hoje em dia considerado mais provável que o ancestral dos eucariotas divergiu cedo dos archaea.[241][242] A descoberta de genes parecidos com os de archaea, em certas bactérias como Thermotoga marítima, torna estas relações difíceis de determinar, uma vez que a transferência horizontal de genes ocorreu.[243] Alguns cientistas sugeriram que os eucariotas apareceram através de uma fusão de Archaea e Eubacteria, que se tornaram no núcleo e no citoplasma; isto conta para várias semelhanças genéticas mas torna-se difícil a explicar a estrutura celular.[244]
Visto que a maioria dos procariontes não possuem morfologias distintivas, as formas dos fósseis não podem ser utilizadas para os identificar como Archaea. Por sua vez, fósseis químicos, na forma de lípidos únicos encontrados em Archaea, são mais informativos porque tais compostos não ocorrem em outros grupos de organismos.[245] Os traços mais antigos destes lípidos (isoprenos) que se conhecem foram encontrados no cinturão de rochas verdes de Isua, na Gronelândia Ocidental, que inclui sedimentos formados há 3,8 mil milhões de anos e que são os mais antigos que se conhecem.[246] A origem das Archaea parece ser muito antiga e as linhagens de Archaea podem ser as mais antigas que existem na Terra.[247]
Algumas publicações sugerem que restos lipídicos arqueais ou eucarióticos estão presentes em xistos que datam de 2,7 mil milhões de anos atrás,[248] embora tais dados tenham sido questionados posteriormente.[249]
Relação com as bactérias[editar | editar código-fonte]
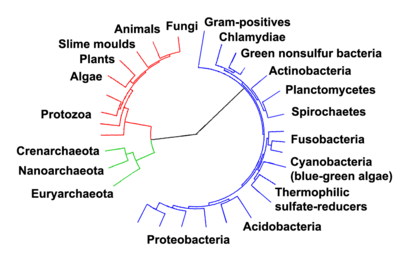
As relações entre os três domínios da vida celular são de importância central para a compreensão da origem da vida. A maioria das vias metabólicas, que são o objeto da maioria dos genes de um organismo, são comuns entre Archaea e Bacteria, enquanto a maioria dos genes envolvidos na expressão do genoma são comuns entre Archaea e Eukarya.[251]
Entre os procariotas, a estrutura celular das arqueias é mais semelhante à das bactérias gram-positivas, principalmente porque ambas têm uma única bicamada lipídica[252] e geralmente contêm um sáculo (sacculus) espesso, quase um exoesqueleto, de composição química variável.[253]
Em algumas árvores filogenéticas baseadas em diferentes sequências de genes e de proteínas de homólogos procarióticos, os genes e proteínas arqueais estão filogenomicamente mais intimamente relacionados com os homólogos de bactérias gram-positivas.[252] Archaea e bactérias gram-positivas também compartilham indels conservados em várias proteínas importantes, como Hsp70 e glutamina sintetase I,[252][254] mas a filogenia desses genes foi interpretada para revelar a transferência de genes entre domínios,[255][256] e pode não refletir a relação entre linhagens.[257]
Foi proposto que os Archaea evoluíram a partir de bactérias gram-positivas em resposta a pressão de seleção causada por antibióticos naturais.[252][254][258] Essa teoria é sugerido pela observação de que os Archaea são resistentes a uma ampla variedade de antibióticos produzidos principalmente por bactérias gram-positivas,[252][254] e que esses antibióticos agem principalmente nos genes que distinguem Archaea das bactérias. A proposta assenta na constatação que a pressão seletiva para a resistência gerada pelos antibióticos gerados pelas bactérias gram-positivas foi suficiente para causar mudanças extensas em muitos dos genes-alvo dos antibióticos, e que essas estirpes representavam os ancestrais comuns da atual Archaea.[258]
A evolução de Archaea em resposta à seleção imposta pelos antibióticos, ou qualquer outra pressão seletiva competitiva, também poderia explicar a sua adaptação a ambientes extremos (como alta temperatura ou acidez) como resultado de uma busca por nichos desocupados para escapar aos organismos produtores de antibióticos.[258][259]
Cavalier-Smith propôs teorias explicativas das origem das Archaea similares.[260] Esta proposta também é apoiada por outro trabalho que investiga relações estruturais de proteínas[261] e estudos que sugerem que bactérias gram-positivas podem constituir as primeiras linhagens ramificadas dentro dos procariotas.[262]
Relação com os eucariotas[editar | editar código-fonte]

As relações evolutivas entre arqueias e eucariotas não são claras. Para além das semelhanças na estrutura e funções celulares, que serão discutidas mais abaixo, muitas árvores genéticas agrupam as duas linhagens.[264]
Entre os fatores que tornam complexa a relação evolutiva entre estes agrupamentos incluem-se alegações de que a proximidade filogenética entre os eucariotos e o filo arqueano Thermoproteota (ex-Crenarchaeota) é maior do que a existente entre os "Euryarchaeota" e o filo Thermoproteota,[265] e a presença de genes do tipo arqueano em certas bactérias, como Thermotoga maritima, devido à transferência horizontal de genes.[266]
A hipótese padrão postula que o antepassado dos eucariotas divergiu muito cedo das Archaea,[267][268] e que os eucariotas surgiram por simbiogénese em resultado da fusão de uma arqueia com uma eubactéria, que deram lugar ao aparecimento do núcleo celular e do citoplasma, respetivamente, o que explicaria várias parecenças genéticas entre os grupos.[263] Contudo, por outro lado, esta hipótese debate-se com múltiplas dificuldades para explicar a estrutura celular.[269]
Uma hipótese alternativa, a hipótese do eócito, postula que o domínio Eukaryota emergiu relativamente tarde do domínios Archaea,[270] o que explicaria as semelhanças entre os grupos. Nesse caso, ficaria estabelecido que Eukaryota se relaciona com Archaea pela proximidade de ambos com o superfilo TACK (siglas de quatro filos arqueanos).[271] Nesse contexto, a descoberta do clado TACK é fundamental para a compreensão da origem das arqueias e da primeira célula eucariota. Nas diferentes árvores filogenéticas elaboradas, Archaea estaria de diversas formas relacionada com Eukaryota. Cruzando estes dados obtém-se um resultado que engrauece a hipótese dos três domínios, apoiando a hipótese do eócito e a simbiogénese pré-eucariota e depreender-se-ia a presença do superfilo que agrupa os vários filos arqueanos com Eukaryota sob as siglas TACK,[272] o que, segundo as árvores filogenéticas dos partidários desta hipótese, pode resumir-se da seguinte forma:
| Prokaryota |
| |||||||||||||||||||||||||||
Esta última hipótese foi fortalecida pela descoberta em 2015 de uma nova linhagem de arqueias, o género Lokiarchaeum (tipo do novo filo proposto "Lokiarchaeota"), nomeado a partir da designação de uma fonte hidrotermal chamada Castelo de Loki, situada no Oceano Ártico, que foi considerado o organismo arqueano filogeneticamente mais próximo dos eucariotas conhecido ao tempo. Tem sido considerado um organismo de transição entre procariontes e eucariontes.[273][274]
Vários filos irmãos de "Lokiarchaeota" já foram encontrados ("Thorarchaeota", "Odinarchaeota", "Heimdallarchaeota"), todos juntos compreendendo um supergrupo recentemente proposto Asgard, que pode ser considerado como um táxon irmão de Proteoarchaeota.[205][3][275]
Detalhes da relação dos membros do agrupamento Asgard e eucariotas ainda estão sob consideração,[276] embora, em janeiro de 2020, cientistas tenham relatado que Candidatus Prometheoarchaeum syntrophicum, um tipo de arqueia Asgard, pode ser uma possível ligação entre microrganismos procarióticos simples e os organismos eucarióticos complexos que existiram cerca de dois mil milhões anos atrás.[277][278]
Usos em biotecnologia[editar | editar código-fonte]
os Archaea extremófilos, particularmente os resistentes ao calor ou a extremos de acidez e alcalinidade, são uma fonte de enzimas que funcionam sob essas condições adversas.[279][280] Essas enzimas encontraram muitos usos. Por exemplo, as DNA polimerases termoestáveis, como a Pfu DNA polimerase de Pyrococcus furiosus, revolucionou a biologia molecular ao permitir a reação em cadeia da polimerase para ser usado em pesquisa como uma técnica simples e rápida para clonagem de DNA. Na indústria, as amilases, galactosidases e pululanases encontradas em outras espécies de Pyrococcus que funcionam em temperaturas superiores a 100 ºC permitem o processamento de alimentos em altas temperaturas, como a produção de leite com baixo teor de lactose e soro.[281]
As enzimas desses Archaea termofílicos também tendem a ser muito estáveis em solventes orgânicos, permitindo o uso em processos ecologicamente sustentáveis de química verde para síntese de compostos orgânicos.[280] Essa estabilidade torna esses enzimas mais fáceis de usar em biologia estrutural. Consequentemente, as contrapartes de enzimas bacterianas ou eucarióticas de arqueias extremófilas são frequentemente usadas em estudos estruturais.[282]
Em contraste com a gama de aplicações das enzimas arqueanas, o uso dos próprios organismos em biotecnologia é menos desenvolvido. Arqueias metanogénicas são uma parte vital do tratamento de esgotos, pois fazem parte da comunidade de microrganismos que realizam a digestão anaeróbia e produzem biogás.[283] No processamento de minerais, os Archaea acidófilos são promissores para a extração de metais de minério, incluindo ouro, cobalto e cobre.[284]
Os Archaea produzem uma nova classe de antibióticos potencialmente úteis. Algumas dessas arqueocinas foram caracterizadas, mas acredita-se que existam centenas mais, especialmente nos géneros Haloarchaea e Sulfolobus. Esses compostos diferem em estrutura dos antibióticos bacterianos, portanto, podem ter novos modos de ação. Além disso, podem permitir a criação de novos marcadores selecionáveis para uso em biologia molecular arqueana.[285]
Referências[editar | editar código-fonte]
- ↑ Woese CR, Kandler O, Wheelis ML (Junho 1990). «Towards a natural system of organisms: proposal for the domains Archaea, Bacteria, and Eucarya». Proceedings of the National Academy of Sciences of the United States of America. 87 (12): 4576–9. Bibcode:1990PNAS...87.4576W. PMC 54159
 . PMID 2112744. doi:10.1073/pnas.87.12.4576
. PMID 2112744. doi:10.1073/pnas.87.12.4576
- ↑ a b Petitjean C, Deschamps P, López-García P, Moreira D (dezembro 2014). «Rooting the domain archaea by phylogenomic analysis supports the foundation of the new kingdom Proteoarchaeota». Genome Biology and Evolution. 7 (1): 191–204. PMC 4316627
 . PMID 25527841. doi:10.1093/gbe/evu274
. PMID 25527841. doi:10.1093/gbe/evu274
- ↑ a b «NCBI taxonomy page on Archaea»
- ↑ Merriam-Webster Online Dictionary
- ↑ FONSECA, F. V. Peixoto da (28 de março de 2000). ««Archeae»». Consultado em 25 de agosto de 2009
- ↑ «O SURPREENDENTE DOMÍNIO ARQUEA». Jornal de Notícias. 25 de maio de 2000. Consultado em 25 de agosto de 2009
- ↑ «Fisiologia de Microrganismos» (PDF). Consultado em 25 de agosto de 2009
- ↑ a b FONSECA, F. V. Peixoto da (4 de abril de 2000). «Arquaia». Consultado em 25 de agosto de 2009
- ↑ Pace, Norman R. (17 de maio de 2006). «Time for a change». Nature (em inglês). 441 (7091): 289–289. ISSN 0028-0836. doi:10.1038/441289a
- ↑ A. L. Hartman et al.: The Complete Genome Sequence of Haloferax volcanii DS2, a Model Archaeon In: PLOS ONE. Band 5, Nr. 3, 2010, S. e9605 PMC 2841640
- ↑ Walsh, David A.; Doolittle, W. Ford (12 de abril de 2005). «The real 'domains' of life». Current Biology. 15 (7). doi:10.1016/j.cub.2005.03.034. Consultado em 19 de julho de 2015
- ↑ Stoeckenius W (outubro 1981). «Walsby's square bacterium: fine structure of an orthogonal procaryote». Journal of Bacteriology. 148 (1): 352–60. PMC 216199
 . PMID 7287626. doi:10.1128/JB.148.1.352-360.1981
. PMID 7287626. doi:10.1128/JB.148.1.352-360.1981
- ↑ «Archaea Basic Biology». Março 2018
- ↑ a b c Krieg, Noel (2005). Bergey’s Manual® of Systematic Bacteriology. USA: Springer. pp. 21–6. ISBN 978-0-387-24143-2
- ↑ Barns S, Burggraf S (1997). «Crenarchaeota». The Tree of Life Web Project. Version 01 January 1997
- ↑ Walsby, A.E. (1980). «A square bacterium». Nature. 283 (5742): 69–71. doi:10.1038/283069a0
- ↑ Hara F, Yamashiro K, Nemoto N,; et al. (2007). «An actin homolog of the archaeon Thermoplasma acidophilum that retains the ancient characteristics of eukaryotic actin». J. Bacteriol. 189 (5): 2039–45. PMID 17189356. doi:10.1128/JB.01454-06
- ↑ Trent JD, Kagawa HK, Yaoi T, Olle E, Zaluzec NJ (Maio 1997). «Chaperonin filaments: the archaeal cytoskeleton?». Proceedings of the National Academy of Sciences of the United States of America. 94 (10): 5383–88. Bibcode:1997PNAS...94.5383T. PMC 24687
 . PMID 9144246. doi:10.1073/pnas.94.10.5383
. PMID 9144246. doi:10.1073/pnas.94.10.5383
- ↑ Hixon WG, Searcy DG (1993). «Cytoskeleton in the archaebacterium Thermoplasma acidophilum? Viscosity increase in soluble extracts». Bio Systems. 29 (2–3): 151–60. PMID 8374067. doi:10.1016/0303-2647(93)90091-P
- ↑ a b Golyshina OV, Pivovarova TA, Karavaiko GI, Kondratéva TF, Moore ER, Abraham WR, et al. (Maio 2000). «Ferroplasma acidiphilum gen. nov., sp. nov., an acidophilic, autotrophic, ferrous-iron-oxidizing, cell-wall-lacking, mesophilic member of the Ferroplasmaceae fam. nov., comprising a distinct lineage of the Archaea». International Journal of Systematic and Evolutionary Microbiology. 50 (3): 997–1006. PMID 10843038. doi:10.1099/00207713-50-3-997

- ↑ Hall-Stoodley L, Costerton JW, Stoodley P (fevereiro 2004). «Bacterial biofilms: from the natural environment to infectious diseases». Nature Reviews. Microbiology. 2 (2): 95–108. PMID 15040259. doi:10.1038/nrmicro821
- ↑ Kuwabara T, Minaba M, Iwayama Y, Inouye I, Nakashima M, Marumo K, et al. (Novembro 2005). «Thermococcus coalescens sp. nov., a cell-fusing hyperthermophilic archaeon from Suiyo Seamount». International Journal of Systematic and Evolutionary Microbiology. 55 (Pt 6): 2507–14. PMID 16280518. doi:10.1099/ijs.0.63432-0

- ↑ Nickell S, Hegerl R, Baumeister W, Rachel R (2003). «Pyrodictium cannulae enter the periplasmic space but do not enter the cytoplasm, as revealed by cryo-electron tomography». J. Struct. Biol. 141 (1): 34–42. PMID 12576018. doi:10.1016/S1047-8477(02)00581-6
- ↑ Horn C, Paulmann B, Kerlen G, Junker N, Huber H (agosto 1999). «In vivo observation of cell division of anaerobic hyperthermophiles by using a high-intensity dark-field microscope». Journal of Bacteriology. 181 (16): 5114–18. PMC 94007
 . PMID 10438790. doi:10.1128/JB.181.16.5114-5118.1999
. PMID 10438790. doi:10.1128/JB.181.16.5114-5118.1999
- ↑ Rudolph C, Wanner G, Huber R (maio de 2001). «Natural communities of novel archaea and bacteria growing in cold sulfurous springs with a string-of-pearls-like morphology». Appl. Environ. Microbiol. 67 (5): 2336–44. PMC 92875
 . PMID 11319120. doi:10.1128/AEM.67.5.2336-2344.2001
. PMID 11319120. doi:10.1128/AEM.67.5.2336-2344.2001
- ↑ a b c Woese CR (março de 1994). «There must be a prokaryote somewhere: microbiology's search for itself». Microbiol. Rev. 58 (1): 1–9. PMC 372949
 . PMID 8177167
. PMID 8177167
- ↑ a b Thomas NA, Bardy SL, Jarrell KF (Abril 2001). «The archaeal flagellum: a different kind of prokaryotic motility structure». FEMS Microbiology Reviews. 25 (2): 147–74. PMID 11250034. doi:10.1111/j.1574-6976.2001.tb00575.x

- ↑ Rachel R, Wyschkony I, Riehl S, Huber H (março de 2002). «The ultrastructure of Ignicoccus: evidence for a novel outer membrane and for intracellular vesicle budding in an archaeon» (PDF). Archaea. 1 (1): 9–18. PMID 15803654
- ↑ Sára M, Sleytr UB (fevereiro 2000). «S-Layer proteins». Journal of Bacteriology. 182 (4): 859–68. PMC 94357
 . PMID 10648507. doi:10.1128/JB.182.4.859-868.2000
. PMID 10648507. doi:10.1128/JB.182.4.859-868.2000
- ↑ Engelhardt H, Peters J (dezembro 1998). «Structural research on surface layers: a focus on stability, surface layer homology domains, and surface layer-cell wall interactions». Journal of Structural Biology. 124 (2–3): 276–302. PMID 10049812. doi:10.1006/jsbi.1998.4070
- ↑ a b Kandler O, König H (Abril 1998). «Cell wall polymers in Archaea (Archaebacteria)». Cellular and Molecular Life Sciences. 54 (4): 305–08. PMID 9614965. doi:10.1007/s000180050156
- ↑ Howland JL (2000). The Surprising Archaea: Discovering Another Domain of Life. Oxford: Oxford University Press. p. 32. ISBN 978-0-19-511183-5
- ↑ Gophna U, Ron EZ, Graur D (Julho 2003). «Bacterial type III secretion systems are ancient and evolved by multiple horizontal-transfer events». Gene. 312: 151–63. PMID 12909351. doi:10.1016/S0378-1119(03)00612-7
- ↑ Nguyen L, Paulsen IT, Tchieu J, Hueck CJ, Saier MH (Abril 2000). «Phylogenetic analyses of the constituents of Type III protein secretion systems». Journal of Molecular Microbiology and Biotechnology. 2 (2): 125–44. PMID 10939240
- ↑ Ng SY, Chaban B, Jarrell KF (2006). «Archaeal flagella, bacterial flagella and type IV pili: a comparison of genes and posttranslational modifications». Journal of Molecular Microbiology and Biotechnology. 11 (3–5): 167–91. PMID 16983194. doi:10.1159/000094053
- ↑ Bardy SL, Ng SY, Jarrell KF (Fevereiro 2003). «Prokaryotic motility structures» (PDF). Microbiology. 149 (Pt 2): 295–304. PMID 12624192. doi:10.1099/mic.0.25948-0. Cópia arquivada (PDF) em 7 de março de 2019
- ↑ a b Koga Y, Morii H (Março 2007). «Biosynthesis of ether-type polar lipids in archaea and evolutionary considerations». Microbiology and Molecular Biology Reviews. 71 (1): 97–120. PMC 1847378
 . PMID 17347520. doi:10.1128/MMBR.00033-06
. PMID 17347520. doi:10.1128/MMBR.00033-06
- ↑ Youssefian S, Rahbar N, Van Dessel S (Maio 2018). «Thermal conductivity and rectification in asymmetric archaeal lipid membranes». The Journal of Chemical Physics. 148 (17). 174901 páginas. Bibcode:2018JChPh.148q4901Y. PMID 29739208. doi:10.1063/1.5018589
- ↑ De Rosa M, Gambacorta A, Gliozzi A (Março 1986). «Structure, biosynthesis, and physicochemical properties of archaebacterial lipids». Microbiological Reviews. 50 (1): 70–80. PMC 373054
 . PMID 3083222. doi:10.1128/MMBR.50.1.70-80.1986
. PMID 3083222. doi:10.1128/MMBR.50.1.70-80.1986
- ↑ Balleza D, Garcia-Arribas AB, Sot J, Ruiz-Mirazo K, Goñi FM (setembro 2014). «Ether- versus ester-linked phospholipid bilayers containing either linear or branched apolar chains». Biophysical Journal. 107 (6): 1364–74. Bibcode:2014BpJ...107.1364B. PMC 4167531
 . PMID 25229144. doi:10.1016/j.bpj.2014.07.036
. PMID 25229144. doi:10.1016/j.bpj.2014.07.036
- ↑ Damsté JS, Schouten S, Hopmans EC, van Duin AC, Geenevasen JA (Outubro 2002). «Crenarchaeol: The characteristic core glycerol dibiphytanyl glycerol tetraether membrane lipid of cosmopolitan pelagic crenarchaeota». Journal of Lipid Research. 43 (10): 1641–51. PMID 12364548. doi:10.1194/jlr.M200148-JLR200

- ↑ Koga Y, Morii H (Novembro 2005). «Recent advances in structural research on ether lipids from archaea including comparative and physiological aspects». Bioscience, Biotechnology, and Biochemistry. 69 (11): 2019–34. PMID 16306681. doi:10.1271/bbb.69.2019
- ↑ Hanford MJ, Peeples TL (janeiro 2002). «Archaeal tetraether lipids: unique structures and applications». Applied Biochemistry and Biotechnology. 97 (1): 45–62. PMID 11900115. doi:10.1385/ABAB:97:1:45
- ↑ Macalady JL, Vestling MM, Baumler D, Boekelheide N, Kaspar CW, Banfield JF (Outubro 2004). «Tetraether-linked membrane monolayers in Ferroplasma spp: a key to survival in acid». Extremophiles. 8 (5): 411–19. PMID 15258835. doi:10.1007/s00792-004-0404-5
- ↑ Baseado em PDB 1FBB, publicado em Subramaniam S, Henderson R (agosto 2000). «Molecular mechanism of vectorial proton translocation by bacteriorhodopsin». Nature. 406 (6796): 653–57. Bibcode:2000Natur.406..653S. PMID 10949309. doi:10.1038/35020614
- ↑ a b c Valentine DL (Abril 2007). «Adaptations to energy stress dictate the ecology and evolution of the Archaea». Nature Reviews. Microbiology. 5 (4): 316–23. PMID 17334387. doi:10.1038/nrmicro1619
- ↑ a b c Schäfer G, Engelhard M, Müller V (setembro 1999). «Bioenergetics of the Archaea». Microbiology and Molecular Biology Reviews. 63 (3): 570–620. PMC 103747
 . PMID 10477309. doi:10.1128/MMBR.63.3.570-620.1999
. PMID 10477309. doi:10.1128/MMBR.63.3.570-620.1999
- ↑ Zillig W (dezembro 1991). «Comparative biochemistry of Archaea and Bacteria». Current Opinion in Genetics & Development. 1 (4): 544–51. PMID 1822288. doi:10.1016/S0959-437X(05)80206-0
- ↑ Romano AH, Conway T (1996). «Evolution of carbohydrate metabolic pathways». Research in Microbiology. 147 (6–7): 448–55. PMID 9084754. doi:10.1016/0923-2508(96)83998-2
- ↑ Koch AL (1998). How did bacteria come to be?. Col: Advances in Microbial Physiology. 40. [S.l.: s.n.] pp. 353–99. ISBN 978-0-12-027740-7. PMID 9889982. doi:10.1016/S0065-2911(08)60135-6
- ↑ DiMarco AA, Bobik TA, Wolfe RS (1990). «Unusual coenzymes of methanogenesis». Annual Review of Biochemistry. 59: 355–94. PMID 2115763. doi:10.1146/annurev.bi.59.070190.002035
- ↑ Klocke M, Nettmann E, Bergmann I, Mundt K, Souidi K, Mumme J, et al. (Agosto 2008). «Characterization of the methanogenic Archaea within two-phase biogas reactor systems operated with plant biomass». Systematic and Applied Microbiology. 31 (3): 190–205. PMID 18501543. doi:10.1016/j.syapm.2008.02.003
- ↑ Mueller-Cajar O, Badger MR (agosto 2007). «New roads lead to Rubisco in archaebacteria». BioEssays. 29 (8): 722–24. PMID 17621634. doi:10.1002/bies.20616
- ↑ Berg IA, Kockelkorn D, Buckel W, Fuchs G (dezembro 2007). «A 3-hydroxypropionate/ 4-hydroxybutyrate autotrophic carbon dioxide assimilation pathway in Archaea» (PDF). Science. 318 (5857): 1782–86. Bibcode:2007Sci...318.1782B. PMID 18079405. doi:10.1126/science.1149976
- ↑ Thauer RK (dezembro 2007). «Microbiology. A fifth pathway of carbon fixation». Science. 318 (5857): 1732–33. PMID 18079388. doi:10.1126/science.1152209
- ↑ Bryant DA, Frigaard NU (Novembro 2006). «Prokaryotic photosynthesis and phototrophy illuminated». Trends in Microbiology. 14 (11): 488–96. PMID 16997562. doi:10.1016/j.tim.2006.09.001
- ↑ a b Könneke M, Bernhard AE, de la Torre JR, Walker CB, Waterbury JB, Stahl DA (setembro 2005). «Isolation of an autotrophic ammonia-oxidizing marine archaeon». Nature. 437 (7058): 543–46. Bibcode:2005Natur.437..543K. PMID 16177789. doi:10.1038/nature03911
- ↑ a b Francis CA, Beman JM, Kuypers MM (Maio 2007). «New processes and players in the nitrogen cycle: the microbial ecology of anaerobic and archaeal ammonia oxidation». The ISME Journal. 1 (1): 19–27. PMID 18043610. doi:10.1038/ismej.2007.8

- ↑ Lanyi JK (2004). «Bacteriorhodopsin». Annual Review of Physiology. 66: 665–88. PMID 14977418. doi:10.1146/annurev.physiol.66.032102.150049
- ↑ Xiang X, Chen L, Huang X, Luo Y, She Q, Huang L (Julho 2005). «Sulfolobus tengchongensis spindle-shaped virus STSV1: virus-host interactions and genomic features». Journal of Virology. 79 (14): 8677–86. PMC 1168784
 . PMID 15994761. doi:10.1128/JVI.79.14.8677-8686.2005
. PMID 15994761. doi:10.1128/JVI.79.14.8677-8686.2005
- ↑ a b Allers T, Mevarech M (janeiro 2005). «Archaeal genetics – the third way» (PDF). Nature Reviews Genetics. 6 (1): 58–73. PMID 15630422. doi:10.1038/nrg1504
- ↑ Galagan JE, Nusbaum C, Roy A, Endrizzi MG, Macdonald P, FitzHugh W, et al. (Abril 2002). «The genome of M. acetivorans reveals extensive metabolic and physiological diversity». Genome Research. 12 (4): 532–42. PMC 187521
 . PMID 11932238. doi:10.1101/gr.223902
. PMID 11932238. doi:10.1101/gr.223902
- ↑ a b Waters E, Hohn MJ, Ahel I, Graham DE, Adams MD, Barnstead M, et al. (Outubro 2003). «The genome of Nanoarchaeum equitans: insights into early archaeal evolution and derived parasitism». Proceedings of the National Academy of Sciences of the United States of America. 100 (22): 12984–88. Bibcode:2003PNAS..10012984W. PMC 240731
 . PMID 14566062. doi:10.1073/pnas.1735403100
. PMID 14566062. doi:10.1073/pnas.1735403100
- ↑ Schleper C, Holz I, Janekovic D, Murphy J, Zillig W (Agosto 1995). «A multicopy plasmid of the extremely thermophilic archaeon Sulfolobus effects its transfer to recipients by mating». Journal of Bacteriology. 177 (15): 4417–26. PMC 177192
 . PMID 7635827. doi:10.1128/jb.177.15.4417-4426.1995
. PMID 7635827. doi:10.1128/jb.177.15.4417-4426.1995
- ↑ Sota M, Top EM (2008). «Horizontal Gene Transfer Mediated by Plasmids». Plasmids: Current Research and Future Trends. [S.l.]: Caister Academic Press. ISBN 978-1-904455-35-6
- ↑ Graham DE, Overbeek R, Olsen GJ, Woese CR (Março 2000). «An archaeal genomic signature». Proceedings of the National Academy of Sciences of the United States of America. 97 (7): 3304–08. Bibcode:2000PNAS...97.3304G. PMC 16234
 . PMID 10716711. doi:10.1073/pnas.050564797
. PMID 10716711. doi:10.1073/pnas.050564797
- ↑ a b Gaasterland T (Outubro 1999). «Archaeal genomics». Current Opinion in Microbiology. 2 (5): 542–47. PMID 10508726. doi:10.1016/S1369-5274(99)00014-4
- ↑ Dennis PP (Junho 1997). «Ancient ciphers: translation in Archaea». Cell. 89 (7): 1007–10. PMID 9215623. doi:10.1016/S0092-8674(00)80288-3

- ↑ Werner F (setembro 2007). «Structure and function of archaeal RNA polymerases». Molecular Microbiology. 65 (6): 1395–404. PMID 17697097. doi:10.1111/j.1365-2958.2007.05876.x

- ↑ Aravind L, Koonin EV (dezembro 1999). «DNA-binding proteins and evolution of transcription regulation in the archaea». Nucleic Acids Research. 27 (23): 4658–70. PMC 148756
 . PMID 10556324. doi:10.1093/nar/27.23.4658
. PMID 10556324. doi:10.1093/nar/27.23.4658
- ↑ Lykke-Andersen J, Aagaard C, Semionenkov M, Garrett RA (Setembro 1997). «Archaeal introns: splicing, intercellular mobility and evolution». Trends in Biochemical Sciences. 22 (9): 326–31. PMID 9301331. doi:10.1016/S0968-0004(97)01113-4
- ↑ Watanabe Y, Yokobori S, Inaba T, Yamagishi A, Oshima T, Kawarabayasi Y, et al. (Janeiro 2002). «Introns in protein-coding genes in Archaea». FEBS Letters. 510 (1–2): 27–30. PMID 11755525. doi:10.1016/S0014-5793(01)03219-7
- ↑ Yoshinari S, Itoh T, Hallam SJ, DeLong EF, Yokobori S, Yamagishi A, et al. (Agosto 2006). «Archaeal pre-mRNA splicing: a connection to hetero-oligomeric splicing endonuclease». Biochemical and Biophysical Research Communications. 346 (3): 1024–32. PMID 16781672. doi:10.1016/j.bbrc.2006.06.011
- ↑ Rosenshine I, Tchelet R, Mevarech M (Setembro 1989). «The mechanism of DNA transfer in the mating system of an archaebacterium». Science. 245 (4924): 1387–89. Bibcode:1989Sci...245.1387R. PMID 2818746. doi:10.1126/science.2818746
- ↑ a b c Fröls S, Ajon M, Wagner M, Teichmann D, Zolghadr B, Folea M, et al. (Novembro 2008). «UV-inducible cellular aggregation of the hyperthermophilic archaeon Sulfolobus solfataricus is mediated by pili formation». Molecular Microbiology. 70 (4): 938–52. PMID 18990182. doi:10.1111/j.1365-2958.2008.06459.x

- ↑ a b c Ajon M, Fröls S, van Wolferen M, Stoecker K, Teichmann D, Driessen AJ, et al. (Novembro 2011). «UV-inducible DNA exchange in hyperthermophilic archaea mediated by type IV pili» (PDF). Molecular Microbiology. 82 (4): 807–17. PMID 21999488. doi:10.1111/j.1365-2958.2011.07861.x

- ↑ Fröls S, White MF, Schleper C (fevereiro 2009). «Reactions to UV damage in the model archaeon Sulfolobus solfataricus». Biochemical Society Transactions. 37 (Pt 1): 36–41. PMID 19143598. doi:10.1042/BST0370036
- ↑ Bernstein H, Bernstein C (2017). «Sexual Communication in Archaea, the Precursor to Eukaryotic Meiosis». In: Witzany G. Biocommunication of Archaea. [S.l.]: Springer Nature. pp. 301–117. ISBN 978-3-319-65535-2. doi:10.1007/978-3-319-65536-9_7
- ↑ Blohs M, Moissl-Eichinger C, Mahnert A, Spang A, Dombrowski N, Krupovic M, Klingl A (Janeiro 2019). «Archaea – An Introduction». In: Schmidt TM. Encyclopedia of Microbiology (em inglês) Fourth ed. Academic Press. pp. 243–252. ISBN 978-0-12-811737-8. doi:10.1016/B978-0-12-809633-8.20884-4
- ↑ a b c d Krupovic M, Cvirkaite-Krupovic V, Iranzo J, Prangishvili D, Koonin EV (Janeiro 2018). «Viruses of archaea: Structural, functional, environmental and evolutionary genomics». Virus Research. 244: 181–193. PMC 5801132
 . PMID 29175107. doi:10.1016/j.virusres.2017.11.025
. PMID 29175107. doi:10.1016/j.virusres.2017.11.025
- ↑ Pietilä MK, Demina TA, Atanasova NS, Oksanen HM, Bamford DH (Junho 2014). «Archaeal viruses and bacteriophages: comparisons and contrasts». Trends in Microbiology. 22 (6): 334–44. PMID 24647075. doi:10.1016/j.tim.2014.02.007
- ↑ a b Woese, Carl R.; Kandler, Otto; Wheelis, Mark L. «Towards a natural system of organisms: proposal for the domains Archaea, Bacteria, and Eucarya.». Proceedings of the National Academy of Sciences. Consultado em 19 de julho de 2015
- ↑ Principi N, Silvestri E, Esposito S (2019). «Advantages and Limitations of Bacteriophages for the Treatment of Bacterial Infections». Frontiers in Pharmacology (em inglês). 10. 513 páginas. PMC 6517696
 . PMID 31139086. doi:10.3389/fphar.2019.00513
. PMID 31139086. doi:10.3389/fphar.2019.00513
- ↑ Prangishvili D (1 de janeiro de 2013). «Viruses of the Archaea». In: Maloy S, Hughes K. Brenner's Encyclopedia of Genetics. Brenner's Encyclopedia of Genetics (Second Edition) (em inglês). Academic Press. pp. 295–298. ISBN 978-0-08-096156-9. doi:10.1016/B978-0-12-374984-0.01627-2. Consultado em 16 de março de 2020
- ↑ a b Prangishvili D, Garrett RA (Abril 2004). «Exceptionally diverse morphotypes and genomes of crenarchaeal hyperthermophilic viruses» (PDF). Biochemical Society Transactions. 32 (Pt 2): 204–08. PMID 15046572. doi:10.1042/BST0320204
- ↑ Pietilä MK, Roine E, Paulin L, Kalkkinen N, Bamford DH (Abril 2009). «An ssDNA virus infecting archaea: a new lineage of viruses with a membrane envelope». Molecular Microbiology. 72 (2): 307–19. PMID 19298373. doi:10.1111/j.1365-2958.2009.06642.x
- ↑ Mochizuki T, Krupovic M, Pehau-Arnaudet G, Sako Y, Forterre P, Prangishvili D (Agosto 2012). «Archaeal virus with exceptional virion architecture and the largest single-stranded DNA genome». Proceedings of the National Academy of Sciences of the United States of America. 109 (33): 13386–91. Bibcode:2012PNAS..10913386M. PMC 3421227
 . PMID 22826255. doi:10.1073/pnas.1203668109
. PMID 22826255. doi:10.1073/pnas.1203668109
- ↑ Mojica FJ, Díez-Villaseñor C, García-Martínez J, Soria E (Fevereiro 2005). «Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements». Journal of Molecular Evolution. 60 (2): 174–82. Bibcode:2005JMolE..60..174M. PMID 15791728. doi:10.1007/s00239-004-0046-3
- ↑ Makarova KS, Grishin NV, Shabalina SA, Wolf YI, Koonin EV (Março 2006). «A putative RNA-interference-based immune system in prokaryotes: computational analysis of the predicted enzymatic machinery, functional analogies with eukaryotic RNAi, and hypothetical mechanisms of action». Biology Direct. 1. 7 páginas. PMC 1462988
 . PMID 16545108. doi:10.1186/1745-6150-1-7
. PMID 16545108. doi:10.1186/1745-6150-1-7
- ↑ Krieg N (2005). Bergey's Manual of Systematic Bacteriology. US: Springer. pp. 21–26. ISBN 978-0-387-24143-2
- ↑ a b Bernander R (Agosto 1998). «Archaea and the cell cycle». Molecular Microbiology. 29 (4): 955–61. PMID 9767564. doi:10.1046/j.1365-2958.1998.00956.x
- ↑ Kelman LM, Kelman Z (setembro 2004). «Multiple origins of replication in archaea». Trends in Microbiology. 12 (9): 399–401. PMID 15337158. doi:10.1016/j.tim.2004.07.001
- ↑
Lindås AC, Karlsson EA, Lindgren MT, Ettema TJ, Bernander R (Dezembro 2008). «A unique cell division machinery in the Archaea». Proceedings of the National Academy of Sciences of the United States of America. 105 (48): 18942–46. Bibcode:2008PNAS..10518942L. PMC 2596248
 . PMID 18987308. doi:10.1073/pnas.0809467105
. PMID 18987308. doi:10.1073/pnas.0809467105
- ↑ Samson RY, Obita T, Freund SM, Williams RL, Bell SD (Dezembro 2008). «A role for the ESCRT system in cell division in archaea». Science. 322 (5908): 1710–13. Bibcode:2008Sci...322.1710S. PMC 4121953
 . PMID 19008417. doi:10.1126/science.1165322
. PMID 19008417. doi:10.1126/science.1165322
- ↑ Pelve EA, Lindås AC, Martens-Habbena W, de la Torre JR, Stahl DA, Bernander R (Novembro 2011). «Cdv-based cell division and cell cycle organization in the thaumarchaeon Nitrosopumilus maritimus». Molecular Microbiology. 82 (3): 555–66. PMID 21923770. doi:10.1111/j.1365-2958.2011.07834.x

- ↑ Caspi Y, Dekker C (2018). «Dividing the Archaeal Way: The Ancient Cdv Cell-Division Machinery». Frontiers in Microbiology. 9. 174 páginas. PMC 5840170
 . PMID 29551994. doi:10.3389/fmicb.2018.00174
. PMID 29551994. doi:10.3389/fmicb.2018.00174
- ↑ Onyenwoke RU, Brill JA, Farahi K, Wiegel J (Outubro 2004). «Sporulation genes in members of the low G+C Gram-type-positive phylogenetic branch ( Firmicutes)». Archives of Microbiology. 182 (2–3): 182–92. PMID 15340788. doi:10.1007/s00203-004-0696-y
- ↑ Kostrikina NA, Zvyagintseva IS, Duda VI (1991). «Cytological peculiarities of some extremely halophilic soil archaeobacteria». Arch. Microbiol. 156 (5): 344–49. doi:10.1007/BF00248708
- ↑ a b c d Chow C, Padda KP, Puri A, Chanway CP (setembro 2022). «An Archaic Approach to a Modern Issue: Endophytic Archaea for Sustainable Agriculture». Current Microbiology. 79 (11). 322 páginas. PMID 36125558. doi:10.1007/s00284-022-03016-y
- ↑ Bang C, Schmitz RA (setembro 2015). «Archaea associated with human surfaces: not to be underestimated». FEMS Microbiology Reviews. 39 (5): 631–48. PMID 25907112. doi:10.1093/femsre/fuv010

- ↑ Moissl-Eichinger C, Pausan M, Taffner J, Berg G, Bang C, Schmitz RA (Janeiro 2018). «Archaea Are Interactive Components of Complex Microbiomes». Trends in Microbiology. 26 (1): 70–85. PMID 28826642. doi:10.1016/j.tim.2017.07.004
- ↑ Rajput A, Kumar M (2017). «Computational Exploration of Putative LuxR Solos in Archaea and Their Functional Implications in Quorum Sensing». Frontiers in Microbiology (em English). 8. 798 páginas. PMC 5413776
 . PMID 28515720. doi:10.3389/fmicb.2017.00798
. PMID 28515720. doi:10.3389/fmicb.2017.00798
- ↑ a b c DeLong EF (1998). «Everything in moderation: archaea as 'non-extremophiles'». Curr. Opin. Genet. Dev. 8 (6): 649–54. PMID 9914204. doi:10.1016/S0959-437X(98)80032-4
- ↑ DeLong EF, Pace NR (Agosto 2001). «Environmental diversity of bacteria and archaea». Systematic Biology. 50 (4): 470–78. CiteSeerX 10.1.1.321.8828
 . PMID 12116647. doi:10.1080/106351501750435040
. PMID 12116647. doi:10.1080/106351501750435040
- ↑ a b Pikuta EV, Hoover RB, Tang J (2007). «Microbial extremophiles at the limits of life». Critical Reviews in Microbiology. 33 (3): 183–209. PMID 17653987. doi:10.1080/10408410701451948
- ↑ Adam PS, Borrel G, Brochier-Armanet C, Gribaldo S (Novembro 2017). «The growing tree of Archaea: new perspectives on their diversity, evolution and ecology». The ISME Journal. 11 (11): 2407–2425. PMC 5649171
 . PMID 28777382. doi:10.1038/ismej.2017.122
. PMID 28777382. doi:10.1038/ismej.2017.122
- ↑ Madigan MT, Martino JM (2006). Brock Biology of Microorganisms 11th ed. [S.l.]: Pearson. p. 136. ISBN 978-0-13-196893-6
- ↑ Takai K, Nakamura K, Toki T, Tsunogai U, Miyazaki M, Miyazaki J, Hirayama H, Nakagawa S, Nunoura T, Horikoshi K (Agosto 2008). «Cell proliferation at 122 °C and isotopically heavy CH4 production by a hyperthermophilic methanogen under high-pressure cultivation». Proceedings of the National Academy of Sciences of the United States of America. 105 (31): 10949–54. Bibcode:2008PNAS..10510949T. PMC 2490668
 . PMID 18664583. doi:10.1073/pnas.0712334105
. PMID 18664583. doi:10.1073/pnas.0712334105
- ↑ Ciaramella M, Napoli A, Rossi M (Fevereiro 2005). «Another extreme genome: how to live at pH 0». Trends in Microbiology. 13 (2): 49–51. PMID 15680761. doi:10.1016/j.tim.2004.12.001
- ↑ Javaux EJ (2006). «Extreme life on Earth--past, present and possibly beyond». Research in Microbiology. 157 (1): 37–48. PMID 16376523. doi:10.1016/j.resmic.2005.07.008
- ↑ Nealson KH (Janeiro 1999). «Post-Viking microbiology: new approaches, new data, new insights» (PDF). Origins of Life and Evolution of the Biosphere. 29 (1): 73–93. Bibcode:1999OLEB...29...73N. PMID 11536899. doi:10.1023/A:1006515817767. Consultado em 24 Junho 2008. Cópia arquivada (PDF) em 16 Outubro 2019
- ↑ Davies PC (1996). «The transfer of viable microorganisms between planets». Ciba Foundation Symposium. Novartis Foundation Symposia. 202: 304–14; discussion 314–17. ISBN 9780470514986. PMID 9243022. doi:10.1002/9780470514986.ch16
- ↑ López-García P, López-López A, Moreira D, Rodríguez-Valera F (Julho 2001). «Diversity of free-living prokaryotes from a deep-sea site at the Antarctic Polar Front». FEMS Microbiology Ecology. 36 (2–3): 193–202. PMID 11451524. doi:10.1016/s0168-6496(01)00133-7
- ↑ Karner MB, DeLong EF, Karl DM (Janeiro 2001). «Archaeal dominance in the mesopelagic zone of the Pacific Ocean». Nature. 409 (6819): 507–10. Bibcode:2001Natur.409..507K. PMID 11206545. doi:10.1038/35054051
- ↑ Giovannoni SJ, Stingl U (Setembro 2005). «Molecular diversity and ecology of microbial plankton». Nature. 437 (7057): 343–48. Bibcode:2005Natur.437..343G. PMID 16163344. doi:10.1038/nature04158
- ↑ DeLong EF, Karl DM (Setembro 2005). «Genomic perspectives in microbial oceanography». Nature. 437 (7057): 336–42. Bibcode:2005Natur.437..336D. PMID 16163343. doi:10.1038/nature04157
- ↑ Agogué H, Brink M, Dinasquet J, Herndl GJ (Dezembro 2008). «Major gradients in putatively nitrifying and non-nitrifying Archaea in the deep North Atlantic» (PDF). Nature. 456 (7223): 788–91. Bibcode:2008Natur.456..788A. PMID 19037244. doi:10.1038/nature07535
- ↑ Teske A, Sørensen KB (Janeiro 2008). «Uncultured archaea in deep marine subsurface sediments: have we caught them all?». The ISME Journal. 2 (1): 3–18. PMID 18180743. doi:10.1038/ismej.2007.90
 . hdl:10379/14139
. hdl:10379/14139
- ↑ Lipp JS, Morono Y, Inagaki F, Hinrichs KU (Agosto 2008). «Significant contribution of Archaea to extant biomass in marine subsurface sediments». Nature. 454 (7207): 991–94. Bibcode:2008Natur.454..991L. PMID 18641632. doi:10.1038/nature07174
- ↑ Danovaro R, Dell'Anno A, Corinaldesi C, Rastelli E, Cavicchioli R, Krupovic M, Noble RT, Nunoura T, Prangishvili D (Outubro 2016). «Virus-mediated archaeal hecatomb in the deep seafloor». Science Advances. 2 (10): e1600492. Bibcode:2016SciA....2E0492D. PMC 5061471
 . PMID 27757416. doi:10.1126/sciadv.1600492
. PMID 27757416. doi:10.1126/sciadv.1600492
- ↑ Liu X, Pan J, Liu Y, Li M, Gu JD (Outubro 2018). «Diversity and distribution of Archaea in global estuarine ecosystems». The Science of the Total Environment. 637–638: 349–358. Bibcode:2018ScTEn.637..349L. PMID 29753224. doi:10.1016/j.scitotenv.2018.05.016
- ↑ Cabello P, Roldán MD, Moreno-Vivián C (Novembro 2004). «Nitrate reduction and the nitrogen cycle in archaea». Microbiology. 150 (Pt 11): 3527–46. PMID 15528644. doi:10.1099/mic.0.27303-0. Consultado em 27 Junho 2008. Cópia arquivada em 7 Junho 2011
- ↑ Mehta MP, Baross JA (Dezembro 2006). «Nitrogen fixation at 92 °C by a hydrothermal vent archaeon». Science. 314 (5806): 1783–86. Bibcode:2006Sci...314.1783M. PMID 17170307. doi:10.1126/science.1134772
- ↑ Coolen MJ, Abbas B, van Bleijswijk J, Hopmans EC, Kuypers MM, Wakeham SG, et al. (Abril 2007). «Putative ammonia-oxidizing Crenarchaeota in suboxic waters of the Black Sea: A basin-wide ecological study using 16S ribosomal and functional genes and membrane lipids». Environmental Microbiology. 9 (4): 1001–16. PMID 17359272. doi:10.1111/j.1462-2920.2006.01227.x. hdl:1912/2034

- ↑ Leininger S, Urich T, Schloter M, Schwark L, Qi J, Nicol GW, Prosser JI, Schuster SC, Schleper C (Agosto 2006). «Archaea predominate among ammonia-oxidizing prokaryotes in soils». Nature. 442 (7104): 806–09. Bibcode:2006Natur.442..806L. PMID 16915287. doi:10.1038/nature04983
- ↑ Baker BJ, Banfield JF (Maio 2003). «Microbial communities in acid mine drainage». FEMS Microbiology Ecology. 44 (2): 139–52. PMID 19719632. doi:10.1016/S0168-6496(03)00028-X

- ↑ Schimel J (Agosto 2004). «Playing scales in the methane cycle: from microbial ecology to the globe». Proceedings of the National Academy of Sciences of the United States of America. 101 (34): 12400–01. Bibcode:2004PNAS..10112400S. PMC 515073
 . PMID 15314221. doi:10.1073/pnas.0405075101
. PMID 15314221. doi:10.1073/pnas.0405075101
- ↑ Eckburg PB, Lepp PW, Relman DA (Fevereiro 2003). «Archaea and their potential role in human disease». Infection and Immunity. 71 (2): 591–96. PMC 145348
 . PMID 12540534. doi:10.1128/IAI.71.2.591-596.2003
. PMID 12540534. doi:10.1128/IAI.71.2.591-596.2003
- ↑ Cavicchioli R, Curmi PM, Saunders N, Thomas T (Novembro 2003). «Pathogenic archaea: do they exist?». BioEssays. 25 (11): 1119–28. PMID 14579252. doi:10.1002/bies.10354
- ↑ Lepp PW, Brinig MM, Ouverney CC, Palm K, Armitage GC, Relman DA (Abril 2004). «Methanogenic Archaea and human periodontal disease». Proceedings of the National Academy of Sciences of the United States of America. 101 (16): 6176–81. Bibcode:2004PNAS..101.6176L. PMC 395942
 . PMID 15067114. doi:10.1073/pnas.0308766101
. PMID 15067114. doi:10.1073/pnas.0308766101
- ↑ Vianna ME, Conrads G, Gomes BP, Horz HP (Abril 2006). «Identification and quantification of archaea involved in primary endodontic infections». Journal of Clinical Microbiology. 44 (4): 1274–82. PMC 1448633
 . PMID 16597851. doi:10.1128/JCM.44.4.1274-1282.2006
. PMID 16597851. doi:10.1128/JCM.44.4.1274-1282.2006
- ↑ Jahn U, Gallenberger M, Paper W, Junglas B, Eisenreich W, Stetter KO, et al. (Março 2008). «Nanoarchaeum equitans and Ignicoccus hospitalis: new insights into a unique, intimate association of two archaea». Journal of Bacteriology. 190 (5): 1743–50. PMC 2258681
 . PMID 18165302. doi:10.1128/JB.01731-07
. PMID 18165302. doi:10.1128/JB.01731-07
- ↑ Chaban B, Ng SY, Jarrell KF (Fevereiro 2006). «Archaeal habitats--from the extreme to the ordinary». Canadian Journal of Microbiology. 52 (2): 73–116. PMID 16541146. doi:10.1139/w05-147
- ↑ Schink B (Junho 1997). «Energetics of syntrophic cooperation in methanogenic degradation». Microbiology and Molecular Biology Reviews. 61 (2): 262–80. PMC 232610
 . PMID 9184013. doi:10.1128/mmbr.61.2.262-280.1997
. PMID 9184013. doi:10.1128/mmbr.61.2.262-280.1997
- ↑ Lange M, Westermann P, Ahring BK (Fevereiro 2005). «Archaea in protozoa and metazoa». Applied Microbiology and Biotechnology. 66 (5): 465–74. PMID 15630514. doi:10.1007/s00253-004-1790-4
- ↑ van Hoek AH, van Alen TA, Sprakel VS, Leunissen JA, Brigge T, Vogels GD, et al. (Fevereiro 2000). «Multiple acquisition of methanogenic archaeal symbionts by anaerobic ciliates». Molecular Biology and Evolution. 17 (2): 251–58. PMID 10677847. doi:10.1093/oxfordjournals.molbev.a026304
- ↑ Preston CM, Wu KY, Molinski TF, DeLong EF (Junho 1996). «A psychrophilic crenarchaeon inhabits a marine sponge: Cenarchaeum symbiosum gen. nov., sp. nov». Proceedings of the National Academy of Sciences of the United States of America. 93 (13): 6241–46. Bibcode:1996PNAS...93.6241P. PMC 39006
 . PMID 8692799. doi:10.1073/pnas.93.13.6241
. PMID 8692799. doi:10.1073/pnas.93.13.6241
- ↑ Eckburg PB, Bik EM, Bernstein CN, Purdom E, Dethlefsen L, Sargent M, et al. (Junho 2005). «Diversity of the human intestinal microbial flora». Science. 308 (5728): 1635–38. Bibcode:2005Sci...308.1635E. PMC 1395357
 . PMID 15831718. doi:10.1126/science.1110591
. PMID 15831718. doi:10.1126/science.1110591
- ↑ Samuel BS, Gordon JI (Junho 2006). «A humanized gnotobiotic mouse model of host-archaeal-bacterial mutualism». Proceedings of the National Academy of Sciences of the United States of America. 103 (26): 10011–16. Bibcode:2006PNAS..10310011S. PMC 1479766
 . PMID 16782812. doi:10.1073/pnas.0602187103
. PMID 16782812. doi:10.1073/pnas.0602187103
- ↑ Wegley L, Yu Y, Breitbart M, Casas V, Kline DI, Rohwer F (2004). «Coral-associated Archaea». Marine Ecology Progress Series. 273: 89–96. Bibcode:2004MEPS..273...89W. doi:10.3354/meps273089

- ↑ Chelius MK, Triplett EW (Abril 2001). «The Diversity of Archaea and Bacteria in Association with the Roots of Zea mays L». Microbial Ecology. 41 (3): 252–263. JSTOR 4251818. PMID 11391463. doi:10.1007/s002480000087
- ↑ Simon HM, Dodsworth JA, Goodman RM (Outubro 2000). «Crenarchaeota colonize terrestrial plant roots». Environmental Microbiology. 2 (5): 495–505. PMID 11233158. doi:10.1046/j.1462-2920.2000.00131.x
- ↑ Gophna U, Charlebois RL, Doolittle WF (Maio 2004). «Have archaeal genes contributed to bacterial virulence?». Trends in Microbiology. 12 (5): 213–219. PMID 15120140. doi:10.1016/j.tim.2004.03.002
- ↑ Shiffman ME, Charalambous BM (2012). «The search for archaeal pathogens». Reviews in Medical Microbiology (em inglês). 23 (3): 45–51. ISSN 0954-139X. doi:10.1097/MRM.0b013e328353c7c9
- ↑ James G. Ferry (1993). "Methanogenesis: Ecology, Physiology, Biochemistry & Genetics". [S.l.: s.n.]
- ↑ Kluyver e van Niel 1936.
- ↑ Brock TD, Brock KM, Belly RT, Weiss RL (1972). «Sulfolobus: a new genus of sulfur-oxidizing bacteria living at low pH and high temperature». Arch. Mikrobiol. (84 (1)): 54–68. PMID 4559703. doi:10.1007/BF00408082
- ↑ "Bergey's Manual of Systematic Bacteriology". 1ª (4 vols.) ed. [S.l.: s.n.] 1984
- ↑ Lake, James A.; et al. (1984). «"Eocytes: A new ribosome structure indicates a kingdom with a close relationship to eukaryotes"». PNAS (81): 3786–3790
- ↑ Woese, C. R.; Kandler, O & Wheelis, M. L. (1990). «Towards a natural system of organisms: proposal for the domains Archaea, Bacteria, and Eucarya». Proc. Natl. Acad. Sci. U.S.A. 87 (12): 4576–9. PMID 2112744. doi:10.1073/pnas.87.12.4576
- ↑ Staley JT (novembro 2006). «The bacterial species dilemma and the genomic-phylogenetic species concept». Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 361 (1475): 1899–909. PMC 1857736
 . PMID 17062409. doi:10.1098/rstb.2006.1914
. PMID 17062409. doi:10.1098/rstb.2006.1914
- ↑ Zuckerkandl E, Pauling L (março 1965). «Molecules as documents of evolutionary history». Journal of Theoretical Biology. 8 (2): 357–66. Bibcode:1965JThBi...8..357Z. PMID 5876245. doi:10.1016/0022-5193(65)90083-4
- ↑ Parks DH, Chuvochina M, Waite DW, Rinke C, Skarshewski A, Chaumeil PA, Hugenholtz P (novembro de 2018). «A standardized bacterial taxonomy based on genome phylogeny substantially revises the tree of life». Nature Biotechnology. 36 (10): 996–1004. PMID 30148503. doi:10.1038/nbt.4229
- ↑ Woese C, Fox G (1977). «Phylogenetic structure of the prokaryotic domain: the primary kingdoms». Proc Natl Acad Sci USA. 74 (11): 5088–90. PMID 270744. doi:10.1073/pnas.74.11.5088
- ↑ a b c d Woese CR, Fox GE (Novembro 1977). «Phylogenetic structure of the prokaryotic domain: the primary kingdoms». Proceedings of the National Academy of Sciences of the United States of America. 74 (11): 5088–90. Bibcode:1977PNAS...74.5088W. PMC 432104
 . PMID 270744. doi:10.1073/pnas.74.11.5088
. PMID 270744. doi:10.1073/pnas.74.11.5088
- ↑ Woese CR, Kandler O, Wheelis ML (1990). «Towards a natural system of organisms: proposal for the domains Archaea, Bacteria, and Eucarya». Proc. Natl. Acad. Sci. U.S.A. 87 (12): 4576–9. PMID 2112744. doi:10.1073/pnas.87.12.4576
- ↑ a b Woese CR, Kandler O, Wheelis ML (Junho 1990). «Towards a natural system of organisms: proposal for the domains Archaea, Bacteria, and Eucarya». Proceedings of the National Academy of Sciences of the United States of America. 87 (12): 4576–9. Bibcode:1990PNAS...87.4576W. PMC 54159
 . PMID 2112744. doi:10.1073/pnas.87.12.4576
. PMID 2112744. doi:10.1073/pnas.87.12.4576
- ↑ Sapp J (2009). The new foundations of evolution: on the tree of life. New York: Oxford University Press. ISBN 978-0-19-973438-2
- ↑ «Archaea». Merriam-Webster Online Dictionary. 2008. Consultado em 1 de julho de 2008
- ↑ Magrum LJ, Luehrsen KR, Woese CR (maio 1978). «Are extreme halophiles actually "bacteria"?». Journal of Molecular Evolution. 11 (1): 1–8. Bibcode:1978JMolE..11....1M. PMID 660662. doi:10.1007/bf01768019
- ↑ Stetter KO (1996). «Hyperthermophiles in the history of life». Ciba Foundation Symposium. 202: 1–10; discussion 11–8. PMID 9243007
- ↑ Dridi, Bédis; Didier Raoult, Michel Drancourt. «Archaea as emerging organisms in complex human microbiomes». Anaerobe. doi:10.1016/j.anaerobe.2011.03.001
- ↑ Theron J, Cloete TE (2000). «Molecular techniques for determining microbial diversity and community structure in natural environments». Critical Reviews in Microbiology. 26 (1): 37–57. PMID 10782339. doi:10.1080/10408410091154174
- ↑ Schmidt TM (setembro 2006). «The maturing of microbial ecology» (PDF). International Microbiology. 9 (3): 217–23. PMID 17061212. Arquivado do original (PDF) em 11 de setembro de 2008
- ↑ Martin, William; Eugene V. Koonin. «A positive definition of prokaryotes». Nature. doi:10.1038/442868c. Consultado em 16 de dezembro de 2016
- ↑ Lozier, R H; R A Bogomolni, and W Stoeckenius. «Bacteriorhodopsin: a light-driven proton pump in Halobacterium Halobium.». Biophysical Journal. doi:10.1016/S0006-3495(75)85875-9
- ↑ Albers, Sonja-Verena; Benjamin H. «The archaeal cell envelope». Nature Reviews Microbiology. 9 (6): 414–426. doi:10.1038/nrmicro2576
- ↑ Jarrell, Ken F; Albers, Sonja-Verena. «The archaellum: an old motility structure with a new name». Trends in Microbiology. doi:10.1016/j.tim.2012.04.007. Consultado em 19 de julho de 2015
- ↑ a b Willey, JM; Sherwood, LM; Woolverton, CJ (2008). «19». "Microbiology" 7ª ed. [S.l.: s.n.] pp. 474–475, excepto nas partes que têm notas.
- ↑ Heimerl T, Flechsler J, Pickl C, Heinz V, Salecker B, Zweck J, Wanner G, Geimer S, Samson RY, Bell SD, Huber H, Wirth R, Wurch L, Podar M, Rachel R (13 Junho 2017). «A Complex Endomembrane System in the Archaeon Ignicoccus hospitalis Tapped by Nanoarchaeum equitans». Frontiers in Microbiology. 8. 1072 páginas. PMC 5468417
 . PMID 28659892. doi:10.3389/fmicb.2017.01072
. PMID 28659892. doi:10.3389/fmicb.2017.01072
- ↑ Jurtshuk P (1996). «Bacterial Metabolism». Medical Microbiology 4th ed. Galveston (TX): University of Texas Medical Branch at Galveston. ISBN 9780963117212
- ↑ Talbert PB, Henikoff S (2010). «Histone variants – ancient wrap artists of the epigenome». Nature Reviews Molecular Cell Biology. 11: 264–275. doi:10.1038/nrm2861
- ↑ Sandman K, Reeve JN (2006). «Archaeal histones and the origin of the histone fold». Curr. Opin. Microbiol. 9: 520–525. doi:10.1016/j.mib.2006.08.003
- ↑ Bell SD, Jackson SP (1 de abril de 2001). «Mechanism and regulation of transcription in archaea». Curr. Opin. Microbiol. 4 (2): 208–13. PMID 11282478. doi:10.1016/S1369-5274(00)00190-9
- ↑ Reeve JN (2003). «Archaeal chromatin and transcription». Mol. Microbiol. 48 (3): 587–98. PMID 12694606
- ↑ Kelman LM, Kelman Z (2003). «Archaea: an archetype for replication initiation studies?». Mol. Microbiol. 48 (3): 605–15. PMID 12694608
- ↑ Phillips G, Chikwana VM, Maxwell A,; et al. (23 de abril de 2010). «Discovery and characterization of an amidinotransferase involved in the modification of archaeal tRNA». J. Biol. Chem. 285 (17): 12706–13. PMC 2857094
 . PMID 20129918. doi:10.1074/jbc.M110.102236
. PMID 20129918. doi:10.1074/jbc.M110.102236
- ↑ Howland JL (2000). The Surprising Archaea: Discovering Another Domain of Life. Oxford: Oxford University Press. pp. 25–30. ISBN 978-0-19-511183-5
- ↑ a b c Cavicchioli R (janeiro 2011). «Archaea--timeline of the third domain». Nature Reviews. Microbiology. 9 (1): 51–61. PMID 21132019. doi:10.1038/nrmicro2482
- ↑ Gupta RS, Shami A (fevereiro 2011). «Molecular signatures for the Crenarchaeota and the Thaumarchaeota». Antonie van Leeuwenhoek. 99 (2): 133–57. PMID 20711675. doi:10.1007/s10482-010-9488-3
- ↑ Gao B, Gupta RS (Março 2007). «Phylogenomic analysis of proteins that are distinctive of Archaea and its main subgroups and the origin of methanogenesis». BMC Genomics. 8. 86 páginas. PMC 1852104
 . PMID 17394648. doi:10.1186/1471-2164-8-86
. PMID 17394648. doi:10.1186/1471-2164-8-86
- ↑ Gupta RS, Naushad S, Baker S (Março 2015). «Phylogenomic analyses and molecular signatures for the class Halobacteria and its two major clades: a proposal for division of the class Halobacteria into an emended order Halobacteriales and two new orders, Haloferacales ord. nov. and Natrialbales ord. nov., containing the novel families Haloferacaceae fam. nov. and Natrialbaceae fam. nov». International Journal of Systematic and Evolutionary Microbiology. 65 (Pt 3): 1050–69. PMID 25428416. doi:10.1099/ijs.0.070136-0

- ↑ Deppenmeier U (2002). The unique biochemistry of methanogenesis. Col: Progress in Nucleic Acid Research and Molecular Biology. 71. [S.l.: s.n.] pp. 223–83. ISBN 978-0-12-540071-8. PMID 12102556. doi:10.1016/s0079-6603(02)71045-3
- ↑ Martin W, Russell MJ (Janeiro 2003). «On the origins of cells: a hypothesis for the evolutionary transitions from abiotic geochemistry to chemoautotrophic prokaryotes, and from prokaryotes to nucleated cells». Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 358 (1429): 59–85. PMC 1693102
 . PMID 12594918. doi:10.1098/rstb.2002.1183
. PMID 12594918. doi:10.1098/rstb.2002.1183
- ↑ Damer B, Deamer D (Abril 2020). «The Hot Spring Hypothesis for an Origin of Life». Astrobiology. 20 (4): 429–452. PMC 7133448
 . PMID 31841362. doi:10.1089/ast.2019.2045
. PMID 31841362. doi:10.1089/ast.2019.2045
- ↑ Woese C. R. & Gupta, R. (1981). «Are archaebacteria merely derived 'prokaryotes'?». Nature. 289 (5793): 95–6. PMID 6161309. doi:10.1038/289095a0
- ↑ Woese C (1998). «The universal ancestor». Proc. Natl. Acad. Sci. U.S.A. 95 (12): 6854–9. PMID 9618502. doi:10.1073/pnas.95.12.6854
- ↑ Gupta RS (2000). «The natural evolutionary relationships among prokaryotes». Crit. Rev. Microbiol. 26 (2): 111–31. PMID 10890353. doi:10.1080/10408410091154219
- ↑ de Queiroz K (maio de 2005). «Ernst Mayr and the modern concept of species». Proceedings of the National Academy of Sciences of the United States of America. 102 (Supplement 1): 6600–6007. Bibcode:2005PNAS..102.6600D. PMC 1131873
 . PMID 15851674. doi:10.1073/pnas.0502030102
. PMID 15851674. doi:10.1073/pnas.0502030102
- ↑ Eppley JM, Tyson GW, Getz WM, Banfield JF (2007). «Genetic exchange across a species boundary in the archaeal genus ferroplasma». Genetics. 177 (1): 407–16. PMC 2013692
 . PMID 17603112. doi:10.1534/genetics.107.072892
. PMID 17603112. doi:10.1534/genetics.107.072892
- ↑ Papke RT, Zhaxybayeva O, Feil EJ, Sommerfeld K, Muise D, Doolittle WF (agosto de 2007). «Searching for species in haloarchaea». Proceedings of the National Academy of Sciences of the United States of America. 104 (35): 14092–97. Bibcode:2007PNAS..10414092P. PMC 1955782
 . PMID 17715057. doi:10.1073/pnas.0706358104
. PMID 17715057. doi:10.1073/pnas.0706358104
- ↑ Kunin V, Goldovsky L, Darzentas N, Ouzounis CA (2005). «The net of life: reconstructing the microbial phylogenetic network». Genome Res. 15 (7): 954–9. PMC 1172039
 . PMID 15965028. doi:10.1101/gr.3666505
. PMID 15965028. doi:10.1101/gr.3666505
- ↑ Robertson CE, Harris JK, Spear JR, Pace NR (dezembro de 2005). «Phylogenetic diversity and ecology of environmental Archaea». Current Opinion in Microbiology. 8 (6): 638–642. PMID 16236543. doi:10.1016/j.mib.2005.10.003
- ↑ Meier-Kolthoff, J.P., Sardà Carbasse, J., Peinado-Olarte, R.L. and Göker, M. (2022). TYGS and LPSN: a database tandem for fast and reliable genome-based classification and nomenclature of prokaryotes. Nucleic Acids Res, 50, D801-D807; DOI: 10.1093/nar/gkab902. Acesso em 6 de julho de 2023).
- ↑ Hugenholtz P (2002). «Exploring prokaryotic diversity in the genomic era». Genome Biol. 3 (2): REVIEWS0003. PMC 139013
 . PMID 11864374. doi:10.1186/gb-2002-3-2-reviews0003
. PMID 11864374. doi:10.1186/gb-2002-3-2-reviews0003
- ↑ Rappé MS, Giovannoni SJ (2003). «The uncultured microbial majority» (PDF). Annual Review of Microbiology. 57: 369–94. PMID 14527284. doi:10.1146/annurev.micro.57.030502.090759. Cópia arquivada (PDF) em 2 de março de 2019
- ↑ Gevers D, Dawyndt P, Vandamme P, Willems A, Vancanneyt M, Swings J, et al. (novembro 2006). «Stepping stones towards a new prokaryotic taxonomy». Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 361 (1475): 1911–16. PMC 1764938
 . PMID 17062410. doi:10.1098/rstb.2006.1915
. PMID 17062410. doi:10.1098/rstb.2006.1915
- ↑ a b Robertson CE, Harris JK, Spear JR, Pace NR (2005). «Phylogenetic diversity and ecology of environmental Archaea». Curr. Opin. Microbiol. 8 (6): 638–42. PMID 16236543. doi:10.1016/j.mib.2005.10.003
- ↑ Huber H, Hohn MJ, Rachel R, Fuchs T, Wimmer VC, Stetter KO (maio 2002). «A new phylum of Archaea represented by a nanosized hyperthermophilic symbiont». Nature. 417 (6884): 63–67. Bibcode:2002Natur.417...63H. PMID 11986665. doi:10.1038/417063a
- ↑ Barns SM, Delwiche CF, Palmer JD, Pace NR (agosto 1996). «Perspectives on archaeal diversity, thermophily and monophyly from environmental rRNA sequences». Proceedings of the National Academy of Sciences of the United States of America. 93 (17): 9188–93. Bibcode:1996PNAS...93.9188B. PMC 38617
 . PMID 8799176. doi:10.1073/pnas.93.17.9188
. PMID 8799176. doi:10.1073/pnas.93.17.9188
- ↑ Elkins JG, Podar M, Graham DE, Makarova KS, Wolf Y, Randau L, et al. (Junho 2008). «A korarchaeal genome reveals insights into the evolution of the Archaea». Proceedings of the National Academy of Sciences of the United States of America. 105 (23): 8102–07. Bibcode:2008PNAS..105.8102E. PMC 2430366
 . PMID 18535141. doi:10.1073/pnas.0801980105
. PMID 18535141. doi:10.1073/pnas.0801980105
- ↑ Baker BJ, Tyson GW, Webb RI, Flanagan J, Hugenholtz P, Allen EE, Banfield JF (dezembro 2006). «Lineages of acidophilic archaea revealed by community genomic analysis». Science. 314 (5807): 1933–35. Bibcode:2006Sci...314.1933B. PMID 17185602. doi:10.1126/science.1132690
- ↑ Baker BJ, Comolli LR, Dick GJ, Hauser LJ, Hyatt D, Dill BD, et al. (maio 2010). «Enigmatic, ultrasmall, uncultivated Archaea». Proceedings of the National Academy of Sciences of the United States of America. 107 (19): 8806–11. Bibcode:2010PNAS..107.8806B. PMC 2889320
 . PMID 20421484. doi:10.1073/pnas.0914470107
. PMID 20421484. doi:10.1073/pnas.0914470107
- ↑ Guy L, Ettema TJ (dezembro 2011). «The archaeal 'TACK' superphylum and the origin of eukaryotes». Trends in Microbiology. 19 (12): 580–87. PMID 22018741. doi:10.1016/j.tim.2011.09.002
- ↑ a b Zaremba-Niedzwiedzka K, Caceres EF, Saw JH, Bäckström D, Juzokaite L, Vancaester E, et al. (janeiro 2017). «Asgard archaea illuminate the origin of eukaryotic cellular complexity» (PDF). Nature. 541 (7637): 353–58. Bibcode:2017Natur.541..353Z. OSTI 1580084. PMID 28077874. doi:10.1038/nature21031
- ↑ Nina Dombrowski, Jun-Hoe Lee, Tom A Williams, Pierre Offre, Anja Spang (2019). Genomic diversity, lifestyles and evolutionary origins of DPANN archaea. Nature.
- ↑ Spang, A.; Saw, J.; Jørgensen, S.; et al. (6 de maio de 2015). «Complex archaea that bridge the gap between prokaryotes and eukaryotes». Nature. 521: 173–179. doi:10.1038/nature14447
- ↑ de Queiroz K (2005). «Ernst Mayr and the modern concept of species». Proc. Natl. Acad. Sci. U.S.A. 102 Suppl 1: 6600–7. PMID 15851674. doi:10.1073/pnas.0502030102
- ↑ Eppley JM, Tyson GW, Getz WM, Banfield JF (2007). «Genetic exchange across a species boundary in the archaeal genus ferroplasma». Genetics. 177 (1): 407–16. PMID 17603112. doi:10.1534/genetics.107.072892
- ↑ Papke RT, Zhaxybayeva O, Feil EJ, Sommerfeld K, Muise D, Doolittle WF (2007). «Searching for species in haloarchaea». Proc. Natl. Acad. Sci. U.S.A. 104 (35): 14092–7. PMID 17715057. doi:10.1073/pnas.0706358104
- ↑ Kunin V, Goldovsky L, Darzentas N, Ouzounis CA (2005). «The net of life: reconstructing the microbial phylogenetic network». Genome Res. 15 (7): 954–9. PMID 15965028. doi:10.1101/gr.3666505
- ↑ Hugenholtz P (2002). «Exploring prokaryotic diversity in the genomic era». Genome Biol. 3 (2): REVIEWS0003. PMID 11864374. doi:10.1186/gb-2002-3-2-reviews0003
- ↑ Rappé MS, Giovannoni SJ (2003). «The uncultured microbial majority». Annu. Rev. Microbiol. 57: 369–94. PMID 14527284. doi:10.1146/annurev.micro.57.030502.090759
- ↑ Oren A, Garrity GM (2021). «Valid publication of the names of forty-two phyla of prokaryotes». Int J Syst Evol Microbiol. 71 (10). 5056 páginas. PMID 34694987. doi:10.1099/ijsem.0.005056

- ↑ a b Williams TA, Szöllősi GJ, Spang A, Foster PG, Heaps SE, Boussau B, et al. (junho de 2017). «Integrative modeling of gene and genome evolution roots the archaeal tree of life». Proceedings of the National Academy of Sciences of the United States of America. 114 (23): E4602–E4611. Bibcode:2017PNAS..114E4602W. PMC 5468678
 . PMID 28533395. doi:10.1073/pnas.1618463114
. PMID 28533395. doi:10.1073/pnas.1618463114
- ↑ a b Castelle CJ, Banfield JF (2018). «Major New Microbial Groups Expand Diversity and Alter our Understanding of the Tree of Life». Cell. 172 (6): 1181–1197. PMID 29522741. doi:10.1016/j.cell.2018.02.016

- ↑ a b «GTDB release 08-RS214». Genome Taxonomy Database. Consultado em 6 de dezembro de 2021
- ↑ a b «ar53_r214.sp_label». Genome Taxonomy Database. Consultado em 10 de maio de 2023
- ↑ a b «Taxon History». Genome Taxonomy Database. Consultado em 6 de dezembro de 2021
- ↑ Seitz KW, Dombrowski N, Eme L, Spang A, Lombard J, Sieber JR, et al. (abril 2019). «Asgard archaea capable of anaerobic hydrocarbon cycling». Nature Communications. 10 (1). 1822 páginas. Bibcode:2019NatCo..10.1822S. PMC 6478937
 . PMID 31015394. doi:10.1038/s41467-019-09364-x
. PMID 31015394. doi:10.1038/s41467-019-09364-x
- ↑ «Age of the Earth». U.S. Geological Survey. 1997. Consultado em 10 de janeiro de 2006. Cópia arquivada em 23 dezembro 2005
- ↑ Dalrymple GB (2001). «The age of the Earth in the twentieth century: a problem (mostly) solved». Special Publications, Geological Society of London. 190 (1): 205–21. Bibcode:2001GSLSP.190..205D. doi:10.1144/GSL.SP.2001.190.01.14
- ↑ Manhesa G, Allègre CJ, Dupréa B, Hamelin B (1980). «Lead isotope study of basic-ultrabasic layered complexes: Speculations about the age of the earth and primitive mantle characteristics». Earth and Planetary Science Letters. 47 (3): 370–82. Bibcode:1980E&PSL..47..370M. doi:10.1016/0012-821X(80)90024-2
- ↑ de Duve C (outubro de 1995). «The Beginnings of Life on Earth». American Scientist. Consultado em 15 janeiro 2014. Cópia arquivada em 6 Junho 2017
- ↑ Timmer J (4 setembro 2012). «3.5 billion year old organic deposits show signs of life». Ars Technica. Consultado em 15 janeiro 2014
- ↑ Ohtomo Y, Kakegawa T, Ishida A, Nagase T, Rosingm MT (8 dezembro 2013). «Evidence for biogenic graphite in early Archaean Isua metasedimentary rocks». Nature Geoscience. 7 (1): 25. Bibcode:2014NatGe...7...25O. doi:10.1038/ngeo2025
- ↑ Borenstein S (13 novembro 2013). «Oldest fossil found: Meet your microbial mom». Associated Press. Consultado em 15 novembro 2013
- ↑ Noffke N, Christian D, Wacey D, Hazen RM (dezembro 2013). «Microbially induced sedimentary structures recording an ancient ecosystem in the ca. 3.48 billion-year-old Dresser Formation, Pilbara, Western Australia». Astrobiology. 13 (12): 1103–24. Bibcode:2013AsBio..13.1103N. PMC 3870916
 . PMID 24205812. doi:10.1089/ast.2013.1030 Erro no estilo Vancouver: wikilink (ajuda)
. PMID 24205812. doi:10.1089/ast.2013.1030 Erro no estilo Vancouver: wikilink (ajuda)
- ↑ Borenstein S (19 outubro 2015). «Hints of life on what was thought to be desolate early Earth». Excite. Yonkers, NY: Mindspark Interactive Network. Associated Press. Consultado em 20 de outubro de 2015
- ↑ Bell EA, Boehnke P, Harrison TM, Mao WL (novembro 2015). «Potentially biogenic carbon preserved in a 4.1 billion-year-old zircon» (PDF). National Academy of Sciences. Proceedings of the National Academy of Sciences of the United States of America. 112 (47): 14518–21. Bibcode:2015PNAS..11214518B. PMC 4664351
 . PMID 26483481. doi:10.1073/pnas.1517557112
. PMID 26483481. doi:10.1073/pnas.1517557112 Erro no estilo Vancouver: wikilink (ajuda)
Erro no estilo Vancouver: wikilink (ajuda)
- ↑ «3.42-billion-year-old fossil threads may be the oldest known archaea microbes». Science News (em inglês). 26 de julho de 2021. Consultado em 27 de julho de 2021
- ↑ Brocks JJ, Logan GA, Buick R, Summons RE (1999). «Archean molecular fossils and the early rise of eukaryotes». Science. 285 (5430): 1033–6. PMID 10446042. doi:10.1126/science.285.5430.1033
- ↑ Schopf JW (Junho 2006). «Fossil evidence of Archaean life». Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 361 (1470): 869–85. PMC 1578735
 . PMID 16754604. doi:10.1098/rstb.2006.1834
. PMID 16754604. doi:10.1098/rstb.2006.1834
- ↑ Chappe B, Albrecht P, Michaelis W (Julho de 1982). «Polar lipids of archaebacteria in sediments and petroleums». Science. 217 (4554): 65–66. Bibcode:1982Sci...217...65C. PMID 17739984. doi:10.1126/science.217.4554.65
- ↑ Woese CR, Gupta R (janeiro 1981). «Are archaebacteria merely derived 'prokaryotes'?». Nature. 289 (5793): 95–96. Bibcode:1981Natur.289...95W. PMID 6161309. doi:10.1038/289095a0
- ↑ a b c Woese C (Junho 1998). «The universal ancestor». Proceedings of the National Academy of Sciences of the United States of America. 95 (12): 6854–59. Bibcode:1998PNAS...95.6854W. PMC 22660
 . PMID 9618502. doi:10.1073/pnas.95.12.6854
. PMID 9618502. doi:10.1073/pnas.95.12.6854
- ↑ a b Kandler OT (agosto 1998). «The early diversification of life and the origin of the three domains: a proposal.». In: Wiegel J, Adams WW. Thermophiles: the keys to molecular evolution and the origin of life. Athens: Taylor and Francis. p. 19-31. ISBN 978-1-4822-7304-5
- ↑ Gupta RS (2000). «The natural evolutionary relationships among prokaryotes». Crit. Rev. Microbiol. 26 (2): 111–31. PMID 10890353. doi:10.1080/10408410091154219
- ↑ Gribaldo S, Brochier-Armanet C (2006). «The origin and evolution of Archaea: a state of the art». Philos. Trans. R. Soc. Lond., B, Biol. Sci. 361 (1470): 1007–22. PMID 16754611. doi:10.1098/rstb.2006.1841. Consultado em 29 de outubro de 2008. Arquivado do original em 4 de junho de 2012
- ↑ Lake JA (janeiro de 1988). «Origin of the eukaryotic nucleus determined by rate-invariant analysis of rRNA sequences». Nature. 331 (6152): 184–6. PMID 3340165. doi:10.1038/331184a0
- ↑ Gouy M, Li WH (maio de 1989). «Phylogenetic analysis based on rRNA sequences supports the archaebacterial rather than the eocyte tree». Nature. 339 (6220): 145–7. PMID 2497353. doi:10.1038/339145a0
- ↑ Yutin N, Makarova KS, Mekhedov SL, Wolf YI, Koonin EV (maio de 2008). «The deep archaeal roots of eukaryotes». Mol. Biol. Evol. 25. 1619 páginas. PMID 18463089. doi:10.1093/molbev/msn108
- ↑ Nelson KE, Clayton RA, Gill SR,; et al. (1999). «Evidence for lateral gene transfer between Archaea and bacteria from genome sequence of Thermotoga maritima». Nature. 399 (6734): 323–9. PMID 10360571. doi:10.1038/20601
- ↑ Lake JA. (1988). «Origin of the eukaryotic nucleus determined by rate-invariant analysis of rRNA sequences». Nature. 331 (6152): 184–6. PMID 3340165. doi:10.1038/331184a0
- ↑ Chappe B, Albrecht P, Michaelis W (julho de 1982). «Polar Lipids of Archaebacteria in Sediments and Petroleums». Science. 217 (4554): 65–66. PMID 17739984. doi:10.1126/science.217.4554.65
- ↑ Hahn, Jürgen; Haug, Pat (1986). «Traces of Archaebacteria in ancient sediments». System Applied Microbiology. 7 (Archaebacteria '85 Proceedings): 178–83
- ↑ Wang M, Yafremava LS, Caetano-Anollés D, Mittenthal JE, Caetano-Anollés G (Novembro 2007). «Reductive evolution of architectural repertoires in proteomes and the birth of the tripartite world». Genome Research. 17 (11): 1572–85. PMC 2045140
 . PMID 17908824. doi:10.1101/gr.6454307
. PMID 17908824. doi:10.1101/gr.6454307
- ↑ Brocks JJ, Logan GA, Buick R, Summons RE (agosto 1999). «Archean molecular fossils and the early rise of eukaryotes». Science. 285 (5430): 1033–36. Bibcode:1999Sci...285.1033B. CiteSeerX 10.1.1.516.9123
 . PMID 10446042. doi:10.1126/science.285.5430.1033
. PMID 10446042. doi:10.1126/science.285.5430.1033
- ↑ Rasmussen B, Fletcher IR, Brocks JJ, Kilburn MR (outubro 2008). «Reassessing the first appearance of eukaryotes and cyanobacteria». Nature. 455 (7216): 1101–4. Bibcode:2008Natur.455.1101R. PMID 18948954. doi:10.1038/nature07381
- ↑ Ciccarelli FD, Doerks T, von Mering C, Creevey CJ, Snel B, Bork P (Março 2006). «Toward automatic reconstruction of a highly resolved tree of life». Science. 311 (5765): 1283–87. Bibcode:2006Sci...311.1283C. CiteSeerX 10.1.1.381.9514
 . PMID 16513982. doi:10.1126/science.1123061
. PMID 16513982. doi:10.1126/science.1123061
- ↑ Koonin EV, Mushegian AR, Galperin MY, Walker DR (Agosto de 1997). «Comparison of archaeal and bacterial genomes: computer analysis of protein sequences predicts novel functions and suggests a chimeric origin for the archaea». Molecular Microbiology. 25 (4): 619–37. PMID 9379893. doi:10.1046/j.1365-2958.1997.4821861.x

- ↑ a b c d e Gupta RS (dezembro 1998). «Protein phylogenies and signature sequences: A reappraisal of evolutionary relationships among archaebacteria, eubacteria, and eukaryotes». Microbiology and Molecular Biology Reviews. 62 (4): 1435–91. PMC 98952
 . PMID 9841678. doi:10.1128/MMBR.62.4.1435-1491.1998
. PMID 9841678. doi:10.1128/MMBR.62.4.1435-1491.1998
- ↑ Koch AL (Abril 2003). «Were Gram-positive rods the first bacteria?». Trends in Microbiology. 11 (4): 166–70. PMID 12706994. doi:10.1016/S0966-842X(03)00063-5
- ↑ a b c Gupta RS (agosto 1998). «What are archaebacteria: life's third domain or monoderm prokaryotes related to gram-positive bacteria? A new proposal for the classification of prokaryotic organisms». Molecular Microbiology. 29 (3): 695–707. PMID 9723910. doi:10.1046/j.1365-2958.1998.00978.x

- ↑ Gogarten JP (novembro 1994). «Which is the most conserved group of proteins? Homology-orthology, paralogy, xenology, and the fusion of independent lineages». Journal of Molecular Evolution. 39 (5): 541–43. Bibcode:1994JMolE..39..541G. PMID 7807544. doi:10.1007/bf00173425
- ↑ Brown JR, Masuchi Y, Robb FT, Doolittle WF (Junho 1994). «Evolutionary relationships of bacterial and archaeal glutamine synthetase genes». Journal of Molecular Evolution. 38 (6): 566–76. Bibcode:1994JMolE..38..566B. PMID 7916055. doi:10.1007/BF00175876
- ↑ Katz LA (setembro 2015). «Recent events dominate interdomain lateral gene transfers between prokaryotes and eukaryotes and, with the exception of endosymbiotic gene transfers, few ancient transfer events persist». Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 370 (1678). 20140324 páginas. PMC 4571564
 . PMID 26323756. doi:10.1098/rstb.2014.0324
. PMID 26323756. doi:10.1098/rstb.2014.0324
- ↑ a b c Gupta RS (2000). «The natural evolutionary relationships among prokaryotes». Critical Reviews in Microbiology. 26 (2): 111–31. CiteSeerX 10.1.1.496.1356
 . PMID 10890353. doi:10.1080/10408410091154219
. PMID 10890353. doi:10.1080/10408410091154219
- ↑ Gupta RS (2005). «Molecular Sequences and the Early History of Life». In: Sapp J. Microbial Phylogeny and Evolution: Concepts and Controversies. New York: Oxford University Press. pp. 160–183
- ↑ Cavalier-Smith T (janeiro 2002). «The neomuran origin of archaebacteria, the negibacterial root of the universal tree and bacterial megaclassification». International Journal of Systematic and Evolutionary Microbiology. 52 (Pt 1): 7–76. PMID 11837318. doi:10.1099/00207713-52-1-7

- ↑ Valas RE, Bourne PE (fevereiro 2011). «The origin of a derived superkingdom: how a gram-positive bacterium crossed the desert to become an archaeon». Biology Direct. 6. 16 páginas. PMC 3056875
 . PMID 21356104. doi:10.1186/1745-6150-6-16
. PMID 21356104. doi:10.1186/1745-6150-6-16
- ↑ Skophammer RG, Herbold CW, Rivera MC, Servin JA, Lake JA (setembro 2006). «Evidence that the root of the tree of life is not within the Archaea». Molecular Biology and Evolution. 23 (9): 1648–51. PMID 16801395. doi:10.1093/molbev/msl046

- ↑ a b Latorre A, Durban A, Moya A, Pereto J (2011). «The role of symbiosis in eukaryotic evolution». In: Gargaud M, López-Garcìa P, Martin H. Origins and Evolution of Life: An astrobiological perspective. Cambridge: Cambridge University Press. pp. 326–339. ISBN 978-0-521-76131-4. Consultado em 27 de agosto de 2017. Cópia arquivada em 24 de março de 2019
- ↑ Eme L, Spang A, Lombard J, Stairs CW, Ettema TJG (novembro 2017). «Archaea and the origin of eukaryotes». Nature Reviews. Microbiology. 15 (12): 711–723. PMID 29123225. doi:10.1038/nrmicro.2017.133
- ↑ Lake JA (janeiro 1988). «Origin of the eukaryotic nucleus determined by rate-invariant analysis of rRNA sequences». Nature. 331 (6152): 184–86. Bibcode:1988Natur.331..184L. PMID 3340165. doi:10.1038/331184a0
- ↑ Nelson KE, Clayton RA, Gill SR, Gwinn ML, Dodson RJ, Haft DH, et al. (Maio 1999). «Evidence for lateral gene transfer between Archaea and bacteria from genome sequence of Thermotoga maritima». Nature. 399 (6734): 323–29. Bibcode:1999Natur.399..323N. PMID 10360571. doi:10.1038/20601
- ↑ Gouy M, Li WH (1989). «Phylogenetic analysis based on rRNA sequences supports the archaebacterial rather than the eocyte tree». Nature. 339 (6220): 145–7. Bibcode:1989Natur.339..145G. PMID 2497353. doi:10.1038/339145a0
- ↑ Yutin N, Makarova KS, Mekhedov SL, Wolf YI, Koonin EV (2008). «The deep archaeal roots of eukaryotes». Mol. Biol. Evol. 25 (8): 1619–30. PMC 2464739
 . PMID 18463089. doi:10.1093/molbev/msn108
. PMID 18463089. doi:10.1093/molbev/msn108
- ↑ Lake JA. (1988). «Origin of the eukaryotic nucleus determined by rate-invariant analysis of rRNA sequences». Nature. 331 (6152): 184–6. Bibcode:1988Natur.331..184L. PMID 3340165. doi:10.1038/331184a0
- ↑ Williams TA, Foster PG, Cox CJ, Embley TM (dezembro 2013). «An archaeal origin of eukaryotes supports only two primary domains of life» (PDF). Nature. 504 (7479): 231–36. Bibcode:2013Natur.504..231W. PMID 24336283. doi:10.1038/nature12779
- ↑ Tom A. Williams; et al. (2012). «A congruent phylogenomic signal places eukaryotes within the Archaea». Proc. R. Soc. (B rspb20121795)
- ↑ L. Guy & T. Ettema (2011). «The archaeal 'TACK' superphylum and the origin of eukaryotes». Sciencedirect. 19 (12): 580–587
- ↑ Zimmer C (6 Maio 2015). «Under the Sea, a Missing Link in the Evolution of Complex Cells». The New York Times. Consultado em 6 Maio 2015
- ↑ Spang A, Saw JH, Jørgensen SL, Zaremba-Niedzwiedzka K, Martijn J, Lind AE, van Eijk R, Schleper C, Guy L, Ettema TJ (Maio 2015). «Complex archaea that bridge the gap between prokaryotes and eukaryotes». Nature. 521 (7551): 173–179. Bibcode:2015Natur.521..173S. PMC 4444528
 . PMID 25945739. doi:10.1038/nature14447
. PMID 25945739. doi:10.1038/nature14447
- ↑ Seitz KW, Lazar CS, Hinrichs KU, Teske AP, Baker BJ (julho 2016). «Genomic reconstruction of a novel, deeply branched sediment archaeal phylum with pathways for acetogenesis and sulfur reduction». The ISME Journal. 10 (7): 1696–705. PMC 4918440
 . PMID 26824177. doi:10.1038/ismej.2015.233
. PMID 26824177. doi:10.1038/ismej.2015.233
- ↑ MacLeod F, Kindler GS, Wong HL, Chen R, Burns BP (2019). «Asgard archaea: Diversity, function, and evolutionary implications in a range of microbiomes». AIMS Microbiology. 5 (1): 48–61. PMC 6646929
 . PMID 31384702. doi:10.3934/microbiol.2019.1.48
. PMID 31384702. doi:10.3934/microbiol.2019.1.48
- ↑ Zimmer C (15 janeiro 2020). «This Strange Microbe May Mark One of Life's Great Leaps - A organism living in ocean muck offers clues to the origins of the complex cells of all animals and plants.». The New York Times. Consultado em 16 janeiro 2020
- ↑ Imachi H, Nobu MK, Nakahara N, Morono Y, Ogawara M, Takaki Y, et al. (janeiro 2020). «Isolation of an archaeon at the prokaryote-eukaryote interface». Nature. 577 (7791): 519–525. Bibcode:2020Natur.577..519I. PMC 7015854
 . PMID 31942073. doi:10.1038/s41586-019-1916-6
. PMID 31942073. doi:10.1038/s41586-019-1916-6
- ↑ Breithaupt H (Novembro 2001). «The hunt for living gold. The search for organisms in extreme environments yields useful enzymes for industry». EMBO Reports. 2 (11): 968–71. PMC 1084137
 . PMID 11713183. doi:10.1093/embo-reports/kve238
. PMID 11713183. doi:10.1093/embo-reports/kve238
- ↑ a b Egorova K, Antranikian G (Dezembro 2005). «Industrial relevance of thermophilic Archaea». Current Opinion in Microbiology. 8 (6): 649–55. PMID 16257257. doi:10.1016/j.mib.2005.10.015
- ↑ Synowiecki J, Grzybowska B, Zdziebło A (2006). «Sources, properties and suitability of new thermostable enzymes in food processing». Critical Reviews in Food Science and Nutrition. 46 (3): 197–205. PMID 16527752. doi:10.1080/10408690590957296
- ↑ Jenney FE, Adams MW (Janeiro 2008). «The impact of extremophiles on structural genomics (and vice versa)». Extremophiles. 12 (1): 39–50. PMID 17563834. doi:10.1007/s00792-007-0087-9
- ↑ Schiraldi C, Giuliano M, De Rosa M (Setembro 2002). «Perspectives on biotechnological applications of archaea». Archaea. 1 (2): 75–86. PMC 2685559
 . PMID 15803645. doi:10.1155/2002/436561
. PMID 15803645. doi:10.1155/2002/436561
- ↑ Norris PR, Burton NP, Foulis NA (Abril 2000). «Acidophiles in bioreactor mineral processing». Extremophiles. 4 (2): 71–76. PMID 10805560. doi:10.1007/s007920050139
- ↑ Shand RF, Leyva KJ (2008). «Archaeal Antimicrobials: An Undiscovered Country». In: Blum P. Archaea: New Models for Prokaryotic Biology. [S.l.]: Caister Academic Press. ISBN 978-1-904455-27-1
Bibliografia[editar | editar código-fonte]
- Howland, John L. (2000). The Surprising Archaea: Discovering Another Domain of Life. Oxford: Oxford University Press. ISBN 0-19-511183-4
- Martinko JM, Madigan MT (2005). Brock Biology of Microorganisms 11th ed. Englewood Cliffs, N.J: Prentice Hall. ISBN 0-13-144329-1
- Garrett RA, Klenk H (2005). Archaea: Evolution, Physiology and Molecular Biology. [S.l.]: WileyBlackwell. ISBN 1-40-514404-1
- Cavicchioli R (2007). Archaea: Molecular and Cellular Biology. [S.l.]: American Society for Microbiology. ISBN 1-55-581391-7
- Blum P (editor) (2008). Archaea: New Models for Prokaryotic Biology. [S.l.]: Caister Academic Press. ISBN 978-1-904455-27-1
- Lipps G (2008). «Archaeal Plasmids». Plasmids: Current Research and Future Trends. [S.l.]: Caister Academic Press. ISBN 978-1-904455-35-6
- Howland JL (2000). The Surprising Archaea: Discovering Another Domain of Life. [S.l.]: Oxford University. ISBN 978-0-19-511183-5
- Martinko JM, Madigan MT (2005). Brock Biology of Microorganisms 11th ed. Englewood Cliffs, N.J: Prentice Hall. ISBN 978-0-13-144329-7
- Garrett RA, Klenk H (2005). Archaea: Evolution, Physiology and Molecular Biology. [S.l.]: WileyBlackwell. ISBN 978-1-4051-4404-9
- Cavicchioli R (2007). Archaea: Molecular and Cellular Biology. [S.l.]: American Society for Microbiology. ISBN 978-1-55581-391-8
- Blum P, ed. (2008). Archaea: New Models for Prokaryotic Biology. [S.l.]: Caister Academic Press. ISBN 978-1-904455-27-1
- Lipps G (2008). «Archaeal Plasmids». Plasmids: Current Research and Future Trends. [S.l.]: Caister Academic Press. ISBN 978-1-904455-35-6
- Sapp J (2009). The New Foundations of Evolution: On the Tree of Life. New York: Oxford University Press. ISBN 978-0-19-538850-3
- Schaechter M (2009). Archaea (Overview) in The Desk Encyclopedia of Microbiology 2nd ed. San Diego and London: Elsevier Academic Press. ISBN 978-0-12-374980-2
Ver também[editar | editar código-fonte]
Ligações externas[editar | editar código-fonte]
- Introduction to the Archaea, ecology, systematics and morphology
- Oceans of Archaea – E.F. DeLong, ASM News, 2003
- NCBI taxonomy page on Archaea
- Genera of the domain Archaea – list of Prokaryotic names with Standing in Nomenclature
- Shotgun sequencing finds nanoorganisms – discovery of the ARMAN group of archaea
- Browse any completed archaeal genome at UCSC
- Comparative Analysis of Archaeal Genomes (at DOE's IMG system)
- (em inglês) Introdução às Archaea, ecologia, sistemática e morfologia
- (em inglês) Oceanos de Archaea[ligação inativa] - E.F. DeLong, ASM News, 2003
- (em inglês) NCBI taxonomy - Archaea
- (em inglês) Géneros do domínio Archaea
- (em inglês) Tree of Life - ilustração de como as Archaea se relacionam com outras formas de vida
- (em inglês) Sequenciação descobre novos nanoorganismos
- (em inglês) Navegue por genomas archaeanos - UCSC
- (em inglês) Análise comparativa de genomas de Archaea



