Proteína: diferenças entre revisões
m Foram revertidas as edições de 201.79.146.71 para a última revisão de Salebot (usando Huggle) |
+expansão; fontes; |
||
| Linha 1: | Linha 1: | ||
{{em construção}} |
|||
{{mais notas|data=junho de 2012| arte=| Brasil=| ciência=| geografia=| música=| Portugal=| sociedade=|1=Este artigo ou se(c)ção|2=|3=|4=|5=|6=}} |
{{mais notas|data=junho de 2012| arte=| Brasil=| ciência=| geografia=| música=| Portugal=| sociedade=|1=Este artigo ou se(c)ção|2=|3=|4=|5=|6=}} |
||
{{portal-bioquímica}} |
|||
[[Ficheiro:Myoglobin.png|thumb|Estrutura 3D da [[mioglobina]], proteína globular de 153 aminoácidos, que contém um grupo prostético heme no centro.]] |
[[Ficheiro:Myoglobin.png|thumb|Estrutura 3D da [[mioglobina]], proteína globular de 153 aminoácidos, que contém um grupo prostético heme no centro.]] |
||
[[Ficheiro:Ligapep2.jpg|thumb|Parte de uma cadeia proteica mostrando as [[ligações peptídicas]].]] |
[[Ficheiro:Ligapep2.jpg|thumb|Parte de uma cadeia proteica mostrando as [[ligações peptídicas]].]] |
||
| Linha 8: | Linha 8: | ||
Uma grande parte das proteínas são completamente sintetizadas no citosol das células pela tradução do [[RNA]] enquanto as proteínas destinadas membrana citoplasmática, lisossomas e as proteínas de secreção possuem um sinal que é reconhecido pela membrana do retículo endoplasmático onde terminam sua síntese. As proteínas são as estruturas, sob o foco da química, de maior complexidade e mais sofisticadas funcionalmente que se conhece.<ref name=albers>{{Referência a livro|autor=Alberts, Bruce; Johnson, Alexander; Lewis, Julian; Raff, Martin; Roberts, Keith; Walter, Peter|título=Biologia Molecular da Célula|subtítulo=|idioma=|edição=5|local=Porto Alegre|editora=Artmed|ano=2010|páginas=45-194|volumes=|volume=|url=|capítulo=2: Química Celular e Biossíntese e 3: Proteínas|isbn=978-85-363-2066-3}}</ref> O corpo humano produz cerca de 100.000 proteínas, com cada proteína tendo algumas centenas de aminoácidos de comprimento.<ref>{{citar livro|autor=Pevzner, Pavel A|título=Computational Molecular Biology|subtítulo=An Algorithmic Approach|editora=MIT Press|local=Cambridge, Massachusetts|idioma=inglês|ano=2000|página=271|páginas=314|isbn=0-262-16197-4}}</ref> |
Uma grande parte das proteínas são completamente sintetizadas no citosol das células pela tradução do [[RNA]] enquanto as proteínas destinadas membrana citoplasmática, lisossomas e as proteínas de secreção possuem um sinal que é reconhecido pela membrana do retículo endoplasmático onde terminam sua síntese. As proteínas são as estruturas, sob o foco da química, de maior complexidade e mais sofisticadas funcionalmente que se conhece.<ref name=albers>{{Referência a livro|autor=Alberts, Bruce; Johnson, Alexander; Lewis, Julian; Raff, Martin; Roberts, Keith; Walter, Peter|título=Biologia Molecular da Célula|subtítulo=|idioma=|edição=5|local=Porto Alegre|editora=Artmed|ano=2010|páginas=45-194|volumes=|volume=|url=|capítulo=2: Química Celular e Biossíntese e 3: Proteínas|isbn=978-85-363-2066-3}}</ref> O corpo humano produz cerca de 100.000 proteínas, com cada proteína tendo algumas centenas de aminoácidos de comprimento.<ref>{{citar livro|autor=Pevzner, Pavel A|título=Computational Molecular Biology|subtítulo=An Algorithmic Approach|editora=MIT Press|local=Cambridge, Massachusetts|idioma=inglês|ano=2000|página=271|páginas=314|isbn=0-262-16197-4}}</ref> |
||
== |
==Bioquímica== |
||
{{ |
{{AP|Bioquímica|Aminoácido|Ligação peptídica}} |
||
[[File:Peptide-Figure-Revised.png|thumb|Estrutura química (em baixo) e estrutura tridimensional (em cima) de uma ligação peptídica entre a [[alanina]] e um aminoácido adjacente.]] |
|||
São [[compostos quaternários]] de [[carbono]] (C), [[hidrogénio]] (H), [[oxigénio]] (O) e [[azoto|nitrogénio]] (N) - também chamado de [[azoto]] em Portugal- às vezes contêm [[enxofre]] (S), como a cisteína. A estrutura geral dos aminoácidos envolve um '''grupo amina''' e um '''grupo carboxilo''', ambos ligados ao carbono α (o primeiro depois do grupo carboxilo). O carbono α também é ligado a um hidrogénio e a uma '''cadeia lateral''', que é representada pela letra R. O grupo R determina a identidade de um aminoácido específico. A fórmula bidimensional mostrada aqui pode transmitir somente parte da estrutura comum dos aminoácidos, porque uma das propriedades mais importantes de tais compostos é a forma tridimensional, ou '''[[estereoquímica]]'''. Os aminoácidos são classificados em polares, não-polares e neutros, dependendo da natureza da cadeia lateral.<ref name=sperelakis>{{citar livro|autor=Sperelakis, Nicholas (editor); Forbes, Michael S. (autor do capítulo); Ferguson, Donald G. (autor do capítulo) |título=Cell Physiology Sourcebook|subtítulo=A Molecular Approach|local=San Diego, California|editora=Academic Press|edição=3ª|idioma=inglês|capítulo=2:Physiological Structure and Functions of Proteins|página=19|páginas=1235|isbn=0-12-656977-0}}</ref> |
|||
A maior parte das proteínas consiste em [[polímero]]s lineares formados a partir de um máximo de 20 <small>L</small>-α-aminoácidos. Todos os [[Aminoácido proteinogénico|aminoácidos proteinogénicos]] têm em comum diversas características estruturais, entre as quais um [[Carbono alfa e beta|carbono alfa]], ao qual estão [[Ligação química|quimicamente ligados]] um grupo de [[amina]]s, um grupo de [[ácido carboxílico]] e uma [[cadeia lateral]] variável. Apenas a [[prolina]] difere desta estrutura básica.{{Harvref|Nelson|2005}} As cadeias laterais dos aminoácidos comuns apresentam uma grande variedade de propriedades e estruturas químicas. É o efeito combinado de todas as cadeias laterais numa proteína que determina a sua estrutura tridimensional e reatividade química.{{Harvref|Gutteridge|2005}} Os aminoácidos numa cadeia de polipeptídios são unidos por [[Ligação peptídica|ligações peptídicas]]. Uma vez unidos na cadeia proteica, cada aminoácido individual é denominado "resíduo", e cada série repetitiva e encadeada de átomos de [[carbono]], [[nitrogénio]] e [[oxigénio]] é denominada "cadeia principal".{{Harvref|Murray|2006|p=19}} |
|||
Existem 20 aminoácidos principais, sendo denominados [[aminoácidos primários]] ou padrão, mas além desses, existem alguns aminoácidos especiais, que só aparecem em alguns tipos de proteínas. Desses 20, nove são ditos [[aminoácido essencial|essenciais]]: isoleucina, leucina, lisina, metionina, fenilalanina, treonina triptofano, valina, histidina e arginina.<ref name=albers /> O organismo humano não é capaz de produzi-los, e por isso é necessária a sua [[ingestão]] através dos [[alimento]]s para evitar a sua deficiência no organismo. Uma cadeia de aminoácidos denomina-se de "[[peptídeo]]", estas podem possuir dois aminoácidos ([[dipeptídeo]]s), três aminoácidos ([[tripeptídeo]]s), quatro aminoácidos ([[tetrapeptídeo]]s), ou muitos aminoácidos ([[polipeptídeo]]s). O termo '''proteína''' é dado quando na composição do polipeptídeo entram centenas ou milhares de aminoácidos. |
|||
A ligação peptídica tem duas formas de [[Ressonância (química)|ressonância]] que contribuem para a formação de uma [[ligação dupla]] e inibem a rotação em torno do seu próprio eixo, pelo que os carbonos alfa são aproximadamente [[coplanar]]es. Os outros dois ângulos [[diedro]]s na ligação peptídica determinam a forma assumida pela cadeia principal.{{Harvref|Murray|2006|p=31}} A extremidade da proteína com um grupo carboxílico livre é denominada [[C-terminal]] ou carboxi-terminal, enquanto que a extremidade com um grupo livre de [[amina]] é denominada [[N-terminal]] ou amino-terminal. Os termos "proteína", "polipetídeo" e "[[peptídeo]]" são ligeiramente ambíguas e o seu significado pode-se sobrepôr. "Proteína" é geralmente usado para nos referirmos à molécula biológica completa na sua forma [[Estrutura biomolecular|terceária]] estável, enquanto que "peptídeo" está geralmente reservado para [[oligómero]]s curtos de aminoácidos, aos quais muitas falta uma estrutura tridimensional estável. No entanto, a diferenla entre ambos não é bem definida e geralmente corresponde a 20-30 resíduos. "Polipetídeo" pode ser referente a qualquer cadeia linear de aminácidos, independentemente do comprimento, mas onde geralmente não existe uma forma terceária.{{Harvref|Lodish|2004}} |
|||
As ligações entre aminoácidos denominam-se ligações peptídicas e estabelecem-se entre o grupo amina e o grupo carboxilo de dois aminoácidos diferentes, com a perda de uma molécula de [[água]].<ref>{{citar livro|autor=Clayden, Jonathan; Greeves, Nick; Warren, Stuart; Wothers, Peter|título=Organic Chemistry|ano=2000|idioma=inglês|local=Oxford|editora=Oxford University Press|página=165|páginas=1536|isbn=0-19850346-6}}</ref> |
|||
==Síntese== |
|||
===Biossíntese=== |
|||
{{AP|Síntese proteica}} |
|||
[[Image:Ribosome mRNA translation en.svg|thumb|Um [[ribossoma]] produz uma proteína usando como molde [[ARN mensageiro]].]] |
|||
[[File:Genetic code.svg|thumb|A sequência de [[ADN]] de um [[gene]] [[Código genético|codifica]] a sequência de aminoácidos de uma proteína.]] |
|||
As proteínas são produzidas a partir de aminoácidos usando informação codificada nos [[gene]]s. Cada proteína tem a sua própria sequência de aminoácidos que é especificada pela sequência de [[nucleótido]]s do gene que codifica a proteína. O [[código genético]] é um grupo de três conjuntos com três nucleótidos cada um, denominados [[codão|codões]]. Cada uma das combinações de três nucleótidos designa um aminoácido. Por exemplo, AUG ([[adenina]]-[[uracilo]]-[[guanina]]) é o código para a [[metionina]]. Uma vez que o [[ADN]] contém quatro nucleótidos, o número total de codões possíveis é de 64. Por este motivo, existe alguma redundância no código genético, havendo alguns aminoácidos que são especificados por mais de um codão.{{Harvref|vanHolde|1996}} Os genes que são codificados no ADN são inicialmente [[Transcrição (genética)|transcritos]] para pré-[[ARN mensageiro]] (ARNm) por proteínas como a [[ARN-polimerase]]. A maior parte dos organismos processa em seguida o pré-ARNm, usando várias formas de [[modificação pós-transcricional]], formando assim o ARNm amadurecido, o qual é então usado como molde para a síntese proteica feita pelo [[ribossoma]]. Nos [[procarionte]]s, o ARNm tanto pode ser utilizado assim que é produzido, como ser ligado a um ribossoma depois de se ter afastado do [[nucleoide]]. Por outro lado, os [[eucarionte]]s produzem ARNm no [[núcleo celular]], o qual é depois translocado através do [[Envoltório nuclear|envelope nuclear]] para o [[citoplasma]], no qual se dá a [[síntese proteica]]. A velocidade de síntese proteica é maior nos procariontes do que nos eucariontes, podendo atingir os 20 aminoácidos por segundo.{{Harvref|Pain|2000}} |
|||
O processo de síntese de uma proteína a partir de um molde de ARNm é denominado [[Tradução (genética)|tradução]]. O ARNm é carregado no ribossoma, no qual são lidos três nucleótidos de cada vez. A leitura é feita fazendo corresponder cada codão com o seu [[ARN transportador|anticodão]] situado numa molécula de [[ARN transportador]] (ARNt), a qual transporta o aminoácido correspondente ao codão por si reconhecido. A enzima [[aminoacil-tRNA sintetase]] carrega as moléculas de ARNt com o aminoácido correto. As proteínas são sempre sintetizadas a partir do N-terminal em direção ao C-terminal.{{Harvref|Van Holde|1996|pp=1002-1042}} |
|||
O tamanho de uma proteína sintetizada pode ser medido através do número de aminoácidos e da sua [[massa molecular]] total, valor que é geralmente expresso em ''daltons'' (Da), sinónimo de [[unidade de massa atómica]]. As proteínas das [[levedura]]s, por exemplo, têm um comprimento de 466 aminoácidos e 53 kDa de massa.{{Harvref|Lodish|2004}} As maiores proteínas conhecidas são as [[titina]]s, com quase 3000 kDA de massa molecular de {{formatnum:27000}} aminoácidos de comprimento.{{Harvref|Fulton|1991}} |
|||
===Síntese química=== |
|||
As proteínas curtas podem também ser sintetizadas quimicamente através de uma série de métodos denominados [[síntese de peptídeos]], os quais têm por base técnicas de [[síntese orgânica]] de elevado rendimento na produção de peptídeos.{{Harvref|Bruckdorfer|2004}} A síntese química permite a introdução de aminoácidos não naturais nas cadeias de peptídeos.{{Harvref|Schwarzer|2005}} Estes métodos são úteis em laboratório, nas éras de [[bioquímica]] e [[biologia celular]], embora não se adequem à produção comercial. A síntese química não é eficiente para polipeptídeos maiores do que 300 aminoácidos, e as proteínas sintetizadas podem não assumir umediatamente a sua estrutura terceária nativa. Grande parte dos métodos de síntese química realizam-se a partir do C-terminal em direção ao N-terminal, ao contrário da reação biológica natural.{{Harvref|Kent|2009}} |
|||
== Estrutura tridimensional == |
== Estrutura tridimensional == |
||
[[File:Chaperonin_1AON.png|thumb|300px|Estrutura cristalina da [[chaperona]]. As chaperonas auxiliam o [[enovelamento de proteínas]].]] |
|||
[[Ficheiro:ProteinStructures.png|esquerda|thumb|Estruturas tridimensionais das proteínas.]] |
|||
[[File:Proteinviews-1tim.png|thumb|300px|Três representações possíveis da estrutura tridimensional da proteína [[triose-fosfato isomerase]]. À esquerda: representação dos átomos, colorida em função do tipo de átomo. Ao centro: representação simplificada que ilustra a conformação da cadeia principal, colorida em função da estrutura secundária. À direita: representação da superfície colorida em função do tipo de resíduo (a vermelho os resíduos ácidos, a azul os resíduos [[Base (química)|base]], a verde os resíduos polares e a branco os resíduos não polares).]] |
|||
As proteínas podem ter 4 tipos de estrutura<ref>{{citar livro|autor=Alphey, Luke|título=DNA Sequencing|subtítulo=From Experimental Methods to Bioinformatics|local=New York|editora=Springer|ano=1997|capítulo=17: Protein Structure Prediction|página=179|idioma=inglês|isbn=0-387-91509-5}}</ref> dependendo do tipo de aminoácidos que possui, do tamanho da cadeia e da configuração espacial da cadeia polipeptídica. As estruturas são: |
As proteínas podem ter 4 tipos de estrutura<ref>{{citar livro|autor=Alphey, Luke|título=DNA Sequencing|subtítulo=From Experimental Methods to Bioinformatics|local=New York|editora=Springer|ano=1997|capítulo=17: Protein Structure Prediction|página=179|idioma=inglês|isbn=0-387-91509-5}}</ref> dependendo do tipo de aminoácidos que possui, do tamanho da cadeia e da configuração espacial da cadeia polipeptídica. As estruturas são: |
||
| Linha 50: | Linha 65: | ||
Um dos principais exemplos de estrutura quaternária é a [[hemoglobina]]. Sua estrutura é formada por quatro cadeias polipeptídicas. |
Um dos principais exemplos de estrutura quaternária é a [[hemoglobina]]. Sua estrutura é formada por quatro cadeias polipeptídicas. |
||
== Aminoácidos == |
|||
{{Ver artigo principal|[[Aminoácido]]s}} |
|||
São [[compostos quaternários]] de [[carbono]] (C), [[hidrogénio]] (H), [[oxigénio]] (O) e [[azoto|nitrogénio]] (N) - também chamado de [[azoto]] em Portugal- às vezes contêm [[enxofre]] (S), como a cisteína. A estrutura geral dos aminoácidos envolve um '''grupo amina''' e um '''grupo carboxilo''', ambos ligados ao carbono α (o primeiro depois do grupo carboxilo). O carbono α também é ligado a um hidrogénio e a uma '''cadeia lateral''', que é representada pela letra R. O grupo R determina a identidade de um aminoácido específico. A fórmula bidimensional mostrada aqui pode transmitir somente parte da estrutura comum dos aminoácidos, porque uma das propriedades mais importantes de tais compostos é a forma tridimensional, ou '''[[estereoquímica]]'''. Os aminoácidos são classificados em polares, não-polares e neutros, dependendo da natureza da cadeia lateral.<ref name=sperelakis>{{citar livro|autor=Sperelakis, Nicholas (editor); Forbes, Michael S. (autor do capítulo); Ferguson, Donald G. (autor do capítulo) |título=Cell Physiology Sourcebook|subtítulo=A Molecular Approach|local=San Diego, California|editora=Academic Press|edição=3ª|idioma=inglês|capítulo=2:Physiological Structure and Functions of Proteins|página=19|páginas=1235|isbn=0-12-656977-0}}</ref> |
|||
Existem 20 aminoácidos principais, sendo denominados [[aminoácidos primários]] ou padrão, mas além desses, existem alguns aminoácidos especiais, que só aparecem em alguns tipos de proteínas. Desses 20, nove são ditos [[aminoácido essencial|essenciais]]: isoleucina, leucina, lisina, metionina, fenilalanina, treonina triptofano, valina, histidina e arginina.<ref name=albers /> O organismo humano não é capaz de produzi-los, e por isso é necessária a sua [[ingestão]] através dos [[alimento]]s para evitar a sua deficiência no organismo. Uma cadeia de aminoácidos denomina-se de "[[peptídeo]]", estas podem possuir dois aminoácidos ([[dipeptídeo]]s), três aminoácidos ([[tripeptídeo]]s), quatro aminoácidos ([[tetrapeptídeo]]s), ou muitos aminoácidos ([[polipeptídeo]]s). O termo '''proteína''' é dado quando na composição do polipeptídeo entram centenas ou milhares de aminoácidos. |
|||
As ligações entre aminoácidos denominam-se ligações peptídicas e estabelecem-se entre o grupo amina e o grupo carboxilo de dois aminoácidos diferentes, com a perda de uma molécula de [[água]].<ref>{{citar livro|autor=Clayden, Jonathan; Greeves, Nick; Warren, Stuart; Wothers, Peter|título=Organic Chemistry|ano=2000|idioma=inglês|local=Oxford|editora=Oxford University Press|página=165|páginas=1536|isbn=0-19850346-6}}</ref> |
|||
== Funções == |
== Funções == |
||
| Linha 102: | Linha 126: | ||
Por exemplo, uma [[ribonuclease]] purificada pode ser desnaturada pela presença de um [[agente redutor]] em solução concentrada de [[ureia]], através da clivagem de quatro de suas oito [[ligação dissulfeto|ligações dissulfureto]] a resíduos de [[cisteína]] e pela desestabilização das [[interação hidrofóbica|interações hidrofóbicas]] causada pela ureia. A desnaturação da ribonuclease é acompanhada por uma completa perda de sua atividade catalítica. Removidos o agente redutor e a ureia, a ribonuclease desnaturada retoma sua estrutura terciária correta, dobrando-se espontaneamente, com restauração completa de sua atividade catalítica. As ligações dissulfureto são refeitas nas mesmas posições anteriores.<ref name="Lehn" /> |
Por exemplo, uma [[ribonuclease]] purificada pode ser desnaturada pela presença de um [[agente redutor]] em solução concentrada de [[ureia]], através da clivagem de quatro de suas oito [[ligação dissulfeto|ligações dissulfureto]] a resíduos de [[cisteína]] e pela desestabilização das [[interação hidrofóbica|interações hidrofóbicas]] causada pela ureia. A desnaturação da ribonuclease é acompanhada por uma completa perda de sua atividade catalítica. Removidos o agente redutor e a ureia, a ribonuclease desnaturada retoma sua estrutura terciária correta, dobrando-se espontaneamente, com restauração completa de sua atividade catalítica. As ligações dissulfureto são refeitas nas mesmas posições anteriores.<ref name="Lehn" /> |
||
==História e etimologia== |
|||
[[File:KendrewMyoglobin.jpg|thumb|John Kendrew com um modelo de [[mioglobina]].]] |
|||
As proteínas foram pela primeira vez descritas pelo químico holandês [[Gerardus Johannes Mulder]] e assim batizadas pelo químico sueco [[Jöns Jacob Berzelius]] em 1838. Mulder levou a cabo análises elementares de proteínas vulgares e constatou que praticamente todas as proteínas apresentavam a mesma [[fórmula empírica]] – C<sub>400</sub>H<sub>620</sub>N<sub>100</sub>O<sub>120</sub>P<sub>1</sub>S<sub>1</sub>. Ainda que erradamente, concluiu que as proteínas deveriam ser constituídas por um único tipo de molécula de grande dimensão.{{Harvref|Perrett|2007}} O termo "proteína" para descrever estas moléculas foi proposto pelo sócio de Mulder, Berzelius. Proteína deriva da palavra [[Língua grega|grega]] πρωτεῖος (''proteios''), a qual significa "na liderança" ou "a que está à frente".{{Harvref|Reynolds|2003}} Mulder prossegui a investigação, identificando produtos da degradação proteica, como o aminoácido [[leucina]], para o qual determinou o peso molecular quase preciso de 131 [[Unidade de massa atómica|Da]].{{Harvref|Perrett|2007}} |
|||
Os cientistas pioneiros no campo da nutrição, como o alemão [[Carl von Voit]], acreditavam que a proteína era o mais importante nutriente na manutenção da estrutura corporal, uma vez que existia a crença generalizada de que seria a carne tinha origem na própria carne.{{Harvref|Bischoff|1860}} [[Karl Heinrich Ritthausen]] alargou o campo das proteínas conhecidas com a identificação do [[ácido glutâmico]]. [[Thomas Burr Osborne]] compilou em 1909 uma revisão detalhada de todas as proteínas vegetais e, no mesmo ano e em conjunto com [[Lafayette Mendel]], determinou os [[aminoácidos essenciais]] à sobrevivência de ratos de laboratório aplicando a [[lei de Liebig]]. A compreensão das proteínas enquanto polipetídeos foi proporcionada por [[Franz Hofmeister]] e [[Hermann Emil Fischer]]. O papel central das proteínas enquanto enzimas nos organismos vivos foi determinado em 1926, quando [[ames Batcheller Sumner]] demonstrou que a [[urease]] era de facto uma proteína.{{Harvref|Sumner|1926}} |
|||
A dificuldade em purificar proteínas em grande quantidade dificultou imenso a investigação dos primeiros bioquímicos. Assim, a investigação inicial focou-se sobretudo em proteínas que podiam ser facilmente purificadas em quantidade, como as do [[sangue]], da [[clara de ovo]], diversas [[toxina]]s e enzimas digestivas obtidas em [[matadouro]]s.{{Harvref|Perrett|2007}} Atribiu-se a [[Linus Pauling]] a primeira previsão bem sucedida de de estruturas secundárias de proteínas com base nas [[Ligação de hidrogénio|ligações de hidrogénio]], uma ideia que já tinha sido proposta em 1933 por [[William Astbury]].{{Harvref|Pauling|1951}} Posteriormente, a investigação de [[Walter Kauzmann]] sobre a desnaturação, baseada em parte nos estudos anteriores de Kaj Ulrik Linderstrøm-Lang, veio a contribuir para a compreensão do [[enovelamento de proteínas]] e das estruturas mediadas por interações hidrófugas.{{Harvref|Kalman|1955}}{{Harvref|Kauzmann|1956}}{{Harvref|Kauzmann|1959}} A primeira proteína a ser [[Sequenciação de proteínas|sequenciada]] foi a [[insulina]], por [[Frederick Sanger]] em 1949. Sanger determinou corretamente a sequência de aminoácidos da proteína, demonstrando de forma conclusiva que as proteínas eram constituídas por polímeros lineares de aminoácidos, em vez de cadeias ramificadas ou [[coloide]]s.{{Harvref|Sanger|1949}} |
|||
As primeiras estruturas proteicas a serem resolvidas foram as da [[hemoglobina]] e da [[mioglobina]], por [[Max Perutz]] e [[John Kendrew]], respetivamente, em 1958.{{Harvref|Muirhead|1963}}{{Harvref|Kendrew|1958}} Nas décadas posteriores, a [[crio-microscopia eletrónica]] de grandes conjuntos macromoleculares e a previsão computacional de estruturas proteicas de pequenos domínios foram métodos que permitiram a investigação de proteínas à escala atómica.{{Harvref|Zhou|2008}}{{Harvref|keskin|2008}} No início de 2014, estavam registadas no ''[[Protein Data Bank]]'' aproximadamente {{formatnum:90000}} estruturas proteicas com resolução atómica.{{cite web |url=http://www.rcsb.org/pdb/home/home.do |title=RCSB Protein Data Bank|accessdate=2013-07-01 |ref=harv}} |
|||
== {{Ver também}} == |
== {{Ver também}} == |
||
| Linha 114: | Linha 149: | ||
{{Referências}} |
{{Referências}} |
||
===Bibliografia=== |
|||
== Referências adicionais== |
|||
{{Div col|2}} |
|||
*{{cite book |author=Bischoff TLW, Voit, C |title=Die Gesetze der Ernaehrung des Pflanzenfressers durch neue Untersuchungen festgestellt |location=Leipzig, Heidelberg |year=1860 |language=German |ref=harv}} |
|||
*{{cite journal |author=Brosnan J |title=Interorgan amino acid transport and its regulation |url=http://jn.nutrition.org/cgi/content/full/133/6/2068S |journal=Journal of Nutrition |volume=133 |issue=6 Suppl 1 |pages=2068S–72S |pmid=12771367 |date=June 2003 |ref=harv}} |
|||
*{{cite journal |author=Bruckdorfer T, Marder O, Albericio F |title=From production of peptides in milligram amounts for research to multi-tons quantities for drugs of the future |journal=Current Pharmaceutical Biotechnology |volume=5 |issue=1 |pages=29–43 |year=2004 |pmid=14965208 |doi=10.2174/1389201043489620 |ref=harv}} |
|||
*{{cite journal |author=Cedrone F, Ménez A, Quéméneur E |title=Tailoring new enzyme functions by rational redesign |journal=Current Opinion in Structural Biology |volume=10 |issue=4 |pages=405–10 |year=2000 |pmid=10981626 |doi=10.1016/S0959-440X(00)00106-8 |ref=harv}} |
|||
*{{cite journal |author=Conrotto P, Souchelnytskyi S |title=Proteomic approaches in biological and medical sciences: principles and applications |journal=Experimental Oncology |volume=30 |issue=3 |pages=171–80 |year=2008 |pmid=18806738 |ref=harv}} |
|||
*{{cite journal |author=Copland JA, Sheffield-Moore M, Koldzic-Zivanovic N, Gentry S, Lamprou G, Tzortzatou-Stathopoulou F, Zoumpourlis V, Urban RJ, Vlahopoulos SA |title=Sex steroid receptors in skeletal differentiation and epithelial neoplasia: is tissue-specific intervention possible? |journal=BioEssays: news and reviews in molecular, cellular and developmental biology |volume=31 |issue=6 |year=2009 |pages=629–41 |doi=10.1002/bies.200800138 |pmid=19382224 |ref=harv}} |
|||
*{{citar periódico|url=http://www.expasy.org/NAR/enz00.pdf |último=Bairoch |primeiro=A |ano=2000 |título=The ENZYME database in 2000 |periódico=Nucleic Acids Research |volume=28 |páginas=304–305 |pmid=10592255 |doi=10.1093/nar/28.1.304 |número=1 |pmc=102465 |ref=harv}} |
|||
*{{cite journal |author=Fernández A, Scott R |title=Dehydron: a structurally encoded signal for protein interaction |journal=Biophysical Journal |volume=85 |issue=3 |pages=1914–28 |year=2003 |pmid=12944304 |pmc=1303363 |doi=10.1016/S0006-3495(03)74619-0 |url=http://linkinghub.elsevier.com/retrieve/pii/S0006-3495(03)74619-0 |bibcode=2003BpJ....85.1914F |ref=harv}} |
|||
*{{cite journal |author=Fulton A, Isaacs W |title=Titin, a huge, elastic sarcomeric protein with a probable role in morphogenesis |journal=BioEssays |volume=13 |issue=4 |pages=157–61 |year=1991 |pmid=1859393 |doi=10.1002/bies.950130403 |ref=harv}} |
|||
*{{cite journal |author=Gonen T, Cheng Y, Sliz P, Hiroaki Y, Fujiyoshi Y, Harrison SC, Walz T |title=Lipid-protein interactions in double-layered two-dimensional AQP0 crystals |journal=Nature |volume=438 |issue=7068 |pages=633–38 |year=2005 |pmid=16319884 |pmc=1350984 |doi=10.1038/nature04321|bibcode = 2005Natur.438..633G |ref=harv }} |
|||
*{{cite journal |author=Görg A, Weiss W, Dunn MJ |title=Current two-dimensional electrophoresis technology for proteomics |journal=Proteomics |volume=4 |issue=12 |pages=3665–85 |year=2004 |pmid=15543535 |doi=10.1002/pmic.200401031 |ref=harv}} |
|||
*{{cite journal |author=Gutteridge A, Thornton JM |title=Understanding nature's catalytic toolkit |journal=Trends in Biochemical Sciences |volume=30 |issue=11 |pages=622–29 |year=2005 |pmid=16214343 |doi=10.1016/j.tibs.2005.09.006 |ref=harv}} |
|||
*{{cite journal |author=Herges T, Wenzel W |title=''In silico'' folding of a three helix protein and characterization of its free-energy landscape in an all-atom force field |journal=Physical Review Letters |volume=94 |issue=1 |page=018101 |year=2005 |pmid=15698135 |doi=10.1103/PhysRevLett.94.018101 |bibcode=2005PhRvL..94a8101H |ref=harv}} |
|||
*{{cite journal |author=Hey J, Posch A, Cohen A, Liu N, Harbers A |title=Fractionation of complex protein mixtures by liquid-phase isoelectric focusing |journal=Methods in Molecular Biology |volume=424 |pages=225–39 |year=2008 |pmid=18369866 |doi=10.1007/978-1-60327-064-9_19 |series=Methods in Molecular Biology |isbn=978-1-58829-722-8 |ref=harv}} |
|||
*{{cite journal |author=Hoffmann M, Wanko M, Strodel P, König PH, Frauenheim T, Schulten K, Thiel W, Tajkhorshid E, Elstner M |title=Color tuning in rhodopsins: the mechanism for the spectral shift between bacteriorhodopsin and sensory rhodopsin II |journal=Journal of the American Chemical Society |volume=128 |issue=33 |pages=10808–18 |year=2006 |pmid=16910676 |doi=10.1021/ja062082i |ref=harv}} |
|||
*{{cite journal |author=Hohsaka T, Sisido M |title=Incorporation of non-natural amino acids into proteins |journal=Current Opinion in Chemical Biology |volume=6 |issue=6 |pages=809–15 |year=2002 |pmid=12470735 |doi=10.1016/S1367-5931(02)00376-9 |ref=harv}} |
|||
*{{cite journal |author=Joos T, Bachmann J |title=Protein microarrays: potentials and limitations |journal=Frontiers in Bioscience |volume=14 |pages=4376–85 |year=2009 |pmid=19273356 |doi=10.2741/3534 |url=http://www.bioscience.org/2009/v14/af/3534/fulltext.htm |issue=14 |ref=harv}} |
|||
*{{cite journal |author=Kalman SM, Linderstrom-Lang K, Ottesen M, Richards FM|title=Degradation of ribonuclease by subtilisin |journal=Biochimica et Biophysica Acta |volume=16 |issue=2 |pages=297–99 |year=1955 |pmid=14363272 |doi=10.1016/0006-3002(55)90224-9 |ref=harv}} |
|||
*{{cite journal |author=Kauzmann W |title=Structural factors in protein denaturation |journal=Journal of Cellular Physiology. Supplement |volume=47 |issue=Suppl 1 |pages=113–31 |year=1956 |pmid=13332017 |doi=10.1002/jcp.1030470410 |ref=harv}} |
|||
*{{cite journal |author=Kauzmann W |title=Some factors in the interpretation of protein denaturation |journal=Advances in Protein Chemistry |volume=14 |pages=1–63 |year=1959 |pmid=14404936 |doi= 10.1016/S0065-3233(08)60608-7 |series=Advances in Protein Chemistry |isbn=978-0-12-034214-3 |ref=harv}} |
|||
*{{cite journal |author=Kendrew J, Bodo G, Dintzis H, Parrish R, Wyckoff H, Phillips D |title=A three-dimensional model of the myoglobin molecule obtained by X-ray analysis |journal=Nature |volume=181 |issue=4610 |pages=662–66 |year=1958 |pmid=13517261 |doi=10.1038/181662a0 |bibcode=1958Natur.181..662K |ref=harv}} |
|||
*{{cite journal |author=Kent SB |title=Total chemical synthesis of proteins |journal=Chemical Society Reviews |volume=38 |issue=2 |pages=338–51 |year=2009 |pmid=19169452 |doi=10.1039/b700141j |ref=harv}} |
|||
*{{cite journal |author=Keskin O, Tuncbag N, Gursoy A |title=Characterization and prediction of protein interfaces to infer protein-protein interaction networks |journal=Current Pharmaceutical Biotechnology |volume=9 |issue=2 |pages=67–76 |year=2008 |pmid=18393863 |doi=10.2174/138920108783955191 |ref=harv}} |
|||
*{{cite journal |author=Koegl M, Uetz P |title=Improving yeast two-hybrid screening systems |journal=Briefings in Functional Genomics & Proteomics |volume=6 |issue=4 |pages=302–12 |year=2007 |pmid=18218650 |doi=10.1093/bfgp/elm035 |url=http://bfgp.oxfordjournals.org/cgi/pmidlookup?view=long&pmid=18218650 |ref=harv}} |
|||
*{{cite journal |author=Kuhlman B, Dantas G, Ireton GC, Varani G, Stoddard BL, Baker D |title=Design of a novel globular protein fold with atomic-level accuracy |journal=Science |volume=302 |issue=5649 |pages=1364–68 |year=2003 |pmid=14631033 |doi=10.1126/science.1089427 |url=http://www.sciencemag.org/cgi/pmidlookup?view=long&pmid=14631033 |bibcode=2003Sci...302.1364K |ref=harv}} |
|||
*{{cite book |author=Lodish H, Berk A, Matsudaira P, Kaiser CA, Krieger M, Scott MP, Zipurksy SL, Darnell J |year=2004 |title=Molecular Cell Biology |edition=5th |publisher=WH Freeman and Company |location=New York, New York |ref=harv}} |
|||
*{{cite journal |author=Margolin W|title=Green fluorescent protein as a reporter for macromolecular localization in bacterial cells |journal=Methods (San Diego, Calif.) |volume=20 |issue=1 |pages=62–72 |year=2000 |pmid=10610805 |doi=10.1006/meth.1999.0906 |url=http://linkinghub.elsevier.com/retrieve/pii/S1046-2023(99)90906-4 |ref=harv}} |
|||
*{{cite journal |author=Mayhew TM, Lucocq JM |title=Developments in cell biology for quantitative immunoelectron microscopy based on thin sections: a review |journal=Histochemistry and Cell Biology |volume=130 |issue=2 |pages=299–313 |year=2008 |pmid=18553098 |pmc=2491712 |doi=10.1007/s00418-008-0451-6 |ref=harv}} |
|||
*{{cite journal |author=Muirhead H, Perutz M |title=Structure of hemoglobin. A three-dimensional fourier synthesis of reduced human hemoglobin at 5.5 Å resolution |journal=Nature |volume=199 |issue=4894 |pages=633–38 |year=1963 |pmid=14074546 |doi=10.1038/199633a0|bibcode = 1963Natur.199..633M |ref=harv}} |
|||
*{{cite book |author=Nelson DL, Cox MM |year=2005 |title=Lehninger's Principles of Biochemistry |edition=4th |publisher=W. H. Freeman and Company |location=New York, New York |ref=harv}} |
|||
*{{cite book |editor=Pain RH (ed.) |author=Dobson CM |title=Mechanisms of Protein Folding |chapter=The nature and significance of protein folding |publisher=Oxford University Press |location=Oxford, Oxfordshire |year=2000 |pages=1–28 |isbn=0-19-963789-X |ref=harv}} |
|||
*{{cite journal |author=Pauling L, Corey RB, Branson HR |year=1951 |title=The structure of proteins: two hydrogen-bonded helical configurations of the polypeptide chain |journal=Proceedings of the National Academy of Sciences U.S.A. |volume=37 |issue=5 |pages=235–40 |url=http://www.pnas.org/site/misc/Protein8.pdf |format=PDF |doi=10.1073/pnas.37.5.235 |pmc=1063348 |pmid=14834145 |bibcode=1951PNAS...37..235P |ref=harv}} |
|||
*{{cite journal |author=Perrett D |year=2007 |title=From 'protein' to the beginnings of clinical proteomics |journal=Proteomics – Clinical Applications |pmid=21136729 |volume=1 |issue=8 |pages=720–38 |doi=10.1002/prca.200700525 |ref=harv}} |
|||
*{{cite journal |author=Plewczyński D, Ginalski K |title=The interactome: predicting the protein–protein interactions in cells |journal=Cellular & Molecular Biology Letters |volume=14 |issue=1 |pages=1–22 |year=2009 |pmid=18839074 |doi=10.2478/s11658-008-0024-7 |ref=harv}} |
|||
*{{cite journal |author=Radzicka A, Wolfenden R |year=1995 |title=A proficient enzyme |journal=Science |volume=267 |issue=5194 |pages=90–93| pmid=7809611 |doi=10.1126/science.7809611|bibcode = 1995Sci...267...90R |ref=harv}} |
|||
*{{cite book |author=Reynolds JA, Tanford C |title=Nature's Robots: A History of Proteins (Oxford Paperbacks) |publisher=Oxford University Press |location=New York, New York |year=2003 |page=15 |isbn=0-19-860694-X |ref=harv}} |
|||
*{{cite journal |author=Ritchie DW |title=Recent progress and future directions in protein–protein docking |journal=Current Protein and Peptide Science |volume=9 |issue=1 |pages=1–15 |year=2008 |pmid=18336319 |doi=10.2174/138920308783565741 |ref=harv}} |
|||
*{{cite journal |author=Rüdiger H, Siebert HC, Solís D, Jiménez-Barbero J, Romero A, von der Lieth CW, Diaz-Mariño T, Gabius HJ |title=Medicinal chemistry based on the sugar code: fundamentals of lectinology and experimental strategies with lectins as targets |journal=Current Medicinal Chemistry |volume=7 |issue=4 |pages=389–416 |year=2000 |pmid=10702616 |doi=10.2174/0929867003375164 |ref=harv}} |
|||
*{{cite journal |pmid=19273120 |author=Samarin S, Nusrat A |title=Regulation of epithelial apical junctional complex by Rho family GTPases |journal=Frontiers in bioscience: a journal and virtual library |volume=14 |year=2009 |pages=1129–42 |doi=10.2741/3298 |issue=14 |ref=harv}} |
|||
*{{cite journal |author=Sanger F |title=The terminal peptides of insulin |journal=Biochemical Journal |volume=45 |issue=5 |pages=563–74 |year=1949 |pmid=15396627 |pmc=1275055 |ref=harv}} |
|||
*{{cite journal |author=Sankaranarayanan R, Moras D |title=The fidelity of the translation of the genetic code |journal=Acta Biochimica Polonica |volume=48 |issue=2 |pages=323–35 |year=2001 |pmid=11732604 |ref=harv}} |
|||
*{{cite journal |author=Scheraga HA, Khalili M, Liwo A |title=Protein-folding dynamics: overview of molecular simulation techniques |journal=Annual Review of Physical Chemistry |volume=58 |pages=57–83 |year=2007 |pmid=17034338 |doi=10.1146/annurev.physchem.58.032806.104614|bibcode = 2007ARPC...58...57S |ref=harv }} |
|||
*{{cite journal |author=Schwarzer D, Cole P |title=Protein semisynthesis and expressed protein ligation: chasing a protein's tail |journal=Current Opinion in Chemical Biology |volume=9 |issue=6 |pages=561–69 |year=2005 |pmid=16226484 |doi=10.1016/j.cbpa.2005.09.018 |ref=harv}} |
|||
*{{cite journal |author=Sleator RD. |title=Prediction of protein functions |journal=Methods in Molecular Biology |year=2012 |volume=815 |pages=15–24 |doi=10.1007/978-1-61779-424-7_2 |series=Methods in Molecular Biology |isbn=978-1-61779-423-0 |pmid=22130980 |ref=harv}} |
|||
*{{cite journal |author=Standley DM, Kinjo AR, Kinoshita K, Nakamura H |title=Protein structure databases with new web services for structural biology and biomedical research |journal=Briefings in Bioinformatics |volume=9 |issue=4 |pages=276–85 |year=2008 |pmid=18430752 |doi=10.1093/bib/bbn015 |url=http://bib.oxfordjournals.org/cgi/pmidlookup?view=long&pmid=18430752 |ref=harv}} |
|||
*{{cite journal |author=Stepanenko OV, Verkhusha VV, Kuznetsova IM, Uversky VN, Turoverov KK |title=Fluorescent proteins as biomarkers and biosensors: throwing color lights on molecular and cellular processes |journal=Current Protein & Peptide Science |volume=9 |issue=4 |pages=338–69 |year=2008 |pmid=18691124 |pmc=2904242 |doi=10.2174/138920308785132668 |ref=harv}} |
|||
*{{cite journal |author=Sumner JB |title=The isolation and crystallization of the enzyme urease. Preliminary paper |url=http://www.jbc.org/content/69/2/435.full.pdf+html |journal=Journal of Biological Chemistry |volume=69 |pages=435–41 |year=1926 |format=PDF |issue=2 |ref=harv}} |
|||
*{{cite journal |author=Terpe K |title=Overview of tag protein fusions: from molecular and biochemical fundamentals to commercial systems |journal=Applied Microbiology and Biotechnology |volume=60 |issue=5 |pages=523–33 |year=2003 |pmid=12536251 |doi=10.1007/s00253-002-1158-6 |ref=harv}} |
|||
*{{citar web |autor=EBI |url=http://www.ebi.ac.uk/thornton-srv/databases/CSA/ |título=The Catalytic Site Atlas at The European Bioinformatics Institute |editora=Ebi.ac.uk |data=20 de janeiro de 2010 |accessodata=16 de janeiro de 2011 |ref=harv}} |
|||
*{{cite journal |author=Walian P, Cross TA, Jap BK |title=Structural genomics of membrane proteins |journal=Genome Biology |volume=5 |issue=4 |page=215 |year=2004 |pmid=15059248 |pmc=395774 |doi=10.1186/gb-2004-5-4-215 |url=http://genomebiology.com/2004/5/4/215 |ref=harv}} |
|||
*{{cite book |author=Walker JH, Wilson K |title=Principles and Techniques of Practical Biochemistry |publisher=Cambridge University Press |location=Cambridge, UK |year=2000 |pages=287–89 |isbn=0-521-65873-X |ref=harv}} |
|||
*{{cite journal |author=Xiang Z|title=Advances in homology protein structure modeling |journal=Current Protein and Peptide Science |volume=7 |issue=3 |pages=217–27 |year=2006 |pmid=16787261 |pmc=1839925 |doi=10.2174/138920306777452312 |ref=harv}} |
|||
*{{cite journal |author=Yuste R |title=Fluorescence microscopy today |journal=Nature Methods |volume=2 |issue=12 |pages=902–904 |year=2005 |pmid=16299474 |doi=10.1038/nmeth1205-902 |ref=harv}} |
|||
*{{cite journal |author=Zagrovic B, Snow CD, Shirts MR, Pande VS |title=Simulation of folding of a small alpha-helical protein in atomistic detail using worldwide-distributed computing |journal=Journal of Molecular Biology |volume=323 |issue=5 |pages=927–37 |year=2002 |pmid=12417204 |doi=10.1016/S0022-2836(02)00997-X |ref=harv}} |
|||
*{{cite journal |author=Zhang C, Kim SH |title=Overview of structural genomics: from structure to function |journal=Current Opinion in Chemical Biology |volume=7 |issue=1 |pages=28–32 |year=2003 |pmid=12547423 |doi=10.1016/S1367-5931(02)00015-7 |ref=harv}} |
|||
*{{cite journal |author=Zhang Y, Skolnick J |title=The protein structure prediction problem could be solved using the current PDB library |journal=Proceedings of the National Academy of Sciences U.S.A. |volume=102 |issue=4 |pages=1029–34 |year=2005 |pmid=15653774 |pmc=545829 |doi=10.1073/pnas.0407152101 |url=http://www.pnas.org/cgi/pmidlookup?view=long&pmid=15653774|bibcode = 2005PNAS..102.1029Z |ref=harv }} |
|||
*{{cite journal |author=Zhang Y |title=Progress and challenges in protein structure prediction |journal=Current Opinion in Structural Biology |volume=18 |issue=3 |pages=342–48 |year=2008 |doi=10.1016/j.sbi.2008.02.004 |pmid=18436442 |pmc=2680823 |ref=harv}} |
|||
*{{cite journal |author=Zhou ZH |title=Towards atomic resolution structural determination by single-particle cryo-electron microscopy |journal=Current Opinion in Structural Biology |volume=18 |issue=2 |pages=218–28 |year=2008 |pmid=18403197 |doi=10.1016/j.sbi.2008.03.004 |pmc=2714865 |ref=harv}} |
|||
*{{citar livro |ultimo= Branden |primeiro=C |coautor=Tooze, J |título=Introduction to Protein Structure |editora=Garland Pub |local=Nova Iorque |ano=1999 |isbn=0-8153-2305-0 |ref=harv}} |
|||
*{{Citar livro |ultimo=Murray |primeiro=RF |autor2=Harper HW |autor3=Granner DK |autor4=Mayes PA |autor5=Rodwell VW |título=Harper's Illustrated Biochemistry |editora=Lange Medical Books/McGraw-Hill |local=Nova Iorque |ano=2006 |isbn=0-07-146197-3 |ref=harv}} |
|||
*{{Citar livro |ultimo=Van Holde |primeiro=KE |autor2= Mathews, CK | título=Biochemistry |editora=Benjamin/Cummings Pub. Co., Inc |local=Menlo Park, California |ano=1996 |isbn=0-8053-3931-0 |ref=harv}} |
|||
*{{Citar livro |ultimo=Voet |primeiro=D |coautor=Voet JG |ano=2004 |título=Biochemistry |volume=1 |edição=3ª |editora=Wiley |local=Hoboken |url=http://books.google.pt/books/about/Biochemistry.html?id=-GwUPP2C8ysC&redir_esc=y |isbn=978-0470917459 |ref=harv}}</ref> |
|||
* Lubert Stryer, Jeremy Mark Berg, John L. Tymoczko (trad. Serge Weinman), ''Biochimie'', Flammarion, « Médecine-Sciences », Paris, 2003, 5ª éd. {{ISBN|2-257-17116-0}}. |
* Lubert Stryer, Jeremy Mark Berg, John L. Tymoczko (trad. Serge Weinman), ''Biochimie'', Flammarion, « Médecine-Sciences », Paris, 2003, 5ª éd. {{ISBN|2-257-17116-0}}. |
||
* Carl-Ivar Brändén, John Tooze (trad. Bernard Lubochinsky, préf. Joël Janin), ''Introduction à la structure des protéines'', De Boeck Université, Bruxelles, 1996 {{ISBN|2-804-12109-7}}. |
* Carl-Ivar Brändén, John Tooze (trad. Bernard Lubochinsky, préf. Joël Janin), ''Introduction à la structure des protéines'', De Boeck Université, Bruxelles, 1996 {{ISBN|2-804-12109-7}}. |
||
* [http://depa.pquim.unam.mx/proteínas/estructura/index.html Rodríguez, Faride. ''La estructura de las proteínas''] |
* [http://depa.pquim.unam.mx/proteínas/estructura/index.html Rodríguez, Faride. ''La estructura de las proteínas''] |
||
* Lehninger, Albert L. ''Princípios da bioquímica'' |
* Lehninger, Albert L. ''Princípios da bioquímica'' |
||
{{Div col fim}} |
|||
== |
== Ligações externas == |
||
{{Correlatos |
{{Correlatos |
||
|commons = |
|commons = |
||
| Linha 145: | Linha 306: | ||
{{Domínios proteicos}} |
{{Domínios proteicos}} |
||
{{Aminoácidos}} |
{{Aminoácidos}} |
||
{{Portal3|Bioquímica}} |
|||
[[Categoria:Proteínas| ]] |
[[Categoria:Proteínas| ]] |
||
Revisão das 01h38min de 19 de fevereiro de 2014
Este artigo ou seção está em processo de expansão ou reestruturação durante um curto período. |


As Proteínas são compostos orgânicos bioquímicos, constituídos por um ou mais polipeptídeos tipicamente dobrada em uma forma globular ou fibrosa, facilitando uma função biológica. São compostos de alto peso molecular, compostos orgânicos de estrutura complementar e massa molecular elevada (de 100.000 a 100.000.000.000 ou mais unidades de massa atômica), sintetizadas pelos organismos vivos através da condensação de um grande número de moléculas de alfa-aminoácidos, através de ligações denominadas ligações peptídicas.[1] São consideradas as macromoléculas mais importantes das células e, para muitos organismos, constituem quase 50% de suas massas.[2] Uma proteína é um conjunto de no minimo 20 [3] aminoácidos, mas sabemos que uma proteína possui muito mais que essa quantidade, sendo os conjuntos menores denominados Polipeptídeos. Em comparação, designa-se Prótido qualquer composto nitrogenado que contém aminoácidos, peptídios e proteínas (pode conter outros componentes). Uma grande parte das proteínas são completamente sintetizadas no citosol das células pela tradução do RNA enquanto as proteínas destinadas membrana citoplasmática, lisossomas e as proteínas de secreção possuem um sinal que é reconhecido pela membrana do retículo endoplasmático onde terminam sua síntese. As proteínas são as estruturas, sob o foco da química, de maior complexidade e mais sofisticadas funcionalmente que se conhece.[4] O corpo humano produz cerca de 100.000 proteínas, com cada proteína tendo algumas centenas de aminoácidos de comprimento.[5]
Bioquímica

A maior parte das proteínas consiste em polímeros lineares formados a partir de um máximo de 20 L-α-aminoácidos. Todos os aminoácidos proteinogénicos têm em comum diversas características estruturais, entre as quais um carbono alfa, ao qual estão quimicamente ligados um grupo de aminas, um grupo de ácido carboxílico e uma cadeia lateral variável. Apenas a prolina difere desta estrutura básica.[6] As cadeias laterais dos aminoácidos comuns apresentam uma grande variedade de propriedades e estruturas químicas. É o efeito combinado de todas as cadeias laterais numa proteína que determina a sua estrutura tridimensional e reatividade química.[7] Os aminoácidos numa cadeia de polipeptídios são unidos por ligações peptídicas. Uma vez unidos na cadeia proteica, cada aminoácido individual é denominado "resíduo", e cada série repetitiva e encadeada de átomos de carbono, nitrogénio e oxigénio é denominada "cadeia principal".[8]
A ligação peptídica tem duas formas de ressonância que contribuem para a formação de uma ligação dupla e inibem a rotação em torno do seu próprio eixo, pelo que os carbonos alfa são aproximadamente coplanares. Os outros dois ângulos diedros na ligação peptídica determinam a forma assumida pela cadeia principal.[9] A extremidade da proteína com um grupo carboxílico livre é denominada C-terminal ou carboxi-terminal, enquanto que a extremidade com um grupo livre de amina é denominada N-terminal ou amino-terminal. Os termos "proteína", "polipetídeo" e "peptídeo" são ligeiramente ambíguas e o seu significado pode-se sobrepôr. "Proteína" é geralmente usado para nos referirmos à molécula biológica completa na sua forma terceária estável, enquanto que "peptídeo" está geralmente reservado para oligómeros curtos de aminoácidos, aos quais muitas falta uma estrutura tridimensional estável. No entanto, a diferenla entre ambos não é bem definida e geralmente corresponde a 20-30 resíduos. "Polipetídeo" pode ser referente a qualquer cadeia linear de aminácidos, independentemente do comprimento, mas onde geralmente não existe uma forma terceária.[10]
Síntese
Biossíntese


As proteínas são produzidas a partir de aminoácidos usando informação codificada nos genes. Cada proteína tem a sua própria sequência de aminoácidos que é especificada pela sequência de nucleótidos do gene que codifica a proteína. O código genético é um grupo de três conjuntos com três nucleótidos cada um, denominados codões. Cada uma das combinações de três nucleótidos designa um aminoácido. Por exemplo, AUG (adenina-uracilo-guanina) é o código para a metionina. Uma vez que o ADN contém quatro nucleótidos, o número total de codões possíveis é de 64. Por este motivo, existe alguma redundância no código genético, havendo alguns aminoácidos que são especificados por mais de um codão.[11] Os genes que são codificados no ADN são inicialmente transcritos para pré-ARN mensageiro (ARNm) por proteínas como a ARN-polimerase. A maior parte dos organismos processa em seguida o pré-ARNm, usando várias formas de modificação pós-transcricional, formando assim o ARNm amadurecido, o qual é então usado como molde para a síntese proteica feita pelo ribossoma. Nos procariontes, o ARNm tanto pode ser utilizado assim que é produzido, como ser ligado a um ribossoma depois de se ter afastado do nucleoide. Por outro lado, os eucariontes produzem ARNm no núcleo celular, o qual é depois translocado através do envelope nuclear para o citoplasma, no qual se dá a síntese proteica. A velocidade de síntese proteica é maior nos procariontes do que nos eucariontes, podendo atingir os 20 aminoácidos por segundo.[12]
O processo de síntese de uma proteína a partir de um molde de ARNm é denominado tradução. O ARNm é carregado no ribossoma, no qual são lidos três nucleótidos de cada vez. A leitura é feita fazendo corresponder cada codão com o seu anticodão situado numa molécula de ARN transportador (ARNt), a qual transporta o aminoácido correspondente ao codão por si reconhecido. A enzima aminoacil-tRNA sintetase carrega as moléculas de ARNt com o aminoácido correto. As proteínas são sempre sintetizadas a partir do N-terminal em direção ao C-terminal.[13]
O tamanho de uma proteína sintetizada pode ser medido através do número de aminoácidos e da sua massa molecular total, valor que é geralmente expresso em daltons (Da), sinónimo de unidade de massa atómica. As proteínas das leveduras, por exemplo, têm um comprimento de 466 aminoácidos e 53 kDa de massa.[10] As maiores proteínas conhecidas são as titinas, com quase 3000 kDA de massa molecular de 27 000 aminoácidos de comprimento.[14]
Síntese química
As proteínas curtas podem também ser sintetizadas quimicamente através de uma série de métodos denominados síntese de peptídeos, os quais têm por base técnicas de síntese orgânica de elevado rendimento na produção de peptídeos.[15] A síntese química permite a introdução de aminoácidos não naturais nas cadeias de peptídeos.[16] Estes métodos são úteis em laboratório, nas éras de bioquímica e biologia celular, embora não se adequem à produção comercial. A síntese química não é eficiente para polipeptídeos maiores do que 300 aminoácidos, e as proteínas sintetizadas podem não assumir umediatamente a sua estrutura terceária nativa. Grande parte dos métodos de síntese química realizam-se a partir do C-terminal em direção ao N-terminal, ao contrário da reação biológica natural.[17]
Estrutura tridimensional


As proteínas podem ter 4 tipos de estrutura[18] dependendo do tipo de aminoácidos que possui, do tamanho da cadeia e da configuração espacial da cadeia polipeptídica. As estruturas são:
Estrutura primária
É dada pela sequência de aminoácidos ao longo da cadeia polipeptídica.[19] É o nível estrutural mais simples e mais importante, pois dele deriva todo o arranjo espacial da molécula. São específicas para cada proteína, sendo, geralmente, determinadas geneticamente. A estrutura primária da proteína resulta em uma longa cadeia de aminoácidos, com uma extremidade "amino terminal" e uma extremidade "carboxi terminal".[20] Sua estrutura é somente a sequência dos aminoácidos, sem se preocupar com a orientação espacial da molécula. Suas ligações são ligações peptídicas e ligações dissulfureto.
Estrutura secundária
É dada pelo arranjo espacial de aminoácidos próximos entre si na sequência primária da proteína.[19]
Ocorre graças à possibilidade de rotação das ligações entre os carbonos alfa dos aminoácidos e os seus grupos amina e carboxilo.[4] O arranjo secundário de uma cadeia polipeptídica pode ocorrer de forma regular; isso acontece quando os ângulos das ligações entre carbonos alfa e seus ligantes são iguais e se repetem ao longo de um segmento da molécula.
A cadeia polipeptídica pode interagir consigo mesma de dois modos principais: pela formação das alfa-hélices e das folhas-beta. Além destas existem estruturas que não são nem hélices nem folhas chamadas laços (loops).[21]
- alfa-hélice: presente na estrutura secundária dos níveis de organização das proteínas. São estruturas cilindricas estabilizadas por pontes de hidrogénio entre aminoácidos. As cadeias laterais dos aminoácidos encontram-se viradas para fora. Existem várias formas de como as hélice alfa podem organizar-se. Na alfa-hélice a espinha dorsal polipeptídica é torcida em uma hélice virada à direita.[20]
- folha-beta: padrão estrutural encontrado em várias proteínas, nas quais regiões vizinhas da cadeia polipeptídica associam-se por meio de ligações de hidrogénio, resultando em uma estrutura achatada e rígida. Esta é também uma estrutura estável na qual os grupos polares da cadeia polipeptídica associam-se por meio de ligações de hidrogénio um ao outro.[20]
- laços: Laços são secções da sequência que se ligam aos outros dois tipos de estrutura secundária. Em contraste com hélices e folhas, que formam o núcleo da proteína, loops não são estruturas regulares e ficam fora da proteína dobrada.[21]
Estrutura terciária
Resulta do enrolamento da hélice ou da folha pregueada, sendo estabilizada por pontes de hidrogênio e ligações dissulfureto. Esta estrutura confere a atividade biológica às proteicas. Ela descreve a conformação da proteína inteira.[19]
A estrutura terciária descreve o dobramento final de uma cadeia, por interações de regiões com estrutura regular ou de regiões sem estrutura definida, podendo haver interações de segmentos distantes de estrutura primaria, por ligações não covalentes.
Enquanto a estrutura secundária é determinada pelo relacionamento estrutural de curta distância, a terciária é caracterizada pelas interações de longa distância entre aminoácidos, denominadas interações hidrofóbicas, pelas interações eletrostáticas, pontes de hidrogênio e dissulfeto. Todas têm seqüências de aminoácidos diferentes, refletindo estruturas e funções diferentes. Efetua interações locais entre os aminoácidos que ficam próximos uns dos outros.
Estrutura quaternária
Algumas proteínas podem ter duas ou mais cadeias polipeptídicas. Essa transformação das proteínas em estruturas tridimensionais é a estrutura quaternária,[19] que são guiadas e estabilizadas pelas mesmas interações da terciária.A junção de cadeias polipeptídicas pode produzir diferentes funções para os compostos.
Um dos principais exemplos de estrutura quaternária é a hemoglobina. Sua estrutura é formada por quatro cadeias polipeptídicas.
Aminoácidos
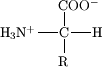
São compostos quaternários de carbono (C), hidrogénio (H), oxigénio (O) e nitrogénio (N) - também chamado de azoto em Portugal- às vezes contêm enxofre (S), como a cisteína. A estrutura geral dos aminoácidos envolve um grupo amina e um grupo carboxilo, ambos ligados ao carbono α (o primeiro depois do grupo carboxilo). O carbono α também é ligado a um hidrogénio e a uma cadeia lateral, que é representada pela letra R. O grupo R determina a identidade de um aminoácido específico. A fórmula bidimensional mostrada aqui pode transmitir somente parte da estrutura comum dos aminoácidos, porque uma das propriedades mais importantes de tais compostos é a forma tridimensional, ou estereoquímica. Os aminoácidos são classificados em polares, não-polares e neutros, dependendo da natureza da cadeia lateral.[22]
Existem 20 aminoácidos principais, sendo denominados aminoácidos primários ou padrão, mas além desses, existem alguns aminoácidos especiais, que só aparecem em alguns tipos de proteínas. Desses 20, nove são ditos essenciais: isoleucina, leucina, lisina, metionina, fenilalanina, treonina triptofano, valina, histidina e arginina.[4] O organismo humano não é capaz de produzi-los, e por isso é necessária a sua ingestão através dos alimentos para evitar a sua deficiência no organismo. Uma cadeia de aminoácidos denomina-se de "peptídeo", estas podem possuir dois aminoácidos (dipeptídeos), três aminoácidos (tripeptídeos), quatro aminoácidos (tetrapeptídeos), ou muitos aminoácidos (polipeptídeos). O termo proteína é dado quando na composição do polipeptídeo entram centenas ou milhares de aminoácidos.
As ligações entre aminoácidos denominam-se ligações peptídicas e estabelecem-se entre o grupo amina e o grupo carboxilo de dois aminoácidos diferentes, com a perda de uma molécula de água.[23]
Funções
As proteínas exercem funções importantes no organismo de todos os seres vivos, das quais se destacam as seguintes.
Estrutural ou plástica
São aquelas que participam dos tecidos dando-lhes rigidez, consistência e elasticidade. São proteínas estruturais: colágeno (constituínte das cartilagens), actina[24] e miosina[24] (presentes na formação das fibras musculares), queratina (principal proteína do cabelo), fibrinogênio (presente no sangue), albumina (encontrada em ovos) e outras.
Hormonal
Exercem alguma função específica sobre algum órgão ou estrutura de um organismo como, por exemplo, a insulina que retira a glicose em excesso do sangue(embora tecnicamente a insulina seja considerada apenas um polipeptídeo, devido a seu pequeno tamanho).
Defesa
Os anticorpos são proteínas que realizam a defesa do organismo, especializados no reconhecimento e neutralização de vírus, bactérias e outras substâncias estranhas.
O fibrinogênio e a trombina são outras proteínas de defesa, responsáveis pela coagulação do sangue e prevenção de perda sanguínea em casos de cortes e ferimentos.
Energética

Obtenção de energia a partir dos canais que compõem as proteínas. Durante a fase de crescimento as crianças são especialmente sensíveis às deficiências de nutrientes, sobretudo proteínas. Deficiência de calorias ou proteínas na dieta desvia as proteínas para a função energética, levando à deficiência de crescimento. A necessidade diária de proteínas é de cerca de 1g/kg durante essa fase e 0,8-0,9g/kg na fase adulta.
Enzimática
Enzimas são proteínas capazes de catalisar reações bioquímicas[25] como, por exemplo, as lipases. As enzimas não reagem, são reutilizadas (sempre respeitando o sítio ativo) e são específicas.
As enzimas reduzem a energia de ativação das reações químicas. A função da enzima depende diretamente de sua estrutura. Proteínas altamente especializadas e com atividade catalítica. Mais de 2000 enzimas são conhecidas, acreditava-se que cada uma era capaz de catalisar apenas um tipo diferente de reação química, porém novas pesquisas provaram que algumas enzimas podem catalisar diferentes reações químicas.
Condutoras de gases
O transporte de gases (principalmente do oxigênio e um pouco do gás carbônico) é realizado por proteínas como a hemoglobina e hemocianina presentes nos glóbulos vermelhos ou hemácias .
Outras proteínas
Há diferentes proteínas cujas funções podem ser consideradas exóticas e de difícil classificação. Uma planta africana possui uma proteína denominada monelina que tem um sabor extremamente adocicado. O seu uso é possível em adoçantes não-tóxicos e quase sem calorias para o uso humano. Alguns peixes da Antártica contém proteínas anticongelantes no plasma sanguíneo, as quais protegem o sangue destes animais do congelamento.[26]
Desnaturação

A desnaturação ocorre quando a proteína perde sua estrutura secundária e/ou terciária, ou seja, o arranjo tridimensional da cadeia polipeptídica é rompido, fazendo com que, quase sempre, perca sua atividade biológica característica.
Quando as proteínas sofrem desnaturação não ocorre rompimento de ligações covalentes do esqueleto da cadeia polipeptídica, preservando a seqüência de aminoácidos característica da proteína.
Os fatores que causam a desnaturação, são:
- Aumento de temperatura[27] (cada proteína suporta certa temperatura máxima, se esse limite é ultrapassado ela desnatura);
- Extremos de pH;
- Solventes orgânicos miscíveis com a água (etanol e acetona);
- Solutos (ureia);[27]
- Exposição da proteína a detergentes;
- Agitação vigorosa da solução proteica até formação abundante de espuma.
Renaturação
Experimentos que demonstram a reversibilidade da desnaturação, ajudam a provar que a estrutura terciária de uma proteína globular é determinada pela sua sequência de aminoácidos. As proteínas desnaturadas retomam sua estrutura nativa e sua atividade biológica assim que sua conformação natural de estabilidade é atingida novamente. Renaturação é o nome que se dá a esse processo.[28]
Por exemplo, uma ribonuclease purificada pode ser desnaturada pela presença de um agente redutor em solução concentrada de ureia, através da clivagem de quatro de suas oito ligações dissulfureto a resíduos de cisteína e pela desestabilização das interações hidrofóbicas causada pela ureia. A desnaturação da ribonuclease é acompanhada por uma completa perda de sua atividade catalítica. Removidos o agente redutor e a ureia, a ribonuclease desnaturada retoma sua estrutura terciária correta, dobrando-se espontaneamente, com restauração completa de sua atividade catalítica. As ligações dissulfureto são refeitas nas mesmas posições anteriores.[28]
História e etimologia

As proteínas foram pela primeira vez descritas pelo químico holandês Gerardus Johannes Mulder e assim batizadas pelo químico sueco Jöns Jacob Berzelius em 1838. Mulder levou a cabo análises elementares de proteínas vulgares e constatou que praticamente todas as proteínas apresentavam a mesma fórmula empírica – C400H620N100O120P1S1. Ainda que erradamente, concluiu que as proteínas deveriam ser constituídas por um único tipo de molécula de grande dimensão.[29] O termo "proteína" para descrever estas moléculas foi proposto pelo sócio de Mulder, Berzelius. Proteína deriva da palavra grega πρωτεῖος (proteios), a qual significa "na liderança" ou "a que está à frente".[30] Mulder prossegui a investigação, identificando produtos da degradação proteica, como o aminoácido leucina, para o qual determinou o peso molecular quase preciso de 131 Da.[29]
Os cientistas pioneiros no campo da nutrição, como o alemão Carl von Voit, acreditavam que a proteína era o mais importante nutriente na manutenção da estrutura corporal, uma vez que existia a crença generalizada de que seria a carne tinha origem na própria carne.[31] Karl Heinrich Ritthausen alargou o campo das proteínas conhecidas com a identificação do ácido glutâmico. Thomas Burr Osborne compilou em 1909 uma revisão detalhada de todas as proteínas vegetais e, no mesmo ano e em conjunto com Lafayette Mendel, determinou os aminoácidos essenciais à sobrevivência de ratos de laboratório aplicando a lei de Liebig. A compreensão das proteínas enquanto polipetídeos foi proporcionada por Franz Hofmeister e Hermann Emil Fischer. O papel central das proteínas enquanto enzimas nos organismos vivos foi determinado em 1926, quando ames Batcheller Sumner demonstrou que a urease era de facto uma proteína.[32]
A dificuldade em purificar proteínas em grande quantidade dificultou imenso a investigação dos primeiros bioquímicos. Assim, a investigação inicial focou-se sobretudo em proteínas que podiam ser facilmente purificadas em quantidade, como as do sangue, da clara de ovo, diversas toxinas e enzimas digestivas obtidas em matadouros.[29] Atribiu-se a Linus Pauling a primeira previsão bem sucedida de de estruturas secundárias de proteínas com base nas ligações de hidrogénio, uma ideia que já tinha sido proposta em 1933 por William Astbury.[33] Posteriormente, a investigação de Walter Kauzmann sobre a desnaturação, baseada em parte nos estudos anteriores de Kaj Ulrik Linderstrøm-Lang, veio a contribuir para a compreensão do enovelamento de proteínas e das estruturas mediadas por interações hidrófugas.[34][35][36] A primeira proteína a ser sequenciada foi a insulina, por Frederick Sanger em 1949. Sanger determinou corretamente a sequência de aminoácidos da proteína, demonstrando de forma conclusiva que as proteínas eram constituídas por polímeros lineares de aminoácidos, em vez de cadeias ramificadas ou coloides.[37]
As primeiras estruturas proteicas a serem resolvidas foram as da hemoglobina e da mioglobina, por Max Perutz e John Kendrew, respetivamente, em 1958.[38][39] Nas décadas posteriores, a crio-microscopia eletrónica de grandes conjuntos macromoleculares e a previsão computacional de estruturas proteicas de pequenos domínios foram métodos que permitiram a investigação de proteínas à escala atómica.[40][41] No início de 2014, estavam registadas no Protein Data Bank aproximadamente 90 000 estruturas proteicas com resolução atómica.«RCSB Protein Data Bank». Consultado em 1 de julho de 2013
Ver também
Referências
- ↑ Watson, James D.; Baker, Tania A.; Bell, Stephen P.; Gann, Alexander; Levine, Michael; Losick, Richard (2004). Molecular Biology of the Gene (em inglês) 5ª ed. San Francisco, CA: Pearson, Benjamin Cummings. p. 72. 732 páginas. ISBN 0-8053-4635-X
- ↑ Portal São Francisco - Proteínas
- ↑ Marzzoco, Anita, "Bioquímica Básica" - Ed. Guanabara Koogan, 3ª edição. pg 11
- ↑ a b c Alberts, Bruce; Johnson, Alexander; Lewis, Julian; Raff, Martin; Roberts, Keith; Walter, Peter (2010). «2: Química Celular e Biossíntese e 3: Proteínas». Biologia Molecular da Célula 5 ed. Porto Alegre: Artmed. pp. 45–194. ISBN 978-85-363-2066-3
- ↑ Pevzner, Pavel A (2000). Computational Molecular Biology. An Algorithmic Approach (em inglês). Cambridge, Massachusetts: MIT Press. p. 271. 314 páginas. ISBN 0-262-16197-4
- ↑ Nelson 2005.
- ↑ Gutteridge 2005.
- ↑ Murray 2006, p. 19.
- ↑ Murray 2006, p. 31.
- ↑ a b Lodish 2004.
- ↑ vanHolde 1996.
- ↑ Pain 2000.
- ↑ Van Holde 1996, pp. 1002-1042.
- ↑ Fulton 1991.
- ↑ Bruckdorfer 2004.
- ↑ Schwarzer 2005.
- ↑ Kent 2009.
- ↑ Alphey, Luke (1997). «17: Protein Structure Prediction». DNA Sequencing. From Experimental Methods to Bioinformatics (em inglês). New York: Springer. p. 179. ISBN 0-387-91509-5
- ↑ a b c d Karp, Gerald (2008). Cell and Molecular Biology. Concepts and Experiments (em inglês) 5ª ed. New Jersey: John Wiley. p. 49-64. ISBN 978-0-470-04217-5
- ↑ a b c Sharma, Kal Renganathan (2009). Bioinformatics. New York: McGraw-Hill. p. 2-6. 320 páginas. ISBN 978-0-07-159306-9
- ↑ a b Setubal, João; Meidanis, João (1997). «8: Molecular Structure Predition». Introduction to Computational Molecular Biology. Boston: PWS Publishing Company. p. 252-253. 296 páginas. ISBN 0-534-95262-3
- ↑ Sperelakis, Nicholas (editor); Forbes, Michael S. (autor do capítulo); Ferguson, Donald G. (autor do capítulo). «2:Physiological Structure and Functions of Proteins». Cell Physiology Sourcebook. A Molecular Approach (em inglês) 3ª ed. San Diego, California: Academic Press. p. 19. 1235 páginas. ISBN 0-12-656977-0
- ↑ Clayden, Jonathan; Greeves, Nick; Warren, Stuart; Wothers, Peter (2000). Organic Chemistry (em inglês). Oxford: Oxford University Press. p. 165. 1536 páginas. ISBN 0-19850346-6
- ↑ a b Bolsover, Stephen R.; Hyams, Jeremy S.; Shephard, Elizabeth A.; White, Hugh A.; Wiedemann, Claudia G (2004). Cell Biology (em inglês). Hoboken, New Jersey: John Wiley & Sons. p. 390-397. 531 páginas. ISBN 0-471-26393-1
- ↑ Stansfield, William D.; Colomé, Jaime S.; Cano, Raúl J. Molecular and Cell Biology (em inglês). New York: McGraw-Hill. p. 1. 122 páginas. ISBN 0-07-139881-3
- ↑ LEHNINGER, Albert Lester; NELSON, David L; COX, Michael. Princípios da Bioquímica. 2.ed. São Paulo: Sarvier, 1995.
- ↑ a b Lesk, Arthur M (2008). Introduction to Bioinformatics (em inglês) 3ª ed. Oxford: Oxford University Press. p. 308-318. 474 páginas. ISBN 978-0-19-920804-3
- ↑ a b L. Nelson, David. Lehninger Principles of Biochemstry (em inglês) 4 ed. [S.l.: s.n.] p. 148
- ↑ a b c Perrett 2007.
- ↑ Reynolds 2003.
- ↑ Bischoff 1860.
- ↑ Sumner 1926.
- ↑ Pauling 1951.
- ↑ Kalman 1955.
- ↑ Kauzmann 1956.
- ↑ Kauzmann 1959.
- ↑ Sanger 1949.
- ↑ Muirhead 1963.
- ↑ Kendrew 1958.
- ↑ Zhou 2008.
- ↑ keskin 2008.
Bibliografia
- Bischoff TLW, Voit, C (1860). Die Gesetze der Ernaehrung des Pflanzenfressers durch neue Untersuchungen festgestellt (em German). Leipzig, Heidelberg: [s.n.]
- Brosnan J (June 2003). «Interorgan amino acid transport and its regulation». Journal of Nutrition. 133 (6 Suppl 1): 2068S–72S. PMID 12771367 Verifique data em:
|data=(ajuda)
- Bruckdorfer T, Marder O, Albericio F (2004). «From production of peptides in milligram amounts for research to multi-tons quantities for drugs of the future». Current Pharmaceutical Biotechnology. 5 (1): 29–43. PMID 14965208. doi:10.2174/1389201043489620
- Cedrone F, Ménez A, Quéméneur E (2000). «Tailoring new enzyme functions by rational redesign». Current Opinion in Structural Biology. 10 (4): 405–10. PMID 10981626. doi:10.1016/S0959-440X(00)00106-8
- Conrotto P, Souchelnytskyi S (2008). «Proteomic approaches in biological and medical sciences: principles and applications». Experimental Oncology. 30 (3): 171–80. PMID 18806738
- Copland JA, Sheffield-Moore M, Koldzic-Zivanovic N, Gentry S, Lamprou G, Tzortzatou-Stathopoulou F, Zoumpourlis V, Urban RJ, Vlahopoulos SA (2009). «Sex steroid receptors in skeletal differentiation and epithelial neoplasia: is tissue-specific intervention possible?». BioEssays: news and reviews in molecular, cellular and developmental biology. 31 (6): 629–41. PMID 19382224. doi:10.1002/bies.200800138
- Bairoch, A (2000). «The ENZYME database in 2000» (PDF). Nucleic Acids Research. 28 (1): 304–305. PMC 102465
 . PMID 10592255. doi:10.1093/nar/28.1.304
. PMID 10592255. doi:10.1093/nar/28.1.304
- Fernández A, Scott R (2003). «Dehydron: a structurally encoded signal for protein interaction». Biophysical Journal. 85 (3): 1914–28. Bibcode:2003BpJ....85.1914F. PMC 1303363
 . PMID 12944304. doi:10.1016/S0006-3495(03)74619-0
. PMID 12944304. doi:10.1016/S0006-3495(03)74619-0
- Fulton A, Isaacs W (1991). «Titin, a huge, elastic sarcomeric protein with a probable role in morphogenesis». BioEssays. 13 (4): 157–61. PMID 1859393. doi:10.1002/bies.950130403
- Gonen T, Cheng Y, Sliz P, Hiroaki Y, Fujiyoshi Y, Harrison SC, Walz T (2005). «Lipid-protein interactions in double-layered two-dimensional AQP0 crystals». Nature. 438 (7068): 633–38. Bibcode:2005Natur.438..633G. PMC 1350984
 . PMID 16319884. doi:10.1038/nature04321
. PMID 16319884. doi:10.1038/nature04321
- Görg A, Weiss W, Dunn MJ (2004). «Current two-dimensional electrophoresis technology for proteomics». Proteomics. 4 (12): 3665–85. PMID 15543535. doi:10.1002/pmic.200401031
- Gutteridge A, Thornton JM (2005). «Understanding nature's catalytic toolkit». Trends in Biochemical Sciences. 30 (11): 622–29. PMID 16214343. doi:10.1016/j.tibs.2005.09.006
- Herges T, Wenzel W (2005). «In silico folding of a three helix protein and characterization of its free-energy landscape in an all-atom force field». Physical Review Letters. 94 (1): 018101. Bibcode:2005PhRvL..94a8101H. PMID 15698135. doi:10.1103/PhysRevLett.94.018101
- Hey J, Posch A, Cohen A, Liu N, Harbers A (2008). «Fractionation of complex protein mixtures by liquid-phase isoelectric focusing». Methods in Molecular Biology. Methods in Molecular Biology. 424: 225–39. ISBN 978-1-58829-722-8. PMID 18369866. doi:10.1007/978-1-60327-064-9_19
- Hoffmann M, Wanko M, Strodel P, König PH, Frauenheim T, Schulten K, Thiel W, Tajkhorshid E, Elstner M (2006). «Color tuning in rhodopsins: the mechanism for the spectral shift between bacteriorhodopsin and sensory rhodopsin II». Journal of the American Chemical Society. 128 (33): 10808–18. PMID 16910676. doi:10.1021/ja062082i
- Hohsaka T, Sisido M (2002). «Incorporation of non-natural amino acids into proteins». Current Opinion in Chemical Biology. 6 (6): 809–15. PMID 12470735. doi:10.1016/S1367-5931(02)00376-9
- Joos T, Bachmann J (2009). «Protein microarrays: potentials and limitations». Frontiers in Bioscience. 14 (14): 4376–85. PMID 19273356. doi:10.2741/3534
- Kalman SM, Linderstrom-Lang K, Ottesen M, Richards FM (1955). «Degradation of ribonuclease by subtilisin». Biochimica et Biophysica Acta. 16 (2): 297–99. PMID 14363272. doi:10.1016/0006-3002(55)90224-9
- Kauzmann W (1956). «Structural factors in protein denaturation». Journal of Cellular Physiology. Supplement. 47 (Suppl 1): 113–31. PMID 13332017. doi:10.1002/jcp.1030470410
- Kauzmann W (1959). «Some factors in the interpretation of protein denaturation». Advances in Protein Chemistry. Advances in Protein Chemistry. 14: 1–63. ISBN 978-0-12-034214-3. PMID 14404936. doi:10.1016/S0065-3233(08)60608-7
- Kendrew J, Bodo G, Dintzis H, Parrish R, Wyckoff H, Phillips D (1958). «A three-dimensional model of the myoglobin molecule obtained by X-ray analysis». Nature. 181 (4610): 662–66. Bibcode:1958Natur.181..662K. PMID 13517261. doi:10.1038/181662a0
- Kent SB (2009). «Total chemical synthesis of proteins». Chemical Society Reviews. 38 (2): 338–51. PMID 19169452. doi:10.1039/b700141j
- Keskin O, Tuncbag N, Gursoy A (2008). «Characterization and prediction of protein interfaces to infer protein-protein interaction networks». Current Pharmaceutical Biotechnology. 9 (2): 67–76. PMID 18393863. doi:10.2174/138920108783955191
- Koegl M, Uetz P (2007). «Improving yeast two-hybrid screening systems». Briefings in Functional Genomics & Proteomics. 6 (4): 302–12. PMID 18218650. doi:10.1093/bfgp/elm035
- Kuhlman B, Dantas G, Ireton GC, Varani G, Stoddard BL, Baker D (2003). «Design of a novel globular protein fold with atomic-level accuracy». Science. 302 (5649): 1364–68. Bibcode:2003Sci...302.1364K. PMID 14631033. doi:10.1126/science.1089427
- Lodish H, Berk A, Matsudaira P, Kaiser CA, Krieger M, Scott MP, Zipurksy SL, Darnell J (2004). Molecular Cell Biology 5th ed. New York, New York: WH Freeman and Company
- Margolin W (2000). «Green fluorescent protein as a reporter for macromolecular localization in bacterial cells». Methods (San Diego, Calif.). 20 (1): 62–72. PMID 10610805. doi:10.1006/meth.1999.0906
- Mayhew TM, Lucocq JM (2008). «Developments in cell biology for quantitative immunoelectron microscopy based on thin sections: a review». Histochemistry and Cell Biology. 130 (2): 299–313. PMC 2491712
 . PMID 18553098. doi:10.1007/s00418-008-0451-6
. PMID 18553098. doi:10.1007/s00418-008-0451-6
- Muirhead H, Perutz M (1963). «Structure of hemoglobin. A three-dimensional fourier synthesis of reduced human hemoglobin at 5.5 Å resolution». Nature. 199 (4894): 633–38. Bibcode:1963Natur.199..633M. PMID 14074546. doi:10.1038/199633a0
- Nelson DL, Cox MM (2005). Lehninger's Principles of Biochemistry 4th ed. New York, New York: W. H. Freeman and Company
- Dobson CM (2000). «The nature and significance of protein folding». In: Pain RH (ed.). Mechanisms of Protein Folding. Oxford, Oxfordshire: Oxford University Press. pp. 1–28. ISBN 0-19-963789-X
- Pauling L, Corey RB, Branson HR (1951). «The structure of proteins: two hydrogen-bonded helical configurations of the polypeptide chain» (PDF). Proceedings of the National Academy of Sciences U.S.A. 37 (5): 235–40. Bibcode:1951PNAS...37..235P. PMC 1063348
 . PMID 14834145. doi:10.1073/pnas.37.5.235
. PMID 14834145. doi:10.1073/pnas.37.5.235
- Perrett D (2007). «From 'protein' to the beginnings of clinical proteomics». Proteomics – Clinical Applications. 1 (8): 720–38. PMID 21136729. doi:10.1002/prca.200700525
- Plewczyński D, Ginalski K (2009). «The interactome: predicting the protein–protein interactions in cells». Cellular & Molecular Biology Letters. 14 (1): 1–22. PMID 18839074. doi:10.2478/s11658-008-0024-7
- Radzicka A, Wolfenden R (1995). «A proficient enzyme». Science. 267 (5194): 90–93. Bibcode:1995Sci...267...90R. PMID 7809611. doi:10.1126/science.7809611
- Reynolds JA, Tanford C (2003). Nature's Robots: A History of Proteins (Oxford Paperbacks). New York, New York: Oxford University Press. p. 15. ISBN 0-19-860694-X
- Ritchie DW (2008). «Recent progress and future directions in protein–protein docking». Current Protein and Peptide Science. 9 (1): 1–15. PMID 18336319. doi:10.2174/138920308783565741
- Rüdiger H, Siebert HC, Solís D, Jiménez-Barbero J, Romero A, von der Lieth CW, Diaz-Mariño T, Gabius HJ (2000). «Medicinal chemistry based on the sugar code: fundamentals of lectinology and experimental strategies with lectins as targets». Current Medicinal Chemistry. 7 (4): 389–416. PMID 10702616. doi:10.2174/0929867003375164
- Samarin S, Nusrat A (2009). «Regulation of epithelial apical junctional complex by Rho family GTPases». Frontiers in bioscience: a journal and virtual library. 14 (14): 1129–42. PMID 19273120. doi:10.2741/3298
- Sanger F (1949). «The terminal peptides of insulin». Biochemical Journal. 45 (5): 563–74. PMC 1275055
 . PMID 15396627
. PMID 15396627
- Sankaranarayanan R, Moras D (2001). «The fidelity of the translation of the genetic code». Acta Biochimica Polonica. 48 (2): 323–35. PMID 11732604
- Scheraga HA, Khalili M, Liwo A (2007). «Protein-folding dynamics: overview of molecular simulation techniques». Annual Review of Physical Chemistry. 58: 57–83. Bibcode:2007ARPC...58...57S. PMID 17034338. doi:10.1146/annurev.physchem.58.032806.104614
- Schwarzer D, Cole P (2005). «Protein semisynthesis and expressed protein ligation: chasing a protein's tail». Current Opinion in Chemical Biology. 9 (6): 561–69. PMID 16226484. doi:10.1016/j.cbpa.2005.09.018
- Sleator RD. (2012). «Prediction of protein functions». Methods in Molecular Biology. Methods in Molecular Biology. 815: 15–24. ISBN 978-1-61779-423-0. PMID 22130980. doi:10.1007/978-1-61779-424-7_2
- Standley DM, Kinjo AR, Kinoshita K, Nakamura H (2008). «Protein structure databases with new web services for structural biology and biomedical research». Briefings in Bioinformatics. 9 (4): 276–85. PMID 18430752. doi:10.1093/bib/bbn015
- Stepanenko OV, Verkhusha VV, Kuznetsova IM, Uversky VN, Turoverov KK (2008). «Fluorescent proteins as biomarkers and biosensors: throwing color lights on molecular and cellular processes». Current Protein & Peptide Science. 9 (4): 338–69. PMC 2904242
 . PMID 18691124. doi:10.2174/138920308785132668
. PMID 18691124. doi:10.2174/138920308785132668
- Sumner JB (1926). «The isolation and crystallization of the enzyme urease. Preliminary paper» (PDF). Journal of Biological Chemistry. 69 (2): 435–41
- Terpe K (2003). «Overview of tag protein fusions: from molecular and biochemical fundamentals to commercial systems». Applied Microbiology and Biotechnology. 60 (5): 523–33. PMID 12536251. doi:10.1007/s00253-002-1158-6
- EBI (20 de janeiro de 2010). «The Catalytic Site Atlas at The European Bioinformatics Institute». Ebi.ac.uk. Consultado em 16 de janeiro de 2011
- Walian P, Cross TA, Jap BK (2004). «Structural genomics of membrane proteins». Genome Biology. 5 (4): 215. PMC 395774
 . PMID 15059248. doi:10.1186/gb-2004-5-4-215
. PMID 15059248. doi:10.1186/gb-2004-5-4-215
- Walker JH, Wilson K (2000). Principles and Techniques of Practical Biochemistry. Cambridge, UK: Cambridge University Press. pp. 287–89. ISBN 0-521-65873-X
- Xiang Z (2006). «Advances in homology protein structure modeling». Current Protein and Peptide Science. 7 (3): 217–27. PMC 1839925
 . PMID 16787261. doi:10.2174/138920306777452312
. PMID 16787261. doi:10.2174/138920306777452312
- Yuste R (2005). «Fluorescence microscopy today». Nature Methods. 2 (12): 902–904. PMID 16299474. doi:10.1038/nmeth1205-902
- Zagrovic B, Snow CD, Shirts MR, Pande VS (2002). «Simulation of folding of a small alpha-helical protein in atomistic detail using worldwide-distributed computing». Journal of Molecular Biology. 323 (5): 927–37. PMID 12417204. doi:10.1016/S0022-2836(02)00997-X
- Zhang C, Kim SH (2003). «Overview of structural genomics: from structure to function». Current Opinion in Chemical Biology. 7 (1): 28–32. PMID 12547423. doi:10.1016/S1367-5931(02)00015-7
- Zhang Y, Skolnick J (2005). «The protein structure prediction problem could be solved using the current PDB library». Proceedings of the National Academy of Sciences U.S.A. 102 (4): 1029–34. Bibcode:2005PNAS..102.1029Z. PMC 545829
 . PMID 15653774. doi:10.1073/pnas.0407152101
. PMID 15653774. doi:10.1073/pnas.0407152101
- Zhang Y (2008). «Progress and challenges in protein structure prediction». Current Opinion in Structural Biology. 18 (3): 342–48. PMC 2680823
 . PMID 18436442. doi:10.1016/j.sbi.2008.02.004
. PMID 18436442. doi:10.1016/j.sbi.2008.02.004
- Zhou ZH (2008). «Towards atomic resolution structural determination by single-particle cryo-electron microscopy». Current Opinion in Structural Biology. 18 (2): 218–28. PMC 2714865
 . PMID 18403197. doi:10.1016/j.sbi.2008.03.004
. PMID 18403197. doi:10.1016/j.sbi.2008.03.004
- Branden, C; Tooze, J (1999). Introduction to Protein Structure. Nova Iorque: Garland Pub. ISBN 0-8153-2305-0
- Murray, RF; Harper HW; Granner DK; Mayes PA; Rodwell VW (2006). Harper's Illustrated Biochemistry. Nova Iorque: Lange Medical Books/McGraw-Hill. ISBN 0-07-146197-3
- Van Holde, KE; Mathews, CK (1996). Biochemistry. Menlo Park, California: Benjamin/Cummings Pub. Co., Inc. ISBN 0-8053-3931-0
- Voet, D; Voet JG (2004). Biochemistry. 1 3ª ed. Hoboken: Wiley. ISBN 978-0470917459 </ref>
- Lubert Stryer, Jeremy Mark Berg, John L. Tymoczko (trad. Serge Weinman), Biochimie, Flammarion, « Médecine-Sciences », Paris, 2003, 5ª éd. ISBN 2-257-17116-0.
- Carl-Ivar Brändén, John Tooze (trad. Bernard Lubochinsky, préf. Joël Janin), Introduction à la structure des protéines, De Boeck Université, Bruxelles, 1996 ISBN 2-804-12109-7.
- Rodríguez, Faride. La estructura de las proteínas
- Lehninger, Albert L. Princípios da bioquímica
Ligações externas
- Aminosäuren und Proteine
- Bau von Proteinen
- Peptide, Polypeptide (Proteine)
- Protein Database (Aminosäurensequenzen, 3D-Strukturen,…)
- Proteinbilder
- Human Protein Atlas
Predefinição:Bom interwiki Predefinição:Bom interwiki Predefinição:Bom interwiki Predefinição:Link FA Predefinição:Link FA
